HMM-профиль семейства белков
Из базы данных Pfam я скопировал информацию по всем записям раздела Browse в файл, и при помощи скрипта отфильтровал записи о доменах, соответствующих требованиям, в отдельный файл.
Я выбрал домен Sugarporin N (AC PF11471, 10 белков в seed, 105 - в full, длина профиля HMM 31 аминокислота) и двухдоменную архитектуру с Porin 8 (AC архитектуры T0P7H4_PHOTE, АС порина 8 PF16966, порядок доменов: Sugarporin N - Porin 8, белков с данной архитектурой 26).
Последовательности всех белков с доменом Sugarporin N лежат в fasta-файле; из данного файла я получил список АС. Список АС белков, содержащих двухдоменную структуру с Porin 8, я взял из раздела Architectures.
Для создания выравнивания я объединил последовательности белков, содержащих выбранную архитектуру, в файл при помощи скрипта. Выравнивание я выполнил в программе Mega X алгоритмом muscle, получив на выходе следующий файл.
После удаления части последовательностей и частей белков до начала первого домена выравнивание приобрело следующий вид.
При помощи команды
hmm2build profile arch_aligned_rev.fasя построил HMM-профиль доменной архитектуры и откалибровал его командой
hmm2calibrate profileНаконец, при помощи команды
hmmsearch --cpu=1 profile full.fastaя произвёл поиск доменной архитектуры по базе данных белков из ранее полученного файла, получив следующий лог-файл. На основе него я сделал таблицу по белкам Длина профиля HMM двухдоменной архитектуры 439 аминокислот
При помощи скрипта я построил ROC-кривую:
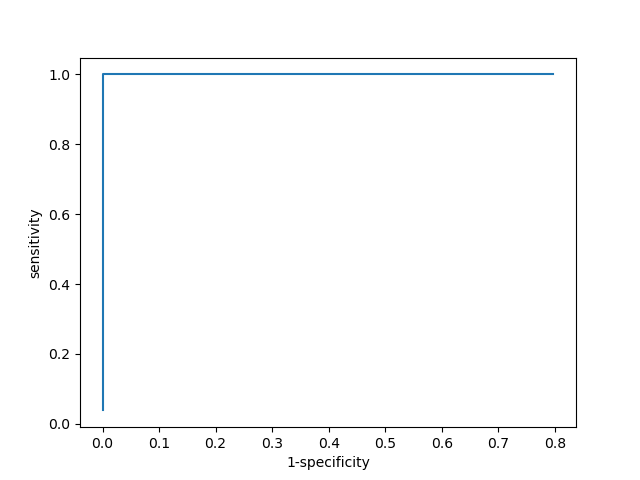
и вычислил значение порогового веса с наибольшим значением F1; оно получилось 758. Сильно прямоугольную форму кривой можно объяснить тем, что в результатах HMMsearch наблюдается очень резкий скачок весов как раз в районе 400-700