Работа с сигнальными последовательностями в геноме
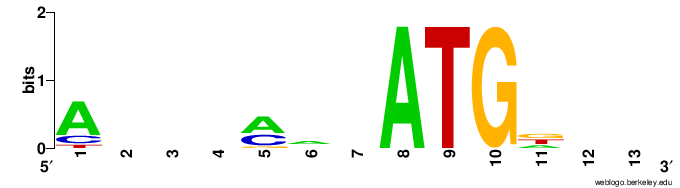
Данные сигналы (их два вида: гептамерные и нонамерные) расположены между участками ДНК, кодирующими V-, D- и J-сегменты будущего антитела или B-/T-клеточного мембранного рецептора. Они узнаются белками RAG1/RAG2, и через них производится V(D)J-рекомбинация.
Эффективность сигнала высокая, ведь V(D)J-рекомбинация происходит во всех лимфоцитах
Консенсусная последовательность для гептамера CACAGTG, для нонамера - ACAAAAACC.
Источник - статья в Annual Review of Immunology:
Schatz, David G., Oettinger, Marjorie A., Schlissel, Mark S. (1992)
"V(D)J Recombination: Molecular Biology and Regulation"
doi:10.1146/annurev.immunol.10.1.359
Для выполнения этого задания я пользовался в основном средствами Python. Из данных я использовал предоставленные таблицу координат старт-кодонов SARS-CoV-2 и последовательность его генома, а также загрузил последовательность хромосомы 12 человека с NCBI и таблицу координат старт-кодонов на ней с предоставленного в задании сервиса
Мной были получены "выравнивания" без гэпов для окрестностей старт-кодонов SARS-CoV-2, для окрестностей остальных ATG SARS-CoV-2 и для хромосомы 12 человека
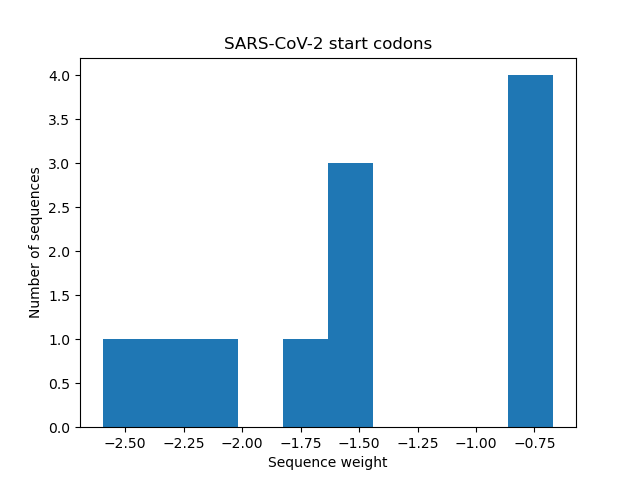
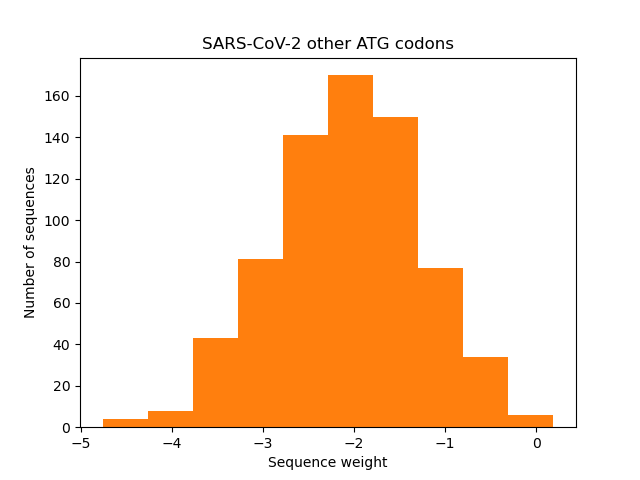
По окрестностям старт-кодонов хромосомы 12 человека я составил PWM для последовательности Козак человека, а затем построил гистограммы, показывающие распределение весов окрестностей старт-кодонов SARS-CoV-2 (верхняя) и окрестностей остальных ATG SARS-CoV-2 (нижняя)
Единственный вывод, который однозначно можно сделать, смотря на эти гистограммы, - 11 старт-кодонов SARS-CoV-2 явно недостаточно для оценки полученной PWM. В целом видно, что веса последовательностей возле старт-кодонов выше, но статистическая значимость результатов под сомнением
Получилась матрица IC(b,j) следующего содержания