Учебный сайт Вероники Мурашка
Практикум 15
Сборка генома de novo
Код доступа, который был мне предложен, - SRR4240361
1. Скачивание архива с помощью команды:
wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/001/SRR4240361/SRR4240361.fastq.gz
2. Добавление файла со списком всех адаптеров
cat /mnt/scratch/NGS/adapters/* > adapters.fasta
3. Удаление адаптеров; вес файла изменился с 193М до 192М, было удалено 0,47% чтений.
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240361.fastq.gz SRR4240361_trim_adapt.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 2> log_trim_adapt.txt
4. Удаление нуклеотидов плохого качества (меньше 20) с 3’ конца, при этом оставлялись только те чтения, длина которых больше 32 нуклеотидов. До удаления вес файлы был 192М, после - 178М; отброшено 5.58% чтений.
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240361_adapttrim.fastq.gz SRR4240361_trim.fastq.gz TRAILING:20 MINLEN:32 2>log_trim.txt
5. Создание директории kmers с k-мерами длины 31
velveth kmers 31 -short -fastq.gz SRR4240361_trim_len.fastq.gz
6. Сборка генома
velvetg kmers
7. Анализ сборки
N50 - 25683
3 самых длинных фрагмента:
| id фрагмента | длина | покрытие |
|---|---|---|
| 6 | 49238 | 26.660851 |
| 2 | 45555 | 26.450466 |
| 34 | 43866 | 23.514977 |
---------
id контига - 6
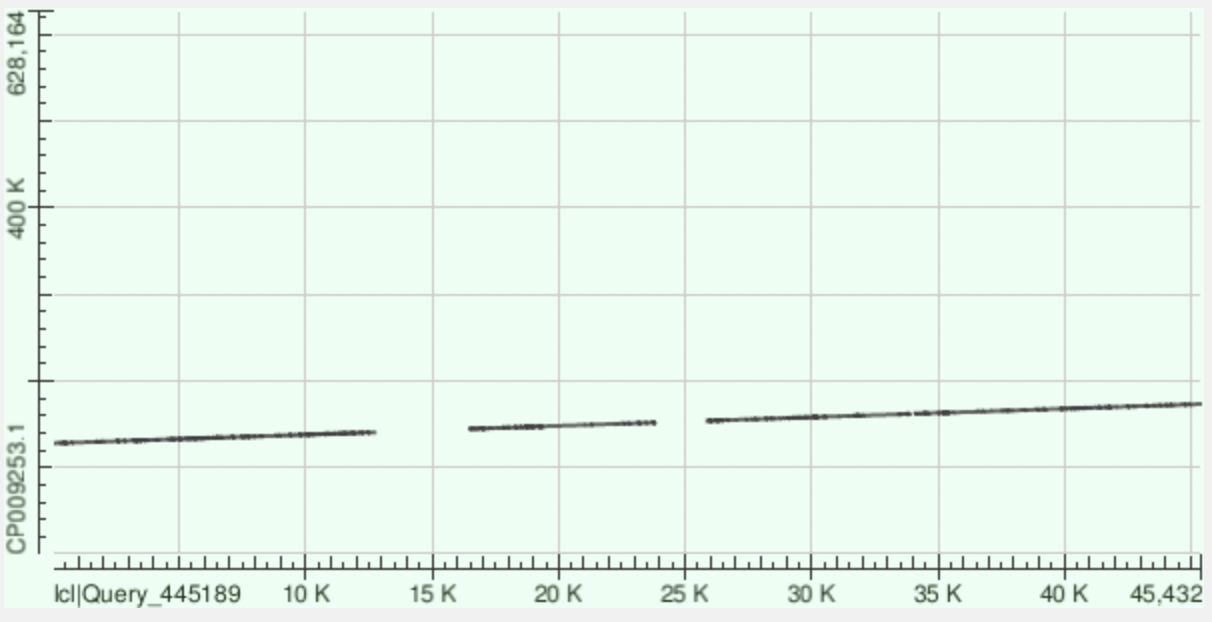
| координаты контига | координаты хромосомы | Gaps | Identities |
|---|---|---|---|
| 50-12790 | 127825-140555 | 548/13010(4%) | 9751/13010(75%) |
| 25809-33893 | 153752-161738 | 264/8168(3%) | 6355/8168(78%) |
| 16429-23828 | 144368-151796 | 243/7536(3%) | 5859/7536(78%) |
| 34098-38958 | 161898-166752 | 112/4914(2%) | 3911/4914(80%) |
| 38989-45432 | 166750-173180 | 159/6517(2%) | 4967/6517(76%) |
---------
id контига - 2
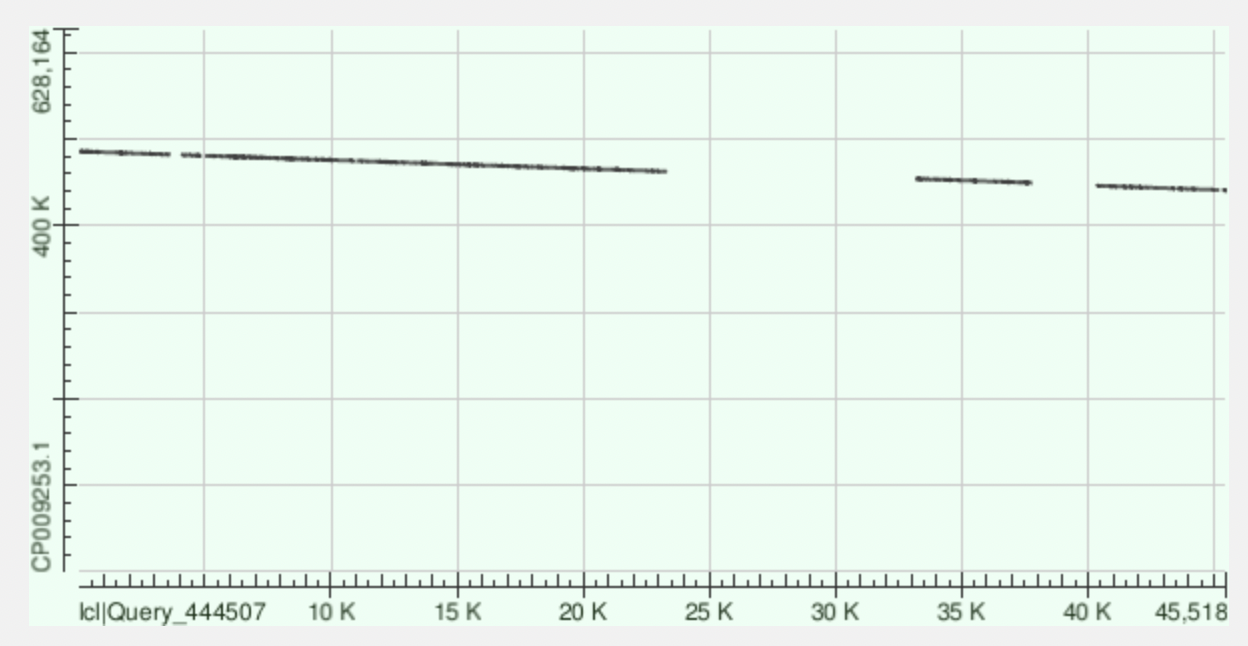
| координаты контига | координаты хромосомы | Gaps | Identities |
|---|---|---|---|
| 10984-18297 | 467412-474667 | 208/7389(2%) | 5691/7389(77%) |
| 18327-23268 | 462496-467421 | 162/5015(3%) | 3862/5015(77%) |
| 40383-43410 | 442877-445895 | 61/3054(1%) | 2451/3054(80%) |
| 5007-10881 | 474844-480660 | 250/5971(4%) | 4426/5971(74%) |
| 33159-37811 | 449411-454069 | 152/4732(3%) | 3571/4732(75%) |
| 12-3647 | 481997-485679 | 129/3724(3%) | 2850/3724(77%) |
| 43540-45215 | 441135-442817 | 29/1694(1%) | 1339/1694(79%) |
| 4122-4801 | 480874-481545 | 20/686(2%) | 564/686(82%) |
| 45337-45518 | 40755-440944 | 8/190(4%) | 169/190(89%) |
---------
id контига - 34
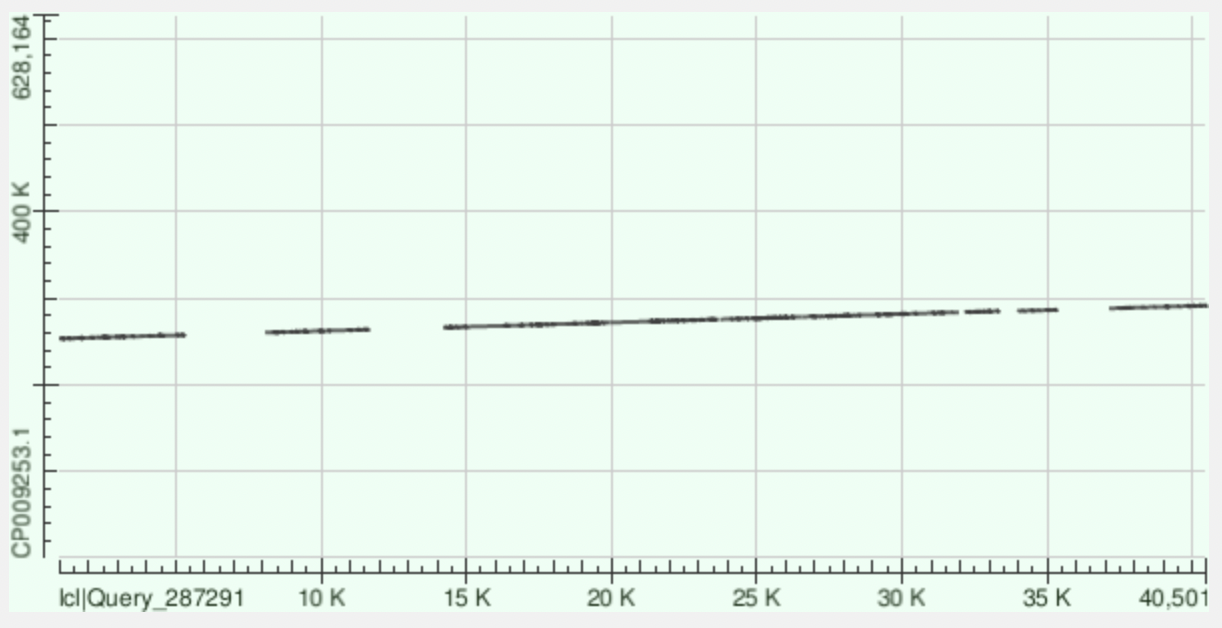
| координаты контига | координаты хромосомы | Gaps | Identities |
|---|---|---|---|
| 14198-23677 | 266073-275551 | 361/9660(3%) | 7611/9660(79%) |
| 23736-31957 | 275566-283706 | 429/8396(5%) | 6371/8396(76%) |
| 8077-11648 | 260224-263784 | 101/3617(2%) | 2788/3617(77%) |
| 37135-40501 | 288181-291560 | 99/3423(2%) | 2654/3423(78%) |
| 977-5299 | 253223-257546 | 195/4421(4%) | 3245/4421(73%) |
| 34011-35345 | 285200-286535 | 27/1349(2%) | 1025/1349(76%) |
| 32205-33314 | 283963-285070 | 46/1132(4%) | 863/1132(76%) |