Матрицы аминокислотных замен
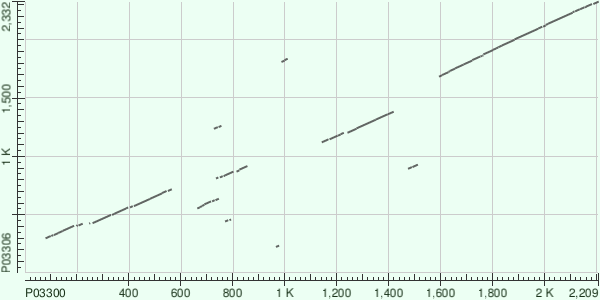
Карта локального сходства двух полипротеинов
Карта локального сходства была построена на осноснве данных о следующих последовательностей: AC P03300 (poliovirus) и AC P03306 (aphthovirus).
Карта локального сходства
Характеристика двух лучших выравниваний
| Характеристика |
I |
II |
| Identities (%) |
29 |
37 |
| Positives (%) |
48 |
51 |
| Length(poliovirus/aphthovirus) |
614/643 |
276/262 |
| Gaps |
59 |
26 |
| Score |
250 bits (638) |
157 bits (397) |
Названия зрелых белков
| Выравнивание |
I |
II |
| Poliovirus |
Protease 3C, RNA-directed RNA polymerase |
Protein 2C |
| Aphthovirus |
RNA-directed RNA polymerase 3D-POL |
Protein 2C |
Сравнение веса выравнивания со случайным
|
Гомологичные |
Неродственные |
| ID 1 |
SYS_ECOLI |
FUR_ECOLI |
| ID 2 |
SYS_BACSU |
SYS_BACSU |
| Вес оптимального локального выравнивания |
1095,5 |
41,0 |
| Медиана весов "случайных" выравниваний |
56,75 |
38,5 |
| Верхний квартиль весов "случайных" выравниваний |
63,5 |
42,25 |
| Пересчёт в биты |
154,(8) |
2,(3) |
| Вероятность |
2,36e-47 |
1,98e-1 |
|
|
|
Поиск гомологов в Blast
С помощью сервиса Blast были найдены два белка, наиболее гомологичных мнимому периплазменному железосвязывающему белку (ANS42615.1) из организма Serratia plymuthica PRI-2C. Ниже представлены их характеристики.
| ID(AC) находки |
Q56952.2 |
Q57449.1 |
| Организм |
Yersinia pestis |
Haemophilus influenzae Rd KW20 |
| Identities |
80% |
74% |
| Positives |
86% |
83% |
| Length(query/subject) |
311 |
293 |
| Gaps |
7 |
0 |
| Score |
491 bits(1265) |
442 bits(1138) |
| Expect |
6e-176 |
7e-157 |
|