BLAST
Определение таксономии и функции
Для определения принадлежности консенсусной последовательности ДНК, полученной анализом хроматограмм сэнгеровского секвенирования в практикуме "Секвенирование по Сэнгеру", был использован поиск алгоритмом blastn с параметрами по умолчанию.
Выбранный алгоритм лучше всего подходит для поиска гомологов исследуемой нуклеотидной последовательности, поскольку для неё неизвестно, является ли она белок-кодирующей, а также не гарантируется присутствие близких гомологов в базе данных.
Из выдачи BLAST можно сделать вывод, что данная последовательность скорее всего пренадлежит виду Loxosomella murmanica и кодирует 18S рибосомальную РНК. У находки с этим организмом высокий bit-score, высокий процент идентичности (97.88%) и наименьшее число гэпов (2).
Таксономия: Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Protostomia; Spiralia; Lophotrochozoa; Entoprocta; Loxosomatidae; Loxosomella
Поиск генов белков
Для аннотации был выбран геном тихоходки Hypsibius dujardini, АС сборки: GCA_001579985.1. Был выбран контиг с AC: LRSR01000001.1.
Для поиска кодирующих последовательностей в выбранном контиге идентификационный номер LRSR01000001.1 был использован в запросе в blastx; все параметры были выставлены по умолчанию, за исключением базы белков (Swiss-prot), и организма (кроме тихоходки). Согласно результату поиска наиболее вероятно в данном контиге обнаруживается последовательность молекулы нейронного распознавания NB-2 у которой Е-value = 5e-05.
Найденный участок гена серой крысы кодирует фрагмент молекулы нейронного распознавания NB-2 длиной 79 аминокислоты, в то время как сам белок состоит из 1099 аминокислот, т.е. процент покрытия находки ~7,2%. Эти показатели далеки от идеала главным образом потому, что тихоходка и крыса очень далёкие друг от друга организмы. Впрочем низкий процент идентичности (32.91%) сильно снижает вероятность гомологии
Интепретация карты локального сходства
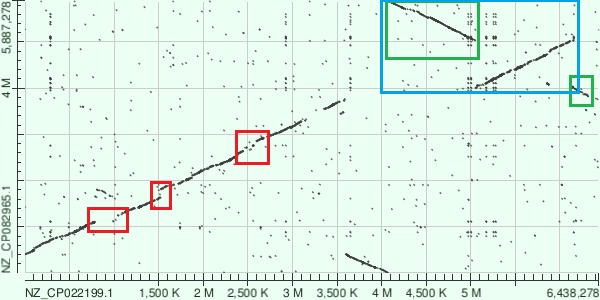
Для сравнения взяли геномы Achromobacter insolitus (NZ_CP022199.1) и Achromobacter mucicolens (NZ_CP082965.1). Карта локальных свойств была построена парным выравниванием геномов изучаемых бактерий с помощью алгоритма blastn. Параметры выставлены по умолчанию.
Красным прямоугольниками отмечены делеции,причём две из них(крайнии прямоугольники) произошли в обоих геномах с последующей вставкой разных последовательностей, синим прямоугольником отмечена транслокация, зелёными прямоугольниками отмечена инверсия.
|