Сборка de novo
|На главную| |
|Обо мне| |
|Семестры| |
|Заметки| |
|Ссылки| |
Методы
Для сборки были использованы прочтения генома бактерии Buchnera aphidicola (AC: SRR4240379) из базы ENA.
| Команда | Действие |
| cat /mnt/scratch/NGS/adapters/* > adapters.fasta | Создание файла с адаптерами |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240379.fastq.gz SRR4240379_wtht_a.fastq.gz ILLUMINACLIP:adapters.fasta:2:7:7 2> rmadapters.log | Очистка чтений от адаптеров |
| jjava -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240379_wtht_a.fastq.gz SRR4240379_cl.fastq.gz TRAILING:20 MINLEN:32 2> clean.log | Триммирование чтений |
| fastqc SRR4240379.fastq.gz | Оценка качества чтений до триммирования |
| fastqc SRR4240379_cl.fastq.gz | Оценка качества чтений после триммирования |
| velveth velveth 31 -short -fastq SRR4240379_cl.fastq.gz | Создание 31-меров из чтений |
| velvetg velveth | Создание сборки деново |
Подготовка чтений программой trimmomatic
Удаление адаптеров. По завершению работы trimmomatic для обрезания адаптеров была получена следующая информация:
Input Reads: 7400155 Surviving: 7269852 (98.24%) Dropped: 130303 (1.76%)
Вес файла изменился с 174943971 б до 172296846 б
Чистка чтений. С концов чтений были удалены нуклеотиды с качеством ниже 20 и остались только чтения длиной не меньше 32 нуклеотидов. trimmomatic рассказала, что:
Input Reads: 7269852 Surviving: 6974267 (95.93%) Dropped: 295585 (4.07%)
Вес файла изменился с 172296846 б до 162990479 б
| Качество чтений без адаптеров до триммирования |
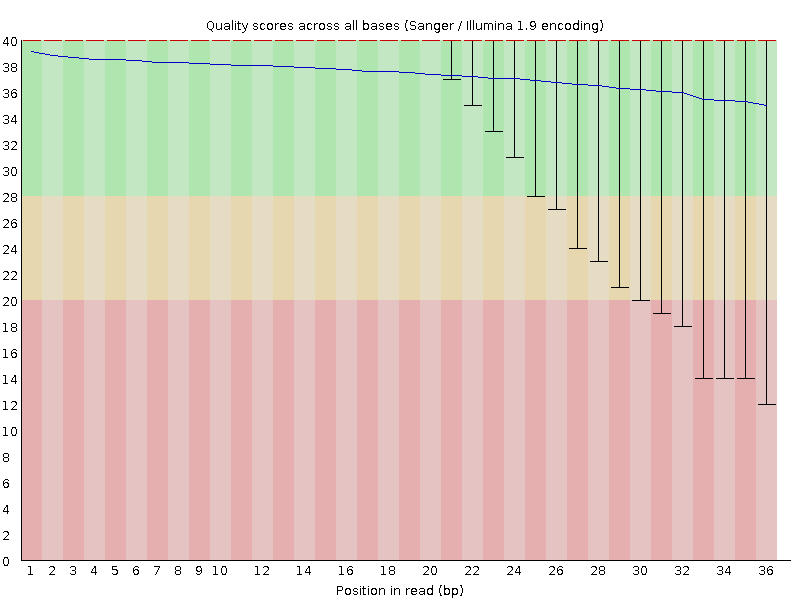 |
| Качество чтений без адаптеров после триммирования |
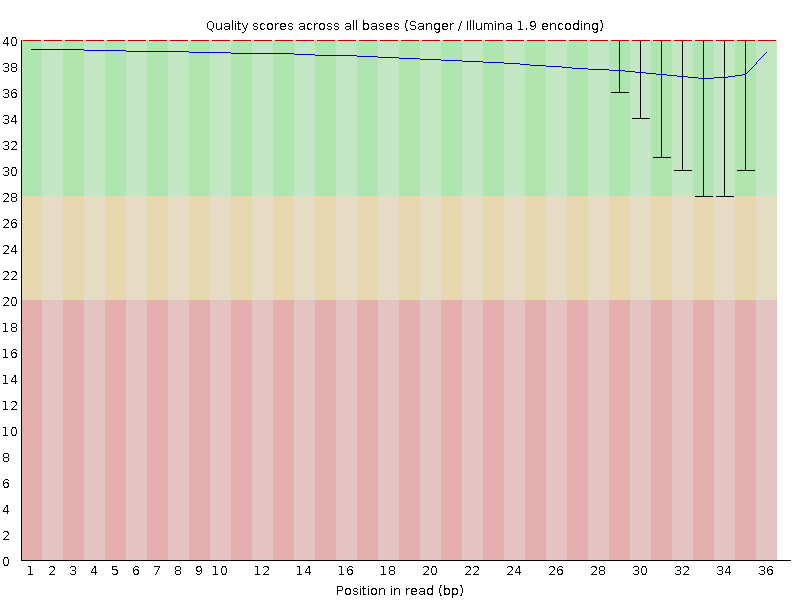 |
Сборка контигов
Сборка контигов на основании триммированных чтений была произведена программой Velvet:
Команда velveth была использована для создания 31-меров на основании чтений.
Далее была запущена команда velvetg для сборки de novo на основе k-меров:
Итогом выполнения команд является информация о N50 (25646), а также файлы contigs.fa (контиги) и stats.txt (характеристики контигов).
3 самых длинных контига:
ID: 6 Длина: 49912 (49942 нукл) покрытие: 35.907238
ID: 9 Длина: 49262 (49292 нукл) покрытие: 34.772179
ID: 5 Длина: 33085 (33115 нукл) покрытие: 36.259029
Контиги с аномальным покрытием:
>ID: 105 Длина: 1 (31 нукл) покрытие: 2694
>ID: 133 Длина: 1 (31 нукл) покрытие: 474299
Оба этих контига имеют длину в 31 нуклеотид и, возможно, являются участками низкой сложности или повторами, часто встречающимися в геноме, или же это шум. Они отсутсвуют в contigs.fa.
Анализ
Запускаем megablast, введя две последовательности: контиг и геном штамма BAg бактерии Buchnera aphidicola (Aphis glycines) (AC: CP009253). Проанализируем каждое выравнивание.
Контиг 6 выравнивается на референсный геном бактерии 5 раздельными фрагментами:
| Координаты в геноме | Число однонуклеотидных различий | Число гэпов |
| 127825-140555 | 2715 | 552/13012(4%) |
| 153752-161738 | 1552 | 270/8171(3%) |
| 144368-151796 | 1426 | 247/7538(3%) |
| 161898-166752 | 898 | 104/4910(2%) |
| 166750-173180 | 1399 | 153/6514(2%) |
| Dot Plot выравниваний | ||
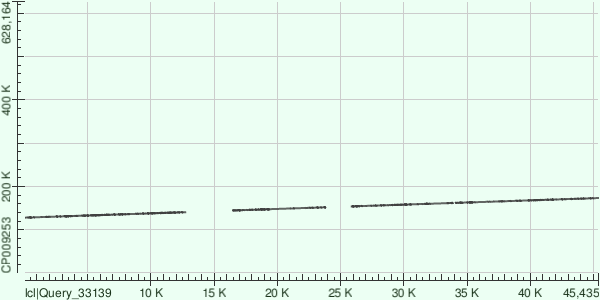 |
||
Контиг 9 выравнивается на референсный геном бактерии 10 раздельными фрагментами:
| Координаты в геноме | Число однонуклеотидных различий | Число гэпов |
| 500370-508806 | 1750 | 351/8617(4%) |
| 510438-516539 | 1150 | 187/6234(2%) |
| 523105-528679 | 1109 | 207/5685(3%) |
| 481997-488106 | 1309 | 308/6238(4%) |
| 517766-521500 | 760 | 101/3783(2%) |
| 496111-500325 | 915 | 154/4324(3%) |
| 493487-494864 | 262 | 13/1384(0.9%) |
| 480874-481545 | 102 | 20/686(2%) |
| 528794-529211 | 42 | 26/425(6%) |
| 495033-495148 | 7 | 5/120(4%) |
| Dot Plot выравниваний | ||
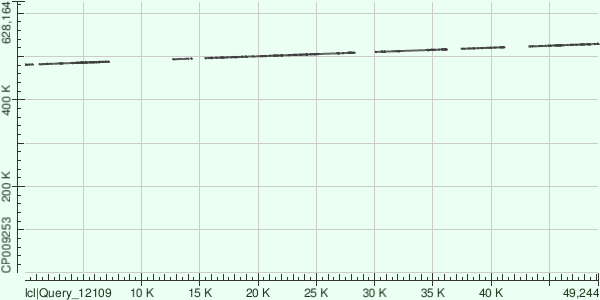 |
||
Контиг 5 выравнивается на референсный геном бактерии 4 раздельными фрагментами:
| Координаты в геноме | Число однонуклеотидных различий | Число гэпов |
| 467412-474667 | 1489 | 208/7388(2%) |
| 462496-467421 | 992 | 162/5015(3%) |
| 474844-480660 | 1288 | 255/5974(4%) |
| 451729-454069 | 488 | 55/2370(2%) |
| Dot Plot выравниваний | ||
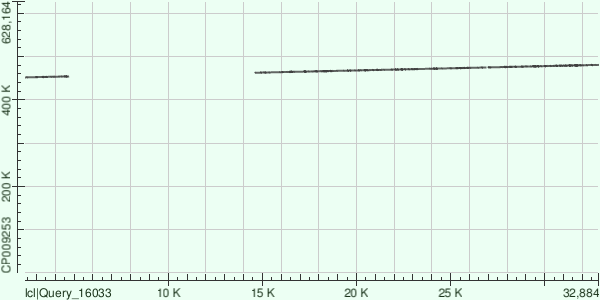 |
||