Комплексы ДНК-белок
Предсказание вторичной структуры заданной тРНК
Ниже представлено сравнение реальной и предсказанной вторичных структур тРНК из файла 1QRT.pdb. Структуры были получены на основе результатов программ find_pair, einverted и RNAfold из пакета EMBOSS
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера (RNAfolder) |
| Акцепторный стебель |
5'-2-7-3'
5'-66-71-3'
всего 6 пар
|
предсказано 7 пар из 6 реальных |
предсказано 7 пар из 6 реальных |
| D-стебель |
5'-10-12-3'
5'-23-25-3'
всего 3 пар
|
предсказано 0 пары из 5 реальных |
предсказано 3 пар из 5 реальных |
| T-стебель |
5'-49-53-3'
5'-61-65-3'
всего 5 пар
|
предсказано 0 пар из 5 реальных |
предсказано 5 пар из 5 реальных |
| Антикодоновый стебель |
5'-37-44-3'
5'-26-33-3'
всего 7 пары
|
предсказано 0 пар из 7 реальных |
предсказано 5 пары из 7 реальных |
| Общее число канонических пар нуклеотидов |
18 |
7 |
20 |
На рисунке 1 показана предсказанная с помощью RNAfolder структура т-РНК. В Антикодоновом стебле есть одна лишняя U-A (поэтому 7/6 пар).
| Рис. 1: Лучшая структура, построенная с помощью RNAfolder |
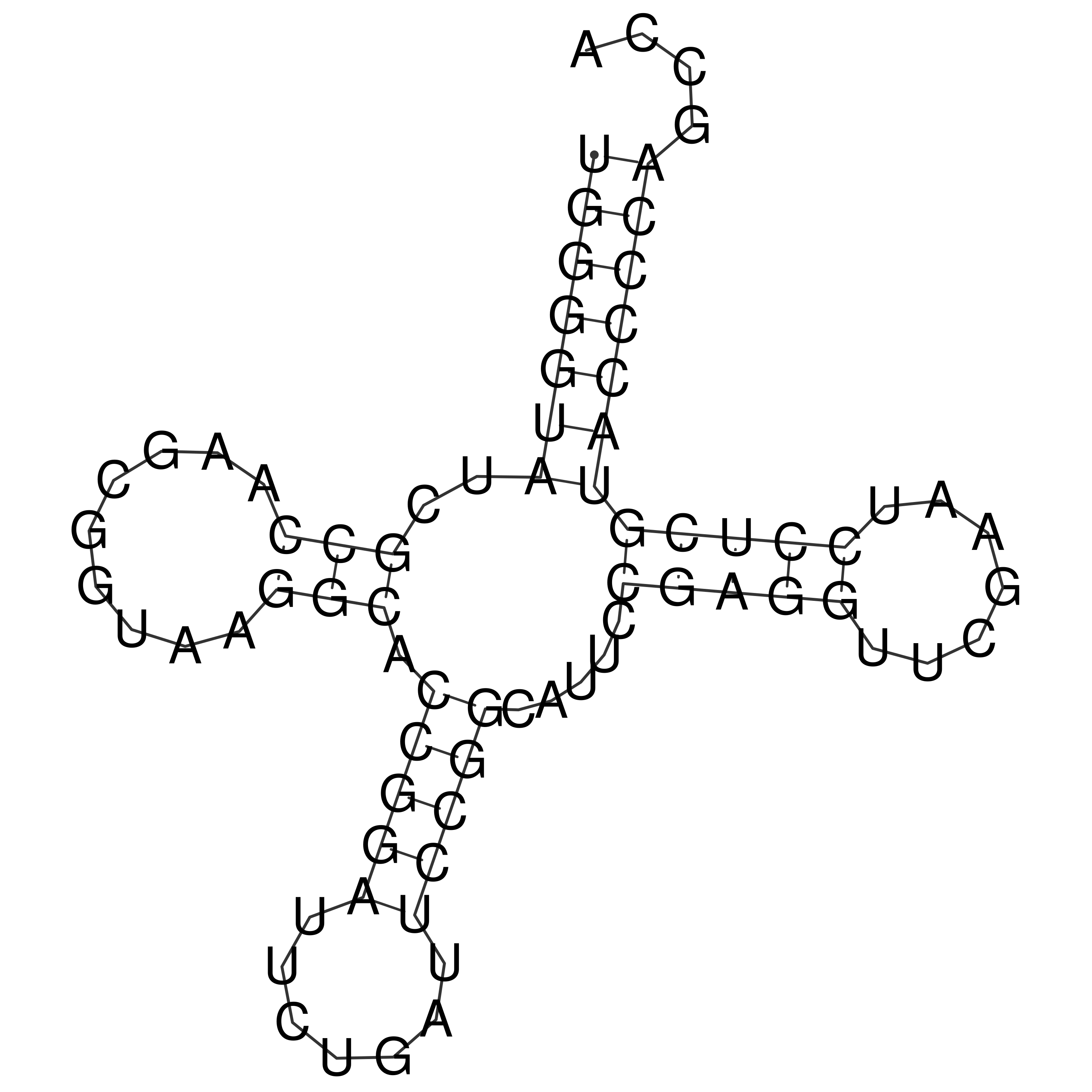 |
Поиск ДНК-белковых контактов в заданной структуре
Визуализация молекулы ДНК в JMol
(PDB: 1KSX)Были созданы два скрипта для JMol: скрипт, задающий множество атомов кислорода дезоксирибозы как "set1", множество атомов килорода фосфатных остатков ДНК как "set2" и множество атомов азота в азотистых основаниях как "set3" (ссылка на скрипт) и скрипт, последовательно показывающий всю исследуемую структуру (цепь A и ДНК); только молекулу ДНК в проволочной модели; в этой модели выделены шариками только атомы из set1; выделены шариками только атомы из set2; выделены шариками только атомы из set3 (ссылка на скрипт).
Визуализация контактов в JMol
Взяв за полярные атомы атомы кислорода и азота, за неполярные углерода, фосфора и серы, за полярный контакт ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3,5A, а за неполярный ананогичную ситуацию для неполярных, но с расттоянием меньше 4,5A был создан скрипт для JMol последовательно визуализирующий для исследуемой структуры атомы дезоксирибозы; атомы фосфатных остатков ДНК; атомы азотистых оснований, направленные в большую бороздку; атомы азотистых оснований, направленные в малую бороздку; полярные и неполярные контакты белка с перечисленными множествами атомов (ссылка на скрипт). С помощью этого скрипта был получен следующий результат:
| Контакты атомов белка с |
Полярные: |
Неполярные: |
Всего: |
| остатками 2'-дезоксирибозы |
4 |
89 |
93 |
| остатками фосфорной кислоты |
63 |
28 |
91 |
| остатками азотистых оснований со стороны большой бороздки |
0 |
59 |
59 |
| остатками азотистых оснований со стороны малой бороздки |
0 |
1 |
1 |
По этой ссылке представлена схема ДНК-белковых контактов, полученная с помощью программы nucplot.
По полученной схеме были выбраны: Phe162(I) , так как у него самое большое количество связей (6) среди остальных аминокислот., тем более Phe162(I) тремя связями связывается непосредственно с тимином, что должно увеличивать специфичность распознавания. Также, непосредсвенно с азотистым основанием связывается Val462(A) (рис. 2) одной связью.
| Рис 2. Предполагаемые контакты Phe162(I) с ДНК |
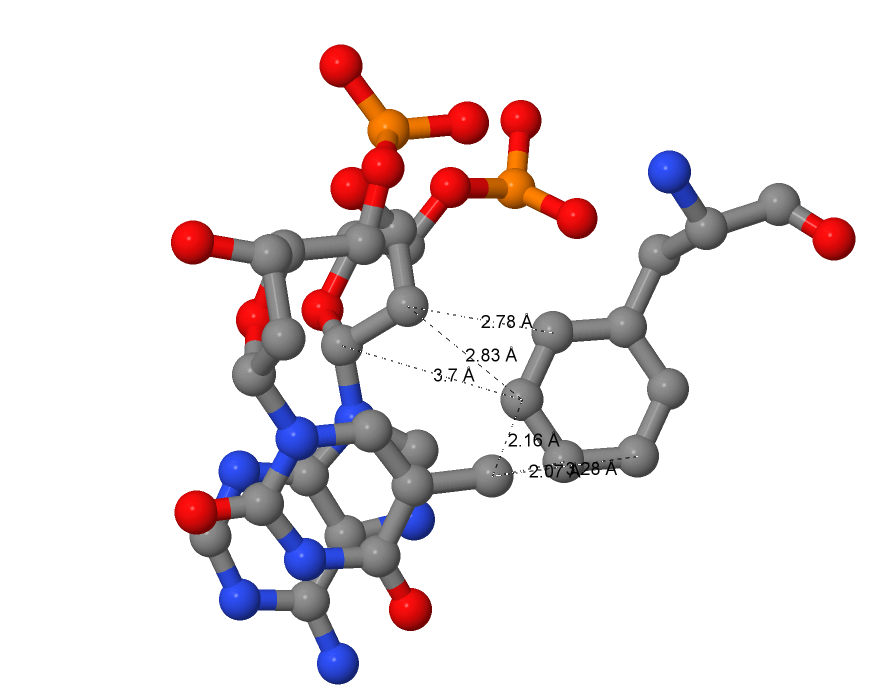 |
|

