Сигналы и мотивы, часть 3
Консервативный мотив в выравнивании последовательностей гомологичных белков
Для выполнения задания был взят домен LAP1_C (PF05609) с seed=22. Был выбран консервативный по всем последовательностям мотив E-[TIV]-E-E-K-V-R-D-[LF]-L в выравнивании 22 последовательностей. С помощью MyHits было получено 8 находок в базе данных SwissProt и они пренадежат к семейству LAP1.
 |
| Рис .1: Выравнивание с выделенным выбранным мотивом. |
Поиск мотива специфичного для клады
С помощью метода NJ в JalView было построено филогенетическое дерево:
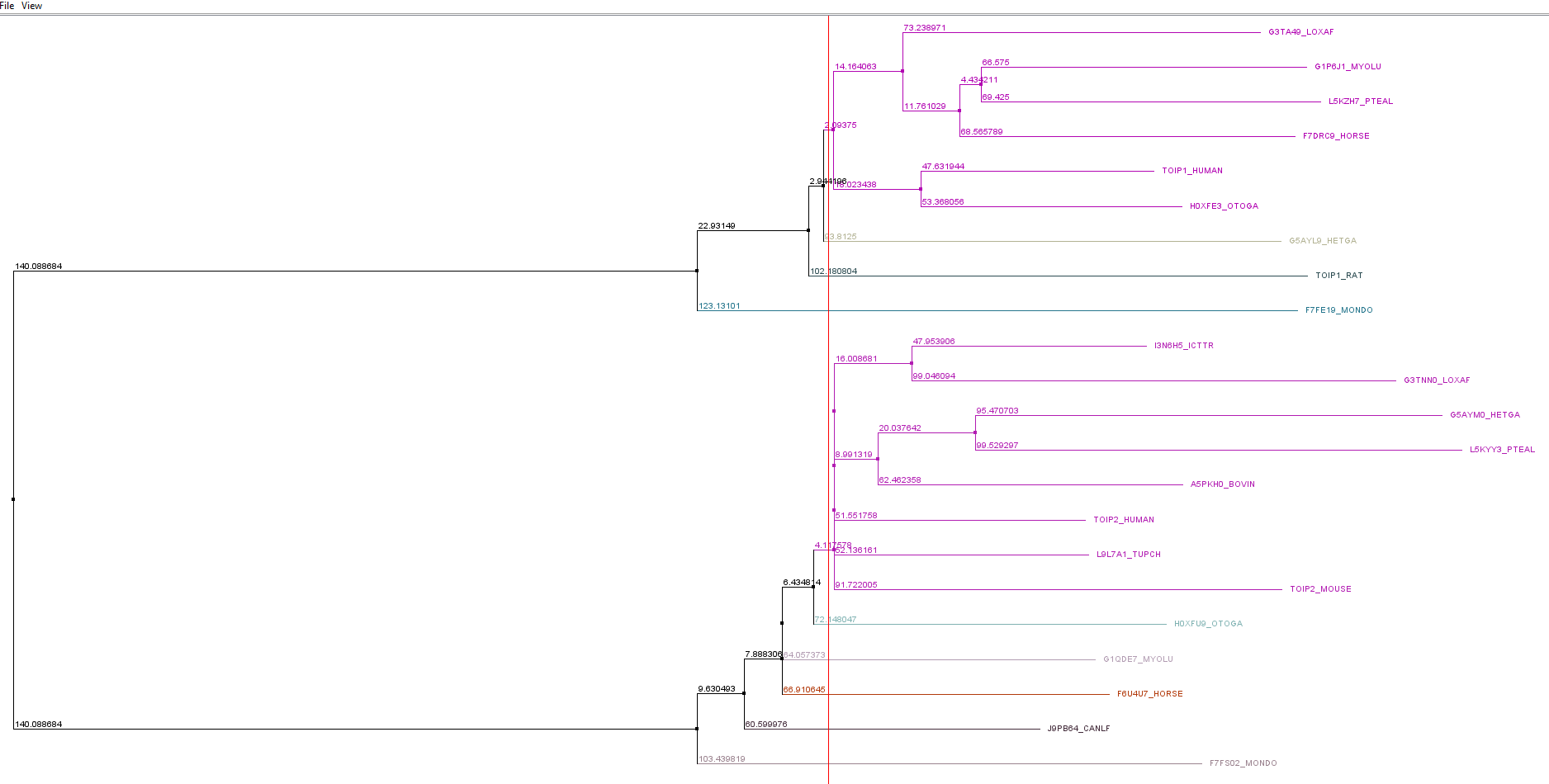 |
| Рис. 2: Филогенетическое дерево. |
Была выбрана клада из 6 верхних белков. Далее был найден характерный только для неё мотив: E[VA]E[TG]T.
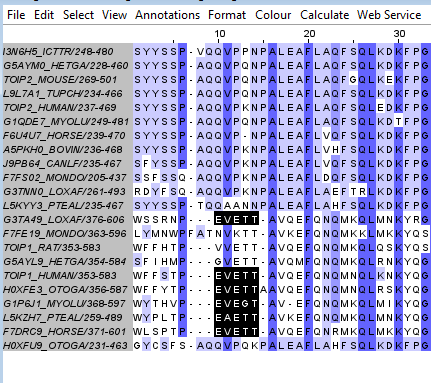 |
| Рис. 3: Характерный для клады паттерн |
PSI-BLAST
Был выбран белок C4Z088 - ингибитор клеточного деления Eubacterium eligens ATCC 27750.
| Номер итерации |
Число находок выше порога (0,005) |
Идентификатор худшей находки выше порога |
E-value этой находки |
Идентификатор лучшей находки ниже порога |
E-value этой находки |
| 1 |
162 |
Q2P036.1 |
0.004 |
Q5F5V4.1 |
0.005 |
| 2 |
188 |
O25693.2 |
2e-09 |
- |
- |
| 3 |
188 |
Q9ZM51.1 |
1e-11 |
7H8E6.1 |
0.012 |
| 4 |
188 |
Q9ZM51.1 |
5e-13 |
A7H8E6.1 |
0.016 |
Уже на третьей итерации число находок с E-value меньше порогового перестало изменяться, а разница между худшим результатом выше порога и лучшим результатом ниже порога составила значительную величину (9 порядков). Это позволяет нам сделать вывод о том, что группа является хорошо обособленной. Следовательно, выявленное семейство белков обосновано сходством их последовательностей.
Подсчет числа ТА в геноме
Ожидаемое число ТА составило 336256, реальное - 218026 (64,8%).
|