Трансмембранные белки
Характеристики данного белка
Знакомство с базой данных OPM
В базе данных OPM был найден белок 7pge (PCOB_ECOLX), являющийся транспортером меди внешней мембраны E.coli. Требуется для индуцируемого медью экспресии сопротивления меди. Белок содержит бэта-бочонок из 12 трансмембранных бэта-листов.
| Тип |
Трансмембранный |
| Класс |
Трансмембранный бэта-бочонок |
| Суперсемейство |
KdgM/CopB (n=12,S=14) |
| Семейство |
Порин, резистивный к меди |
| Организм |
Escherichia coli |
| Толщина гидрофобной части |
22.4 A |
| Координаты трансмембранных участков |
1( 92- 102), 2( 110- 118), 3( 125- 131), 4( 142- 149), 5( 155- 163), 6( 173- 181), 7( 188- 193), 8( 200- 210), 9( 218- 226),10( 242- 251),11( 260- 267),12( 286- 294) |
| Среднее количество остатков в одном бэта-тяже белка |
12 |
| Локализация |
Внешняя мембрана Грам-отрицательных бактерий |
Ниже представленна вторичная структура белка 7pge. Синим показана n-сторона, красным - p-сторона.
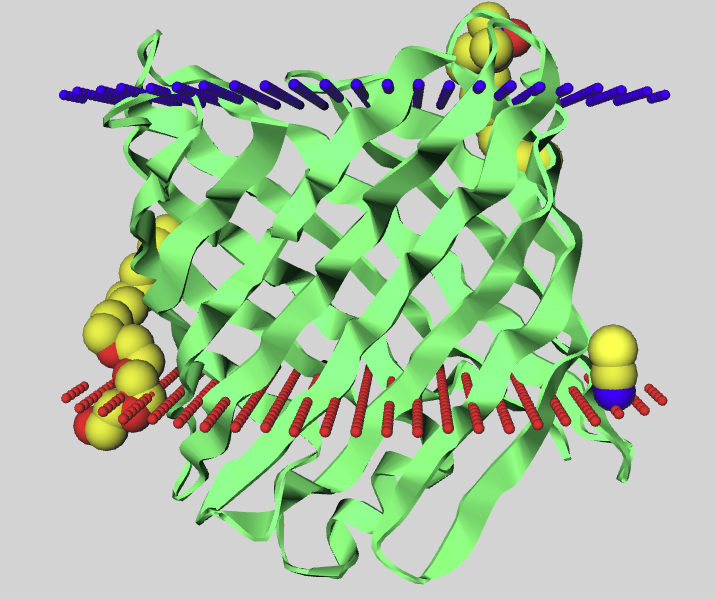 |
| Рис. 1: Вторичная структура белка 7pge |
DeepTMHMM: Предсказание трансмембранных элементов по последовательности белка
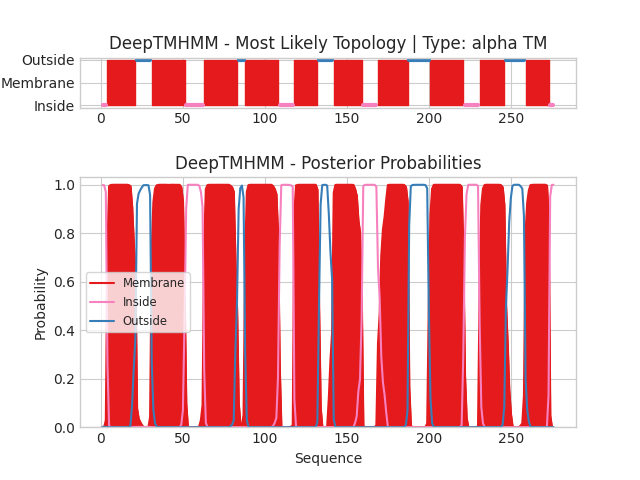
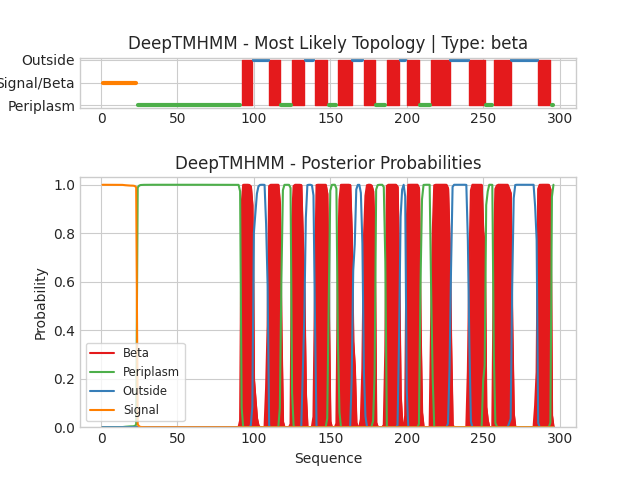
Сервис DeepTMHMM предлагает возможность прогнозировать трансмембранные домены на основе первичной структуры белка. Графический результат отображает координаты аминокислотных остатков по горизонтальной оси, а предсказанные положения определенных доменов (вверху) и вероятности принадлежности конкретных остатков к различным топологиям (внизу) отмечаются на вертикальной оси. Ниже представленны графические и текстовые выдачи для выданного альфа-спирального белка (Y266_ARCFU) и для выбранного бэта-листового белка (PCOB_ECOLX) соответспенно:
Цвет показывает определенную локализацию участка. В первом случае: Membrane - трансмембранный, Inside - внутриклеточный, Outside - внеклеточный. Во втором случае: Beta - бета-слой (мембранный), Periplasm - периплазматический (аналог внутриклеточной части), Outside - внеклеточный, Signal - сигнальный пептид для периплазматической локализации.
Для выданного белка было предсказано 10 трансмембранных участков (альфа-спиралей). N- и C-конецы белка согласно предсказанию являются внутриклеточными.
Для выбранного белка было предсказано 12 трансмембранных участков (бэта-слоев). N-конец белка является сигнальным пептидом, за ним согласно предсказанию следует периплазматический участок, а С-конец - внеклеточным. Предсказания не совпадают с OPM, однако это из-за того, что в выдаче учитывается сигнальный пептид. Поэтому координаты всех предсказанных бэта-слоев смещаются на его длину пептида.
PPM: Предсказание положения выданного белка в мембране
В данном разделе проведено предсказание топологии для выданного альфа-спирального с помощью сервиса PPM 3.0 на сайте OPM. Параметры запуска:
Number of Membranes: 1
Type of membrane: Archeabacteria cell membrane (по таксономии из UniProt архея)
Allow curvature: no (ибо сложности ни к чему)
Topology (N-ter): in (по предсказанию DeepTMHMM)
Для этого белка не существует достоверной структуры, однако существует ее модель, которая предсказана с помощью AlphaFold. Программе получила на вход файл с предсказанной структуры в формате pdb. С полученной моделью можно ознакомиться по ссылке.
| Толщина гидрофобной части (Глубина) |
30.8 ± 0.9 A |
| delta G transfer |
-75.3 kcal/mol |
| Угол изгиба |
8 ± 1° |
| Трансмембранные сегменты |
1( 3- 25), 2( 30- 51), 3( 60- 86), 4( 87- 105), 5( 115- 131), 6( 138- 159), 7( 168- 188), 8( 200- 223), 9( 228- 250),10( 256- 273) |
| Средняя длина трансмембранного сегмента |
|
Ниже представлена предсказанная структура белка:
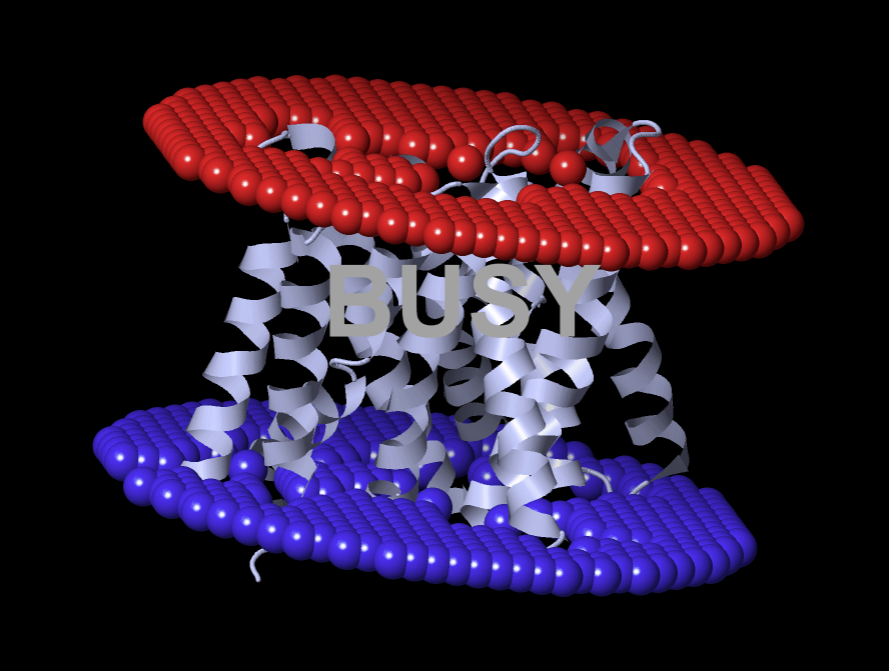 |
| Рис. 4: Предсказание положения Y266_ARCFU в мембране. Синим показана n-сторона, красным - p-сторона. |
Сравнение алгоритмов предсказания трансмембранных участков
| DeepTMHMM |
| PPM |
| Предсказанные трансмембранные участки: |
| Предсказанные трансмембранные участки: |
| 1( 4-21), 2( 31-51), 3( 63-83), 4( 88-108), 5( 118-132), 6( 142-159), 7 ( 169-187), 8( 201-221), 9( 231-246), 10( 259-273) |
| 1( 3- 25), 2( 30- 51), 3( 60- 86), 4( 87- 105), 5( 115- 131), 6( 138- 159), 7( 168- 188), 8( 200- 223), 9( 228- 250),10( 256- 273) |
Сравнение результатов двух моделей прогнозирования структуры альфа-спирального белка показывает значительное совпадение. Несмотря на разницу в пару аминокислотных остатков, общее количество и расположение спиралей совпадают. Возможно это из-за того, что в AplhaFold структуре некоторые спирали частично имеют доверительный балл 90 > pLDDT > 70:
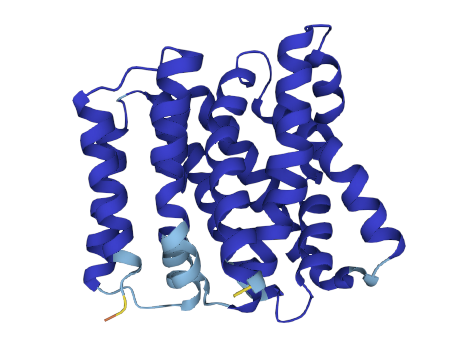 |
| Рис. 4: AplhaFold структура выданного белка Y266_ARCFU. Тёмно-синий - pLDDT > 70; Голубой - 90 > pLDDT > 70 |
|