Визуализация деревьев
Эталонное дерево
Для срвнения результатов этого практикума было взято эталонное дерево:
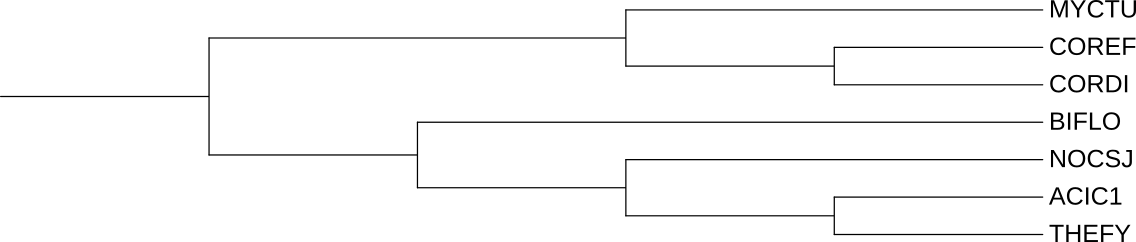 |
| Рис 1.: Эталонное филогенетическое дерево выбранных бактерий |
Составление списка гомологичных белков, включающих паралоги
Из директории /P/y21/term4/Proteomes были скачаны fasta файлы, содержащие протеомы выбранных бактерий. Они были объеденены в один файл с протеомами. Далее командами
makeblastdb -in odnako.fasta -dbtype prot -parse_seqids
blastp -query P0A6H1.fasta -db odnako.fasta -out final.tab -evalue 0.001 -outfmt 7
были произведены индексация файла и поиск гомологов белка CLPX_ECOLI в файле (порог для e-value = 0,001). С помощью выдачи команды был составлен fasta файл, на основе которого строилось дальнейшее дерево.
Реконструкция и визуализация
С помощбю программы FastME на сайте ngphylogeny.fr c параметрами: 'Gamma distributed rates across sites' — No, 'Starting tree' — BIONJ, 'No refinement', 100 бутстреп реплик ыло реконструированно дерево найденных гомологов.
Полученное дерево в формате Newick сохранено в файле.
Считая дерево реконструированным верно, можно привести примеры ортологов и паралогов:
Ортологи: CLPX_COREF и CLPX_CORDI, Q8FMH5_COREF и Q6NFB1_CORDI, A0LRB8_ACIC1 и Q47KU4_THEFY.
Паралоги: FTSH_ACIC1 и A0LRB8_ACIC1, RUVB_BIFLO и Q8G3S2_BIFLO, RUVB_NOCSJ и A1SDV1_NOCSJ.
Далее, после укоренения дерева опцией Midpoint root в разделе Advanced, были покрашены большие ортологичные группы белков. Маленькие группы (ортологов три и меньше) были оставлены чёрными. Поручившее дерево продемонстрированно на рисунке ниже:
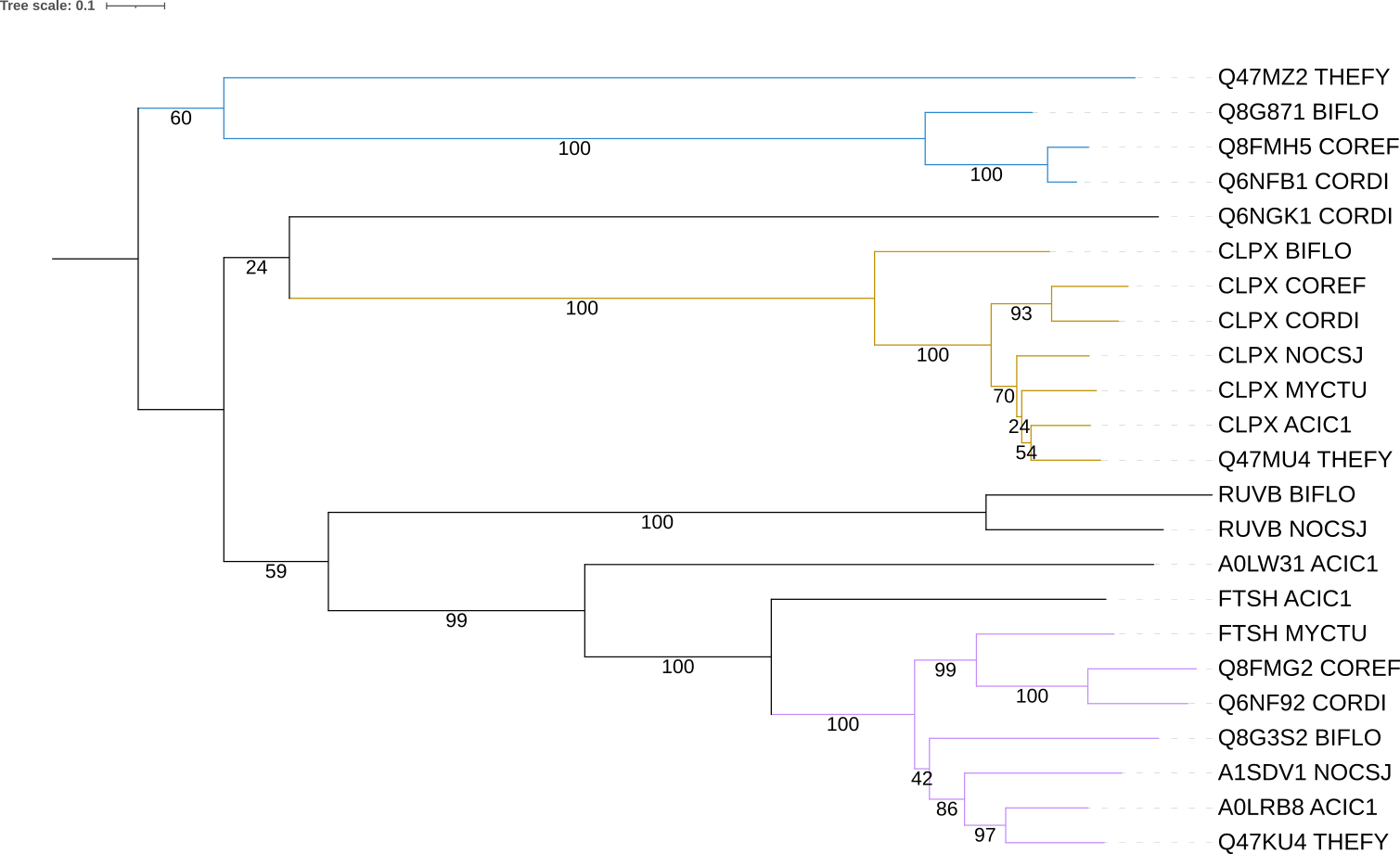 |
| Рис 2.: Дерево гомологов CLPX_ECOLI. цветами выделены большие ортологичные группы. |
Иронично, но окрашенная сиреневым клада полностью совпадает с эталонным деревом, хотя все белки из этой клады являются АТФ-зависимыми цинк-металлопротеазами FtsH. В золотой кладе, где расположены АТФ-зависимые протеазы Clp лишь ветви (CLPX_COREF, CLPX_CORDI) и (CLPX_ACIC1, Q47MU4_THEFY) совпадают с эталонным деревом, всё остальное нет. В синей кладе присутсвуют разные белки (лишь Q6NFB1_CORDI является АТФ-зависимой протеазой Clp) и в ней не присутсвуют все из семи выбранных бактерий, но есть ветвь (Q8FMH5_COREF, Q6NFB1_CORDI) (Q8FMH5_COREF - Предполагаемая эндопептидаза Clp АТФ-связывающая цепь C). Все эти клады были "схлопнуты", рисунок с соответсвующим деревом представлен ниже:
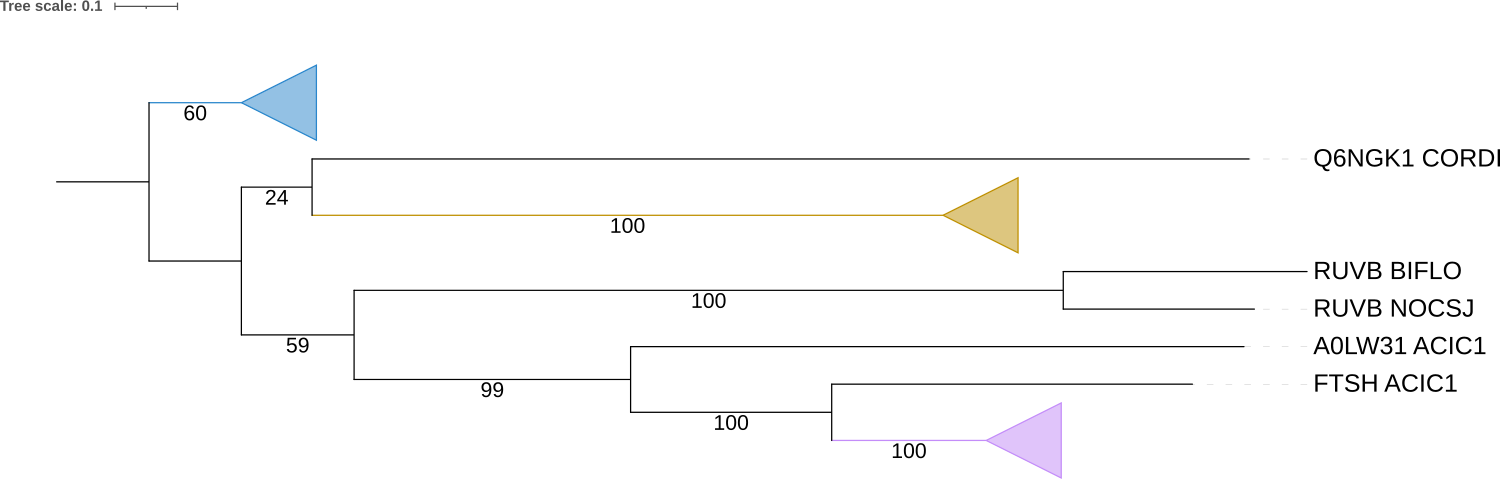 |
| Рис 3.: Дерево, на котором ортологичные группы с соответствующими цветами "схлопнуты". |
|


