Задание 2
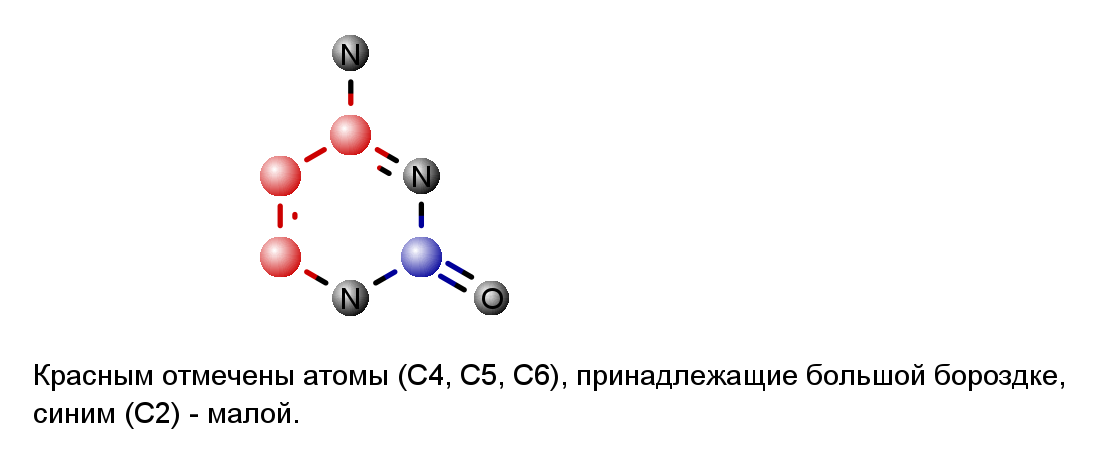
Я рассматривала Z-форму ДНК, так как она наименее привычна из всех трех. На приведенном изображении цитозина отмечено, какие атомы принадлежат какой бороздке.
-
С помощью программы fiber в пакете 3DNA построены A-, B-, и Z-формы фрагмента GATC. Результат программы лежит по адресу ~/term3/block1/pr2.
Я рассматривала Z-форму ДНК, так как она наименее привычна из всех трех. На приведенном изображении цитозина отмечено, какие атомы принадлежат какой бороздке.

| Признак | A-форма | B-форма | Z-форма |
|---|---|---|---|
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.2 | 33.2 | 45.4 |
| Число оснований на виток | 11 | 10.5 | 12 |
| Ширина большой бороздки | 18.5 ([A]30:B.P-[T]15:A.P) | 20.6 ([C]4:A.P-[C]32:B.P) | 29.3 ([C]4:A.P-[G]31:B.P) |
| Ширина малой бороздки | 9.6 ([C]4:A.P-[A]30:B.P) | 13.2 ([C]32:B.P-[A]14:A.P) | 14.3 ([G]31:B.P-[C]16:A.P) |
1) Торсионные углы нуклеотидов
Файл trna4.out содержит графу о торсионных углах:
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- --- 152.9 84.9 -159.0 -63.7 165.9
2 G -107.1 -135.5 58.5 84.9 -172.7 -69.2 -153.2
3 G -85.2 -158.6 55.0 84.1 -153.0 -63.4 -157.4
4 C -67.6 173.2 49.6 78.6 -154.7 -65.1 -154.4
5 U -48.8 173.0 54.4 82.3 -155.6 -87.2 -151.5
6 U -66.3 179.2 48.3 84.1 -166.4 -69.9 -167.1
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C 152.9 -171.3 171.5 80.7 -134.5 -69.5 -172.0
2 C -62.3 178.5 52.6 81.6 -165.8 -75.7 -134.9
3 C -76.1 -170.0 48.0 82.8 -154.0 -64.8 -149.2
4 G -61.4 -178.3 41.9 81.4 -166.8 -70.3 -159.6
5 G 176.1 146.4 165.7 83.4 -138.4 -64.0 -173.5
6 A -66.9 -178.2 54.2 82.6 -177.7 -30.4 -162.8
- фрагмент файла.
2) Форма НК
В файле также есть таблица, отражающая форму спиралей.
Classification of each dinucleotide step in a right-handed nucleic acid
structure: A-like; B-like; TA-like, or other cases.
Согласно этой таблице, моя tRNA имеет А-форму.
Фрагмент файла:
step Xp Yp Zp XpH YpH ZpH Form
1 GG/CC -1.86 8.28 1.68 -5.91 7.48 3.91 A
2 GG/CC -1.72 8.36 1.62 -5.19 6.84 5.01 A
3 GC/GC -1.67 8.58 2.01 -4.26 8.17 3.29 A
Фрагмент файла с необходимыми данными:
Overlap area in Angstrom^2 between polygons defined by atoms on successive
bases. Polygons projected in the mean plane of the designed base-pair step.
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 1.65( 0.32) 0.00( 0.00) 0.54( 0.00) 0.39( 0.00) 2.58( 0.32)
2 GG/CC 1.74( 0.06) 0.00( 0.00) 0.00( 0.00) 2.70( 1.04) 4.45( 1.10)
3 GC/GC 7.17( 4.47) 0.00( 0.00) 0.00( 0.00) 4.11( 1.43) 11.28( 5.89)
4 CU/GG 0.93( 0.27) 0.00( 0.00) 0.06( 0.00) 3.30( 1.87) 4.29( 2.14)
5 UU/AG 0.08( 0.00) 0.00( 0.00) 1.49( 0.00) 1.29( 1.10) 2.87( 1.10)
6 UG/CA 0.06( 0.00) 0.00( 0.00) 2.69( 1.23) 0.08( 0.00) 2.83( 1.23)
7 GG/UC 1.57( 0.31) 0.00( 0.00) 1.38( 0.01) 0.00( 0.00) 2.96( 0.32)
8 GG/CU 2.57( 1.04) 0.00( 0.00) 0.45( 0.00) 0.14( 0.00) 3.16( 1.04)
9 GU/AC 5.02( 2.21) 0.00( 0.00) 0.00( 0.00) 5.59( 4.53) 10.61( 6.74)
10 UG/CA 0.08( 0.00) 0.00( 0.00) 2.72( 1.52) 0.25( 0.00) 3.05( 1.52)
11 GG/CC 3.50( 1.74) 0.00( 0.00) 1.65( 0.07) 0.00( 0.00) 5.15( 1.81)
12 GU/AC 6.79( 4.21) 0.00( 0.00) 0.00( 0.00) 3.91( 1.50) 10.70( 5.71)
13 UU/GA 7.21( 4.25) 0.00( 0.00) 0.00( 0.00) 4.91( 2.57) 12.13( 6.82)
14 UU/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Наибольшее перекрывание между 3 GC/GC, 9 GU/AC, 12 GU/AC, 13 UU/GA, 15 UA/CU, 19 GU/AC, 23 GC/GC парами нуклеотидов
Изображение 12-13 нуклеотидов:
