Задание 1
Геном манула не аннотирован, поэтому я работаю с аннотированным геномом довольно близкой к нему рыси - сборка mLynCan4.pri.v2 (GCF_007474595.2), Lynx canadensis (Canada lynx)
Я выбрала CDS белка SAP8, Sin3A associated protein 18 [Source:NCBI gene;Acc:115508722], Primary_assembly A1: 2,084,187-2,090,085 forward strand.
Находится на хромосоме A2, координаты 2,084,187..2,090,085; длина - 6030 нуклеотидов, длина белка - 172 аминокислоты.
GeneBank AC: NC_044303
Сплайсинг проходит по схеме join(21..149,332..441,5245..5367,5627..5783)
Используемый мной фрагмент хромосомы - эта CDS с припусками по обеим концам, координаты - 2084165..2090330, ограничена маркерами по обеим сторонам.
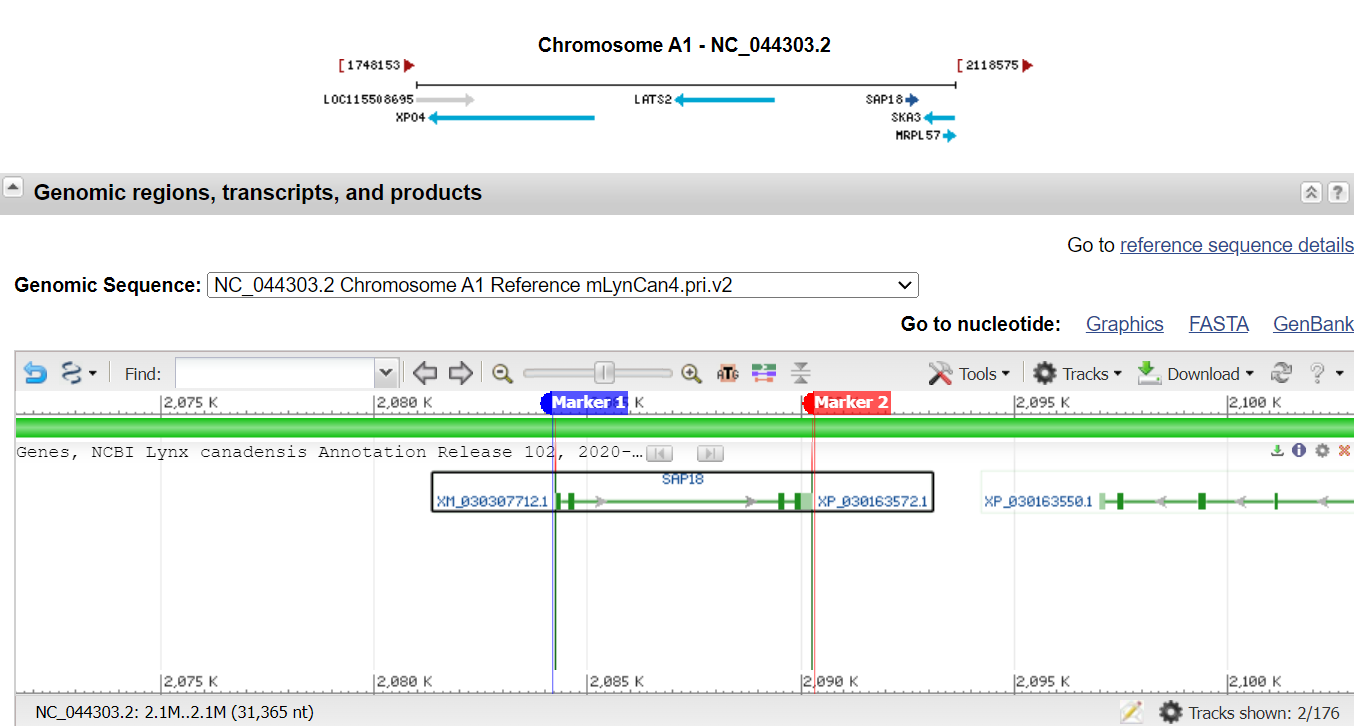
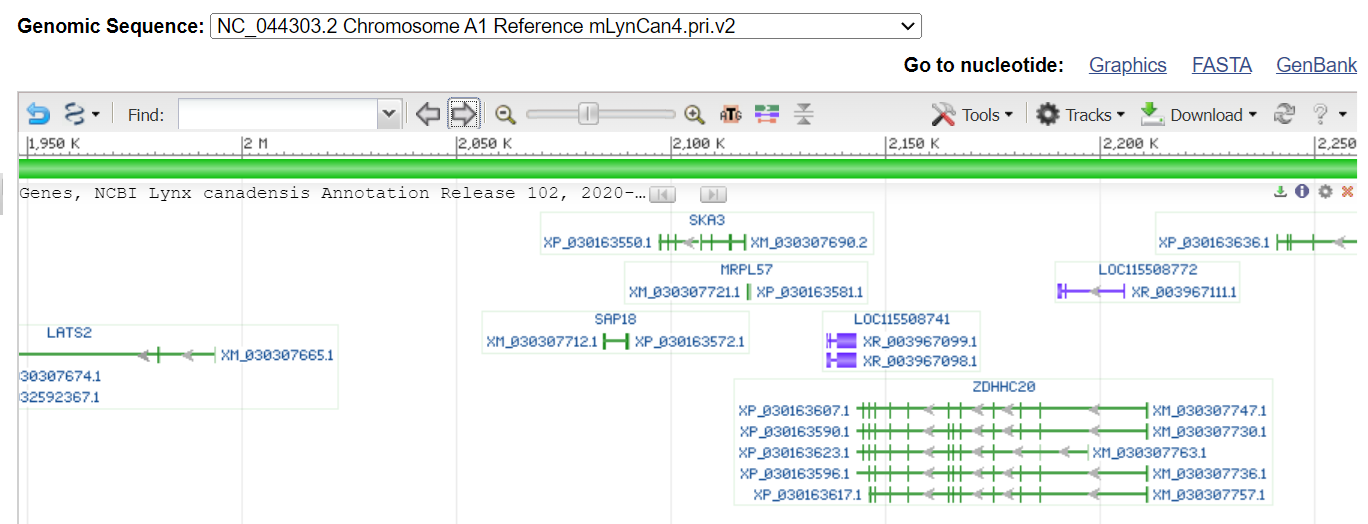
На картинках выше мы видим, что в нашей анализируемой последовательности только CGS SAP8, но рядом с ним на большом масштабе лежат другие гены, в основном они гораздо длиннее. Зеленым обозначены protein-coding sequences, фиолетовым - функциональные РНК.
В качестве таксона для сравнения выбрала ехидну - она тоже относится к классу млекопитающие, но к другому подклассу - прототерии
Результаты BLAST:
1) MEGABLAST - нет находок. Этот алгоритм предназначен для поиска высокосходных последовательностей нуклеотидов, поэтому довольно логично, что он не обнаружил сходства между этими организмами. Этот алгоритм хорош, когда надо проанализировать очень близкие последовательности, например, может подойти для выравнивания геномов разных подвидов одного вида
2) Выдача blastn:
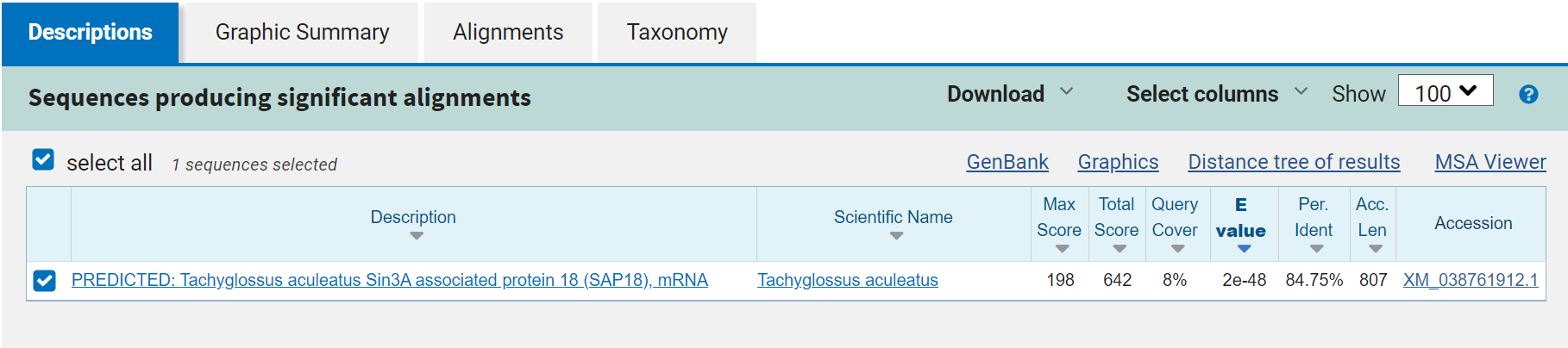
BLASTN ищет гомологичные, похожие последовательности, и он более чувствителен, чем мегабласт. Поэтому очень хорошо и закономерно, что он нашел хотя бы один хит: тот же Sin3A associated protein, только 18 изоформа, а не 8.
На ген выровнялась мРНК, поэтому выравнивание разрывно в местах интронов, но само по себе довольно точное.
3) Выдача blastx:
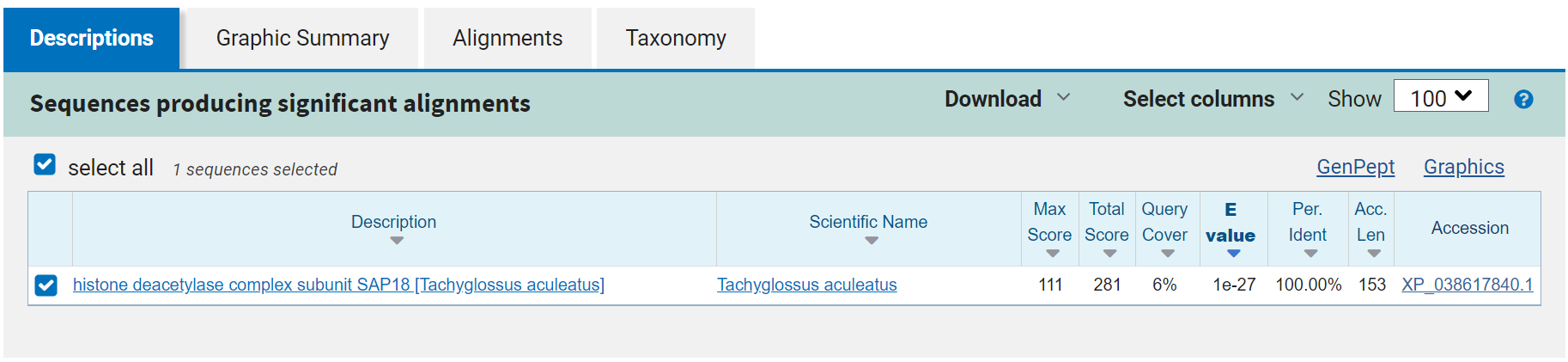
BLASTX ищет сходство между всеми возможными рамками считывания query и банком белков. Он в среднем более чувствительный, так как могли произойти синонимичные замены, не отразившиеся на белке, но попортившие нуклеотидные выравнивания. Используется для поиска более далеких гомологов.
4) Выдача tblastn:
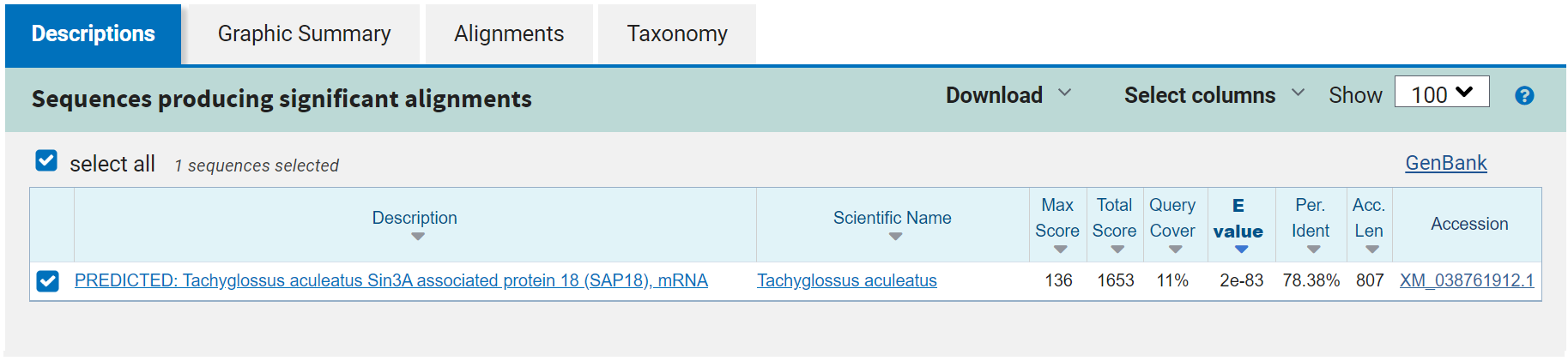
TBLASTN сначала транслирует query, потом сравнивает ее с нуклеотидами базы. Он так же нужен для поиска более дальних гомолов.
Я пользовалась параметрами по умолчанию, и меня удовлетворили результаты поиска - организмы довольно далеки друг от друга, так что в этом случае получить в результате одну находку - ожидаемо.
Задание 2
Скачанный предварительно геном рыси проиндексировала для BLAST:
makeblastdb -in genome.fna -dbtype nucl
Команды для работы blastn:
blastn -task blastn -query 16s.fna -db genome.fna -out 16s.out -outfmt 7 -evalue 0.0001
blastn -task blastn -query 23s.fna -db genome.fna -out 23s.out -outfmt 7 -evalue 0.0001
blastn выбран потому что анализируемые последовательности не очень таксономически близки и состоят обе из нуклеотидов. Отсеиваем по e-value=0.001, длина слова - 7, что дает большую чувствительность.
Выравнивание проводим по 16S и 23S рибосомальным РНК
Результаты для 16S РНК - ген, аннотированный как rrna-18s ribosomal rna, что очень ожидаемо - это гомологи, они составляют малую субъединицу рРНК.
Результаты для 23S РНК - а это целых три гена, аннотированны как rrna-28s ribosomal rna и rrna-5.8s ribosomal rna, что тоже очень радует. Эти рРНК входят в большую субъединицу эукариотической рибосомы и действительно гомологичны прокариотической 23S. Есть неспецифичные выравнивания с белками низкого качества, они отсутствуют в бласте с длиной слова 11. Несколько разных РНК появляются вследствии того, что у эукариот есть разные типы рибосом.