Задание 1. Консервативные мотивы в выравнивании
Выбрала NYN domain, entry PF01936. Относится к клану PIN, суперсемейство нуклеаз. Скачала seed моего домена, он включает 142 последовательности.
Выровняла JalView:

После reduse redundancy 90% осталось 140 последовательностей, то есть почти не было сильно схожих.
Дальше поиск мотива: снижаю порог идентичности до 95, появилась первая аминокислота из мотива: D колонка 7
На пороге 90% появились четыре аспарагиновых кислоты в колонках 7, 153, 183 и 185, но мне кажется это недостаточным из-за неспецифичности свойств аминокислоты и разброса
На пороге 81% появляются и ароматические аминокислоты: видим фенилаланин в 189 колонке и тирозин в 76. Еще появился аспарагин в 10

Из этого на мотив тянет скопление 153-185
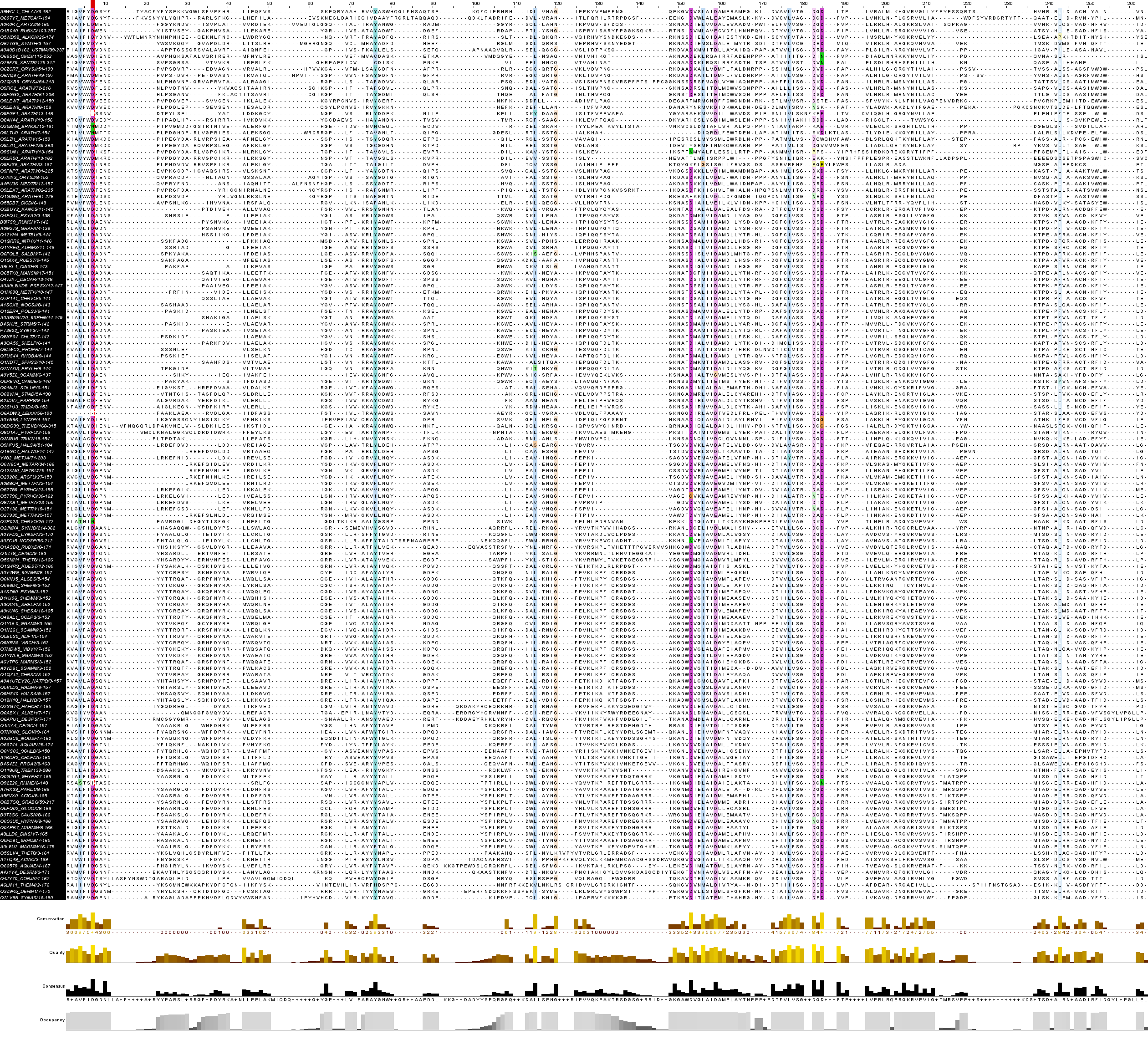
Отсюда предположим, что имеется мотив 153-189 коллонки, с консервативными аспартатами. Это не очень хороший мотив в смысле информационного содержания, вероятность встретить такое случайно достаточно велика. Однако этот домен отвечает за связывание иона, значит важно иметь заряженные аминокислоты в активном центре, аспарагин в принципе подходит на эту роль.
Лого мотива с сайта Pfam выглядит так:

Видим, что хорошо соотносится с найденными нами аминокислотами.
По JalView Consensus/1-38 Percentage Identity Consensus: GKNA+DVGLAIDAMELAYTNPPP+PDTFVLVSG++DGD
Создадим сильный мотив PROSITE, просто изменив синтаксис: G-K-N-A-x-D-V-G-L-A-I-D-A-M-E-L-A-Y-T-N-P-P-P-x-P-D-T-F-V-L-V-S-G-x-x-D-G-D
Поиск в PROSITE дал no hits..
Но мы будем считать что отсутствие результата это тоже результат. Мотив, во-первых, слишком длинный, во-вторых, слишком детерминированный. Возьмем мотив покороче и послабее.
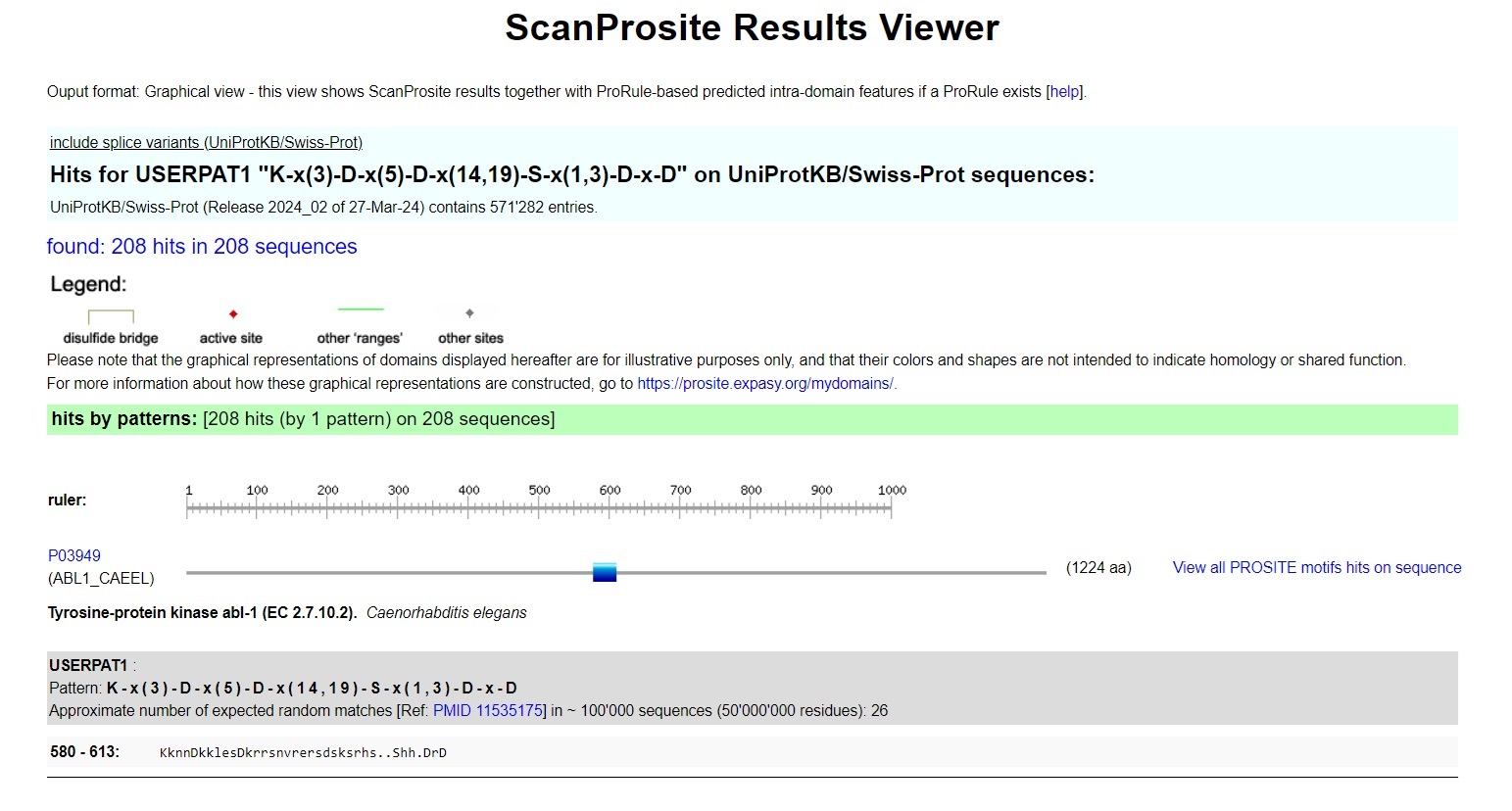
Я поставила порог Identity 70%, все выделившиеся аминокислоты дают мотив K-x(3)-D-x(5)-D-x(14,19)-S-x(1,3)-D-x-D
По базе seed (искала с помощью PROSITE) получилось 77 hits in 77 sequences, что не очень хорошо - общая мощность базы 142, то есть найдено примерно в половине последовательностей. Но для такого несильного мотива это ок
Получила 208 hits in 208 sequences. Довольно слишком много, значит, мотив получился и строгий, и не специфичный.
Approximate number of expected random matches [Ref: PMID 11535175] in ~ 100'000 sequences (50'000'000 residues): 26 - это не очень плохо.
Задание 2. Подсемейство.
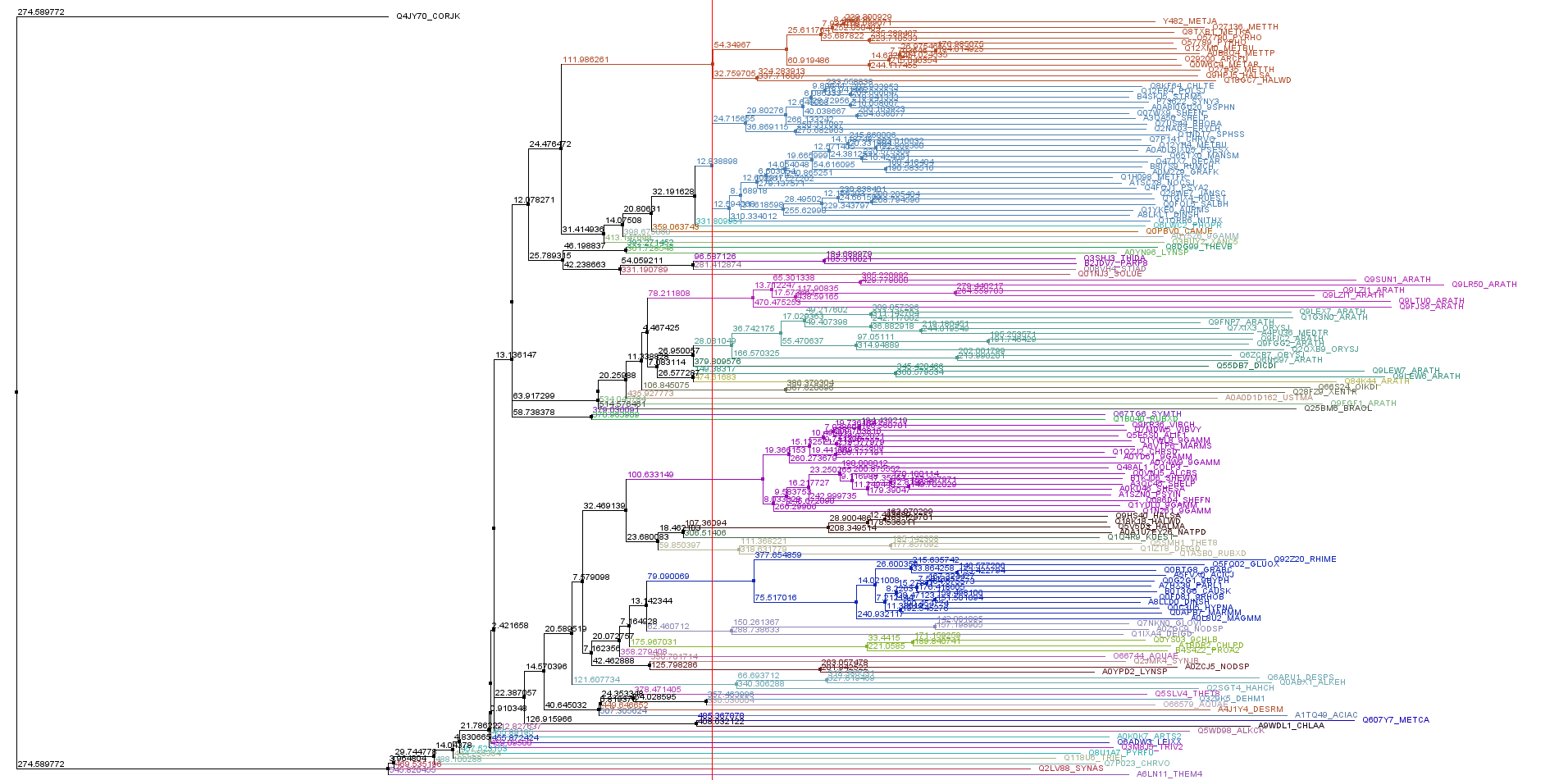
На древе синим выделена ветвь, которая мне нравится для дальнейшего анализа.
Множественное выравнивание соответствующих последовательностей, покрашенное Blosum62 с порогом Identity 90%:
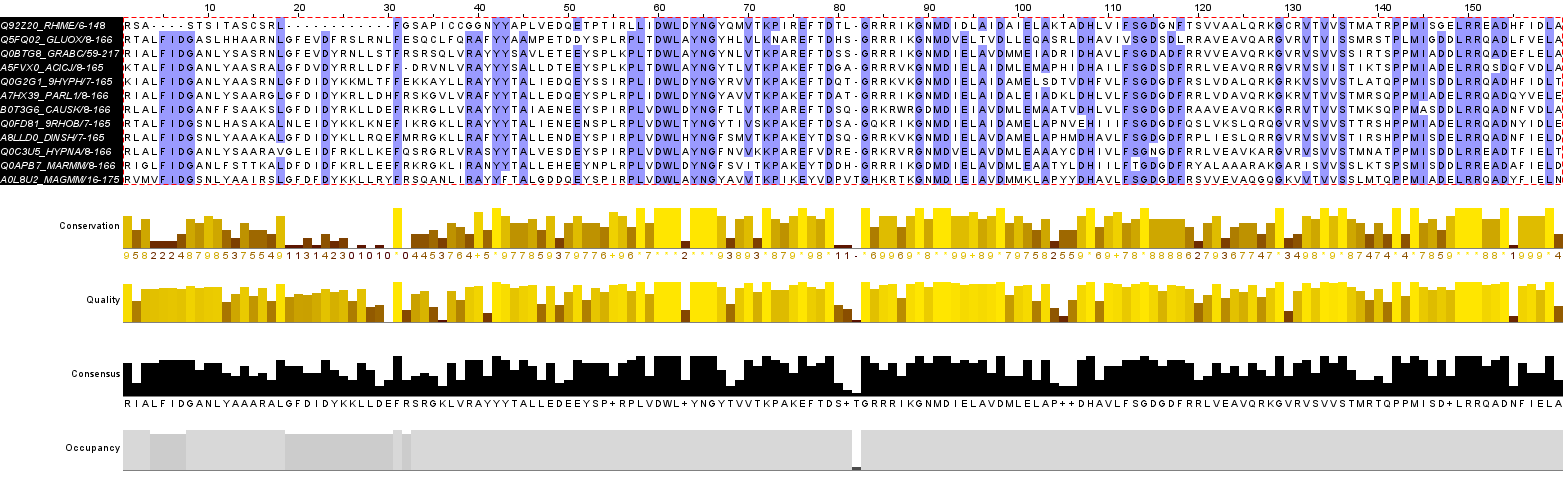
Первая последовательность сильно отличается от остальных, исключим ее из анализа. Оставшиеся ветви тоже образуют кладу.
Полученный мотив из колонок 57-66: P-L-[T,I,V]-D-W-L-[D,T,A,H]-Y-N-G
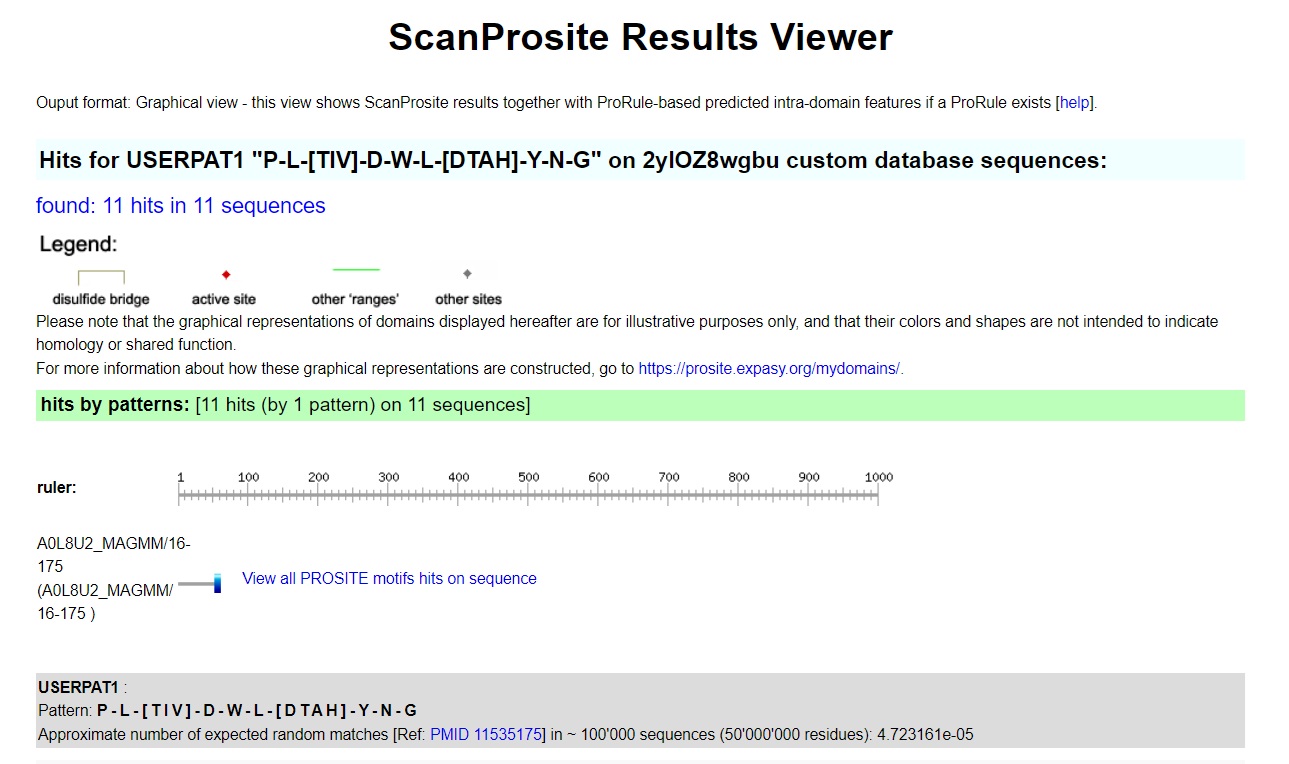
Мотив искала среди своих последовательностей с помощью PROSITE. В базе нашлось ровно 11 последовательностей, сколько и было в кладе. Значит, мотив специфичный и сильный - он хорошо разделяет нужные последовательности от остальных.
Задание 3. PSI-BLAST
Я выбрала AC P39450 - S-(hydroxymethyl)glutathione dehydrogenase, принадлежит Photobacterium damsela subsp. piscicida (Pasteurella piscicida).
Хватило всего две итерации PSI BLAST:
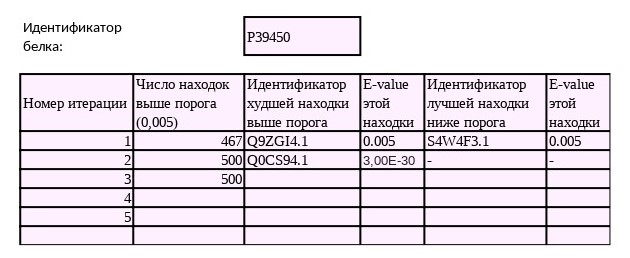
На второй итерации нижнее значение E-value получилось очень маленьким, это говорит в пользу того, что эти 500 белков прекрасно кластеризуются в семейство.