Задание 1.
Выбрала двудоменную архитектуру NYN и Xre/MbcA/ParS-like_toxin-bd (IPR024467), она включает 52 белка.

Множественное выравнивание в Jalview:
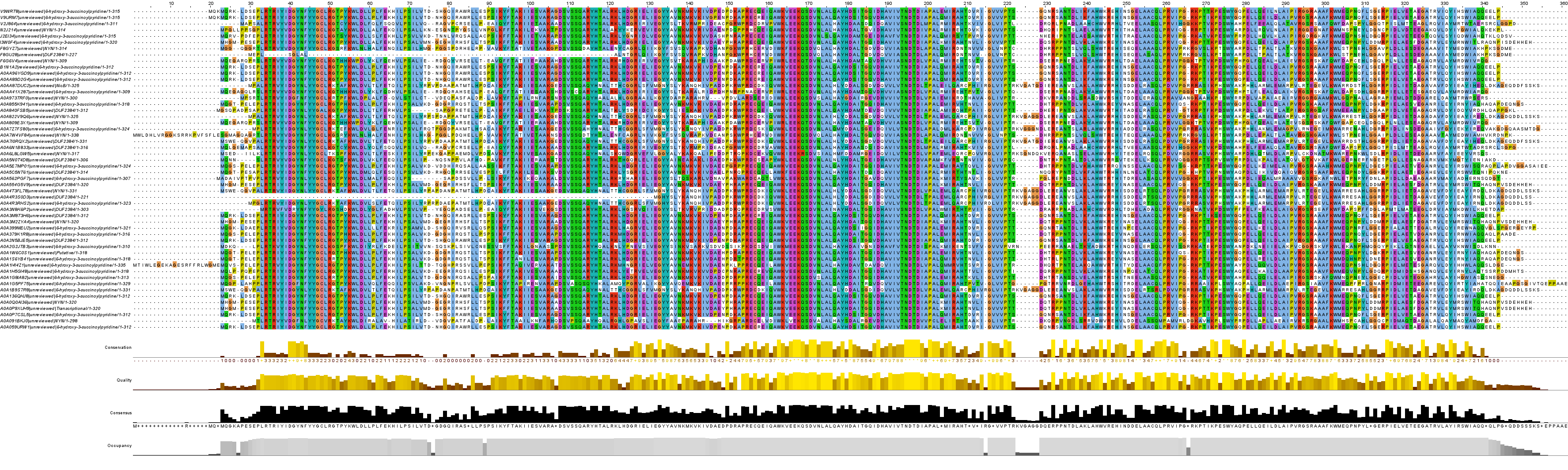
Колонки домена NYN: 33-222, в основном это 10 АК белка, встречаются 4, 6, 32, 33.
Колонки домена Xre/MbcA/ParS-like_toxin-bd: 232-333. Это у большинства 201 аминокислота, но есть разброс от 188 до 224.
После создания и ревизии выравнивания исключительно доменов я оставила 27 последовательностей.
В качестве референсной я оставила последовательность >A0A1S6YB41|unreviewed|6-hydroxy-3-succinoylpyridine/10-301 hydroxylase|taxID:321
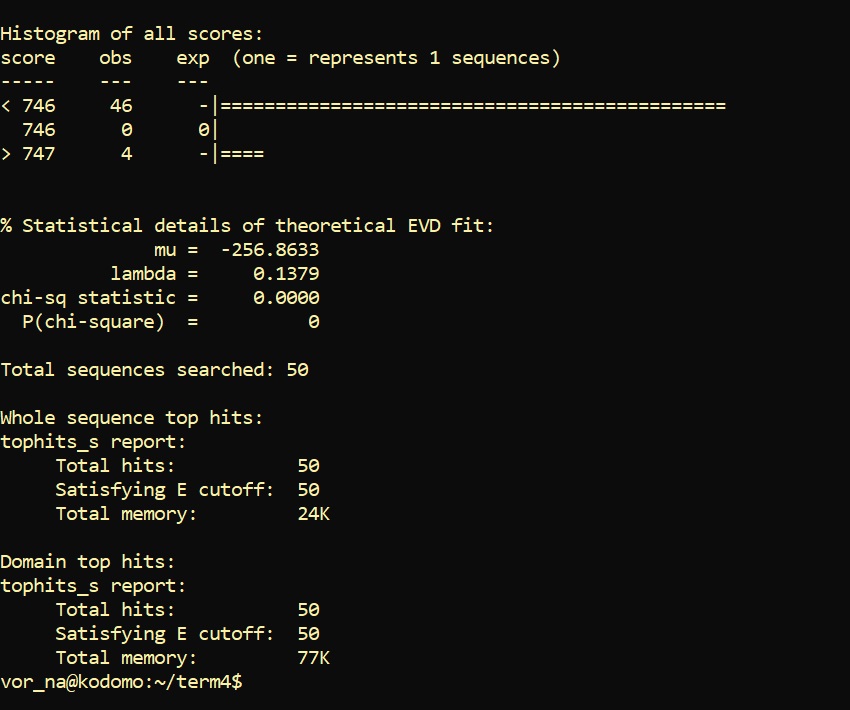
Создала HMM-profile, проверка исходной базы с 50 белками дала 50 hits с очень хорошим E-value, как и ожидалось.
Использованные команды:
hmm2build profile build_hmm.fa
hmm2calibrate profile
hmm2search nyn_50_positive.fa
Для дальнейшей оценки качества я сделала базу из моей конфигурации + двудоменной конфигурации NYN + CSP_DNA-bd, она мне понравилась количеством последовательностей и тем, что эта конфигурация напоминает нужную (длина около 300, домены расположены близко), то есть будет наиболее эффективна для обучения.
Порог по умолчанию E-Value = 10, и нашлось 291 hits.
Если посмотреть на табицу выдачи, будет четкая граница между нужными 50 и всеми остальными, там score становится сильно отрицательным и e-value переходит порог 0.005.
Построила таблицу, посчитала значения, построила ROC-кривую. Она очень выраженно приживается вверх-вправо, значит, разделение происходит четко. F1-score дает однозначное указание на порог e-Value 0.005.