Анализ трансмембранного белка с трансмембранной частью, представленной бета-листами.
По классам на сайте OPM нашла трансмембранные белки с бета-бочнонком - порины. Из них выбрала показавшееся мне интересным семейство поринов, обеспечивающих карбапенем-устойчивость. Оно называетсяImipenum resistance-associated porin (имипенум - бета-лактамный антибиотик, относящийся к классу карбапенемов.)
Буду рассматривать белок Carbapenem-associated resistance porin CarO2 - ассоциированный с устойчивостью к карбапенему порин CarO2. Этот белок локализуется в наружней мембране грам-негативных бактерий Acinetobacter baumannii.
Uniprot ID: Q5DL42_ACIBA
Primary PDB represention: 4rlb
Про функцию на uniprot не написано, но название говорит само за себя.
Трансмембранные сегменты: 1( 16- 24), 2( 31- 37), 3( 44- 50), 4( 74- 80), 5( 97- 103), 6( 149- 156), 7( 164- 171), 8( 216- 224). Одна субъединица.
Взяла последовательность с uniprot.
GFF3 выдача DeepTMHMM
Предсказанные ТМ: 42-47, 51-57, 63-70, 96-103, 116-123, 170-176, 185-190, 239-244. Их тоже восемь, частично координаты совпали, но в основном - нет.
Визуальная выдача:
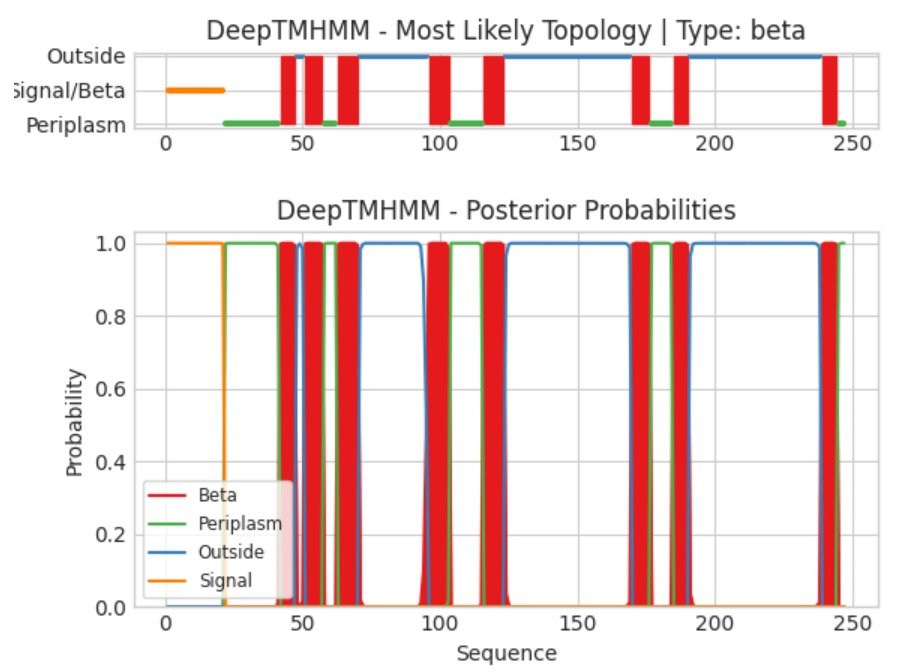
Здесь на первом изображении обозначен бислой: сверху - наружняя часть, снизу - внутренняя. Красные вертикальные полоски - прокалывающие мембрану бета-слои.
На втором изображении отложена вероятность предсказанной структуры - степень уверенности программы. Во внемембранных участках она в принципе не сомневается, но на границе вероятность падает в ноль.
Получается, в границах бета-тяжей программа не очень уверена, и это согласуется с наблюдаемыми различиями: предсказанный тяж 96-103, а по OPM - 97-103. Количество тяжей совпало, а их координаты - совсем нет, что странно, учитывая увереннность программы в немембранности этих доменов. Видимо, программа предсказывает сильно примерно расположение бета-слоев. Так как в бета-бочонке одна сторона бета-тяжа гидрофильная, а другая - гидрофобная, предсказывать общую гидрофобность участка довольно трудно.
Анализ трансмембранного белка с неразрешенной структурой и трансмембранной частью, представленной альфа-спиралями.
Мой белок - swissprot id: NHAB_HAHCH - Na(+)/H(+) antiporter NhaB, Na+/H+ антипотер, меняет внутренние ионы натрия на наружные протоны. Организм: Hahella chejuensis (strain KCTC 2396)
Запустила DeepTMHMM:
Предсказано 15 TMRs: 24-38, 42-59, 64-77, 95-106, 133-145, 147-157, 204-222, 240-262, 301-317, 322-336, 352-369, 392-401, 403-412, 442-462, 477-497.
Визуальная выдача:
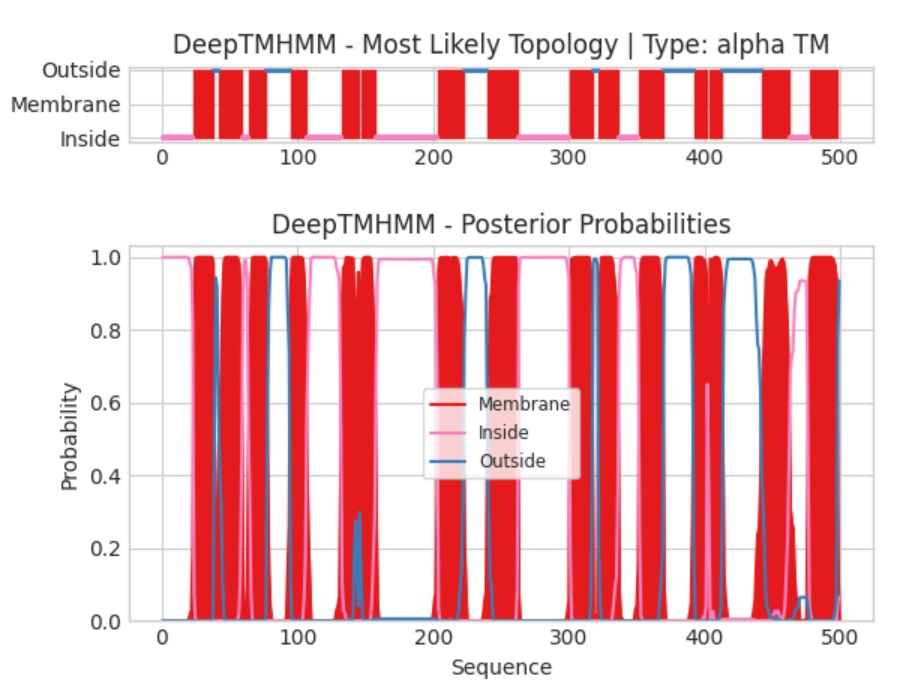
Логика изображений та же самая: на первом бислой, на втором - вероятность.
Для работы с PPM нашла гомологичный белок E. coli в OPM: вот - оттуда узнала, что он должен локализоваться во внутренней мембране грам- бактерий. N-конец находится внутри, согласно DeepTMHMM.
Transmembrane secondary structure segments - 15:
1(20 - 41), 2(42 - 61), 3(62 - 77), 4(91 - 110), 5(128 - 146), 6(147 - 154), 7(205 - 221), 8(243 - 265), 9(299 - 317), 10(322 - 337), 11(351 - 372), 12(391 - 406), 13(407- 417), 14(435 - 452), 15(476 - 497)
То есть программа обнаружила в предсказанной AlphaFold структуре все 15 трансмембранных доменов, как и предсказано выше. Координаты тоже совмещаются очень хорошо: отличаются в пределах пяти, очень мало.
AlphaFold предсказывает все альфа-спирали с высоким уровнем уверенности, это хороший материал для PPM.