G G A G A G G U G C C C G A G U G G C DHU G A A G G G A C A C G A C U G G A A A U C G U G U A G G G G G G C U U A A A C C U C C C U C G C G G G T PSU C G A A U C C C G C C C U C U C C G C C A |
find_pair -t 1SER.pdb 1ser_list
find_pair -td 1SER.pdb 1ser_list
| Спираль_№1 | Спираль_№2 | Спираль_№3 | Спираль_№4 |
# 1 | T:...4_:[..G]G-----C[..C]:..69_:T # 2 | T:...5_:[..A]A-----U[..U]:..68_:T # 3 | T:...6_:[..G]G-----C[..C]:..67_:T # 4 | T:...7_:[..G]G-----C[..C]:..66_:T # 5 | T:..49_:[..G]G-----C[..C]:..65_:T # 6 | T:..50_:[..C]C-----G[..G]:..64_:T # 7 | T:..51_:[..G]G-----C[..C]:..63_:T # 8 | T:..52_:[..G]G-----C[..C]:..62_:T # 9 | T:..53_:[..G]G-----C[..C]:..61_:T # 10 | T:..54_:[..T]T-**--A[..A]:..58_:T # 11 | T:..55_:[PSU]P-**+-G[..G]:..18_:T |
# 1 | T:..10_:[..C]C-----G[..G]:..25_:T # 2 | T:..11_:[..C]C-----G[..G]:..24_:T # 3 | T:..12_:[..C]C-----G[..G]:..23_:T # 4 | T:..13_:[..G]G-**--G[..G]:...9_:T # 5 | T:..14_:[..A]A-**--U[..U]:...8_:T # 6 | T:..15_:[..G]G-**+-C[..C]:..48_:T |
# 1 + T:..19_:[..G]G-----C[..C]:..56_:T |
# 1 | T:..45_:[..A]A-----U[..U]:..47Q:T # 2 | T:..46_:[..G]G-----C[..C]:..47P:T # 3 | T:..47_:[..G]G-----C[..C]:..47O:T # 4 | T:..47A:[..G]G-----C[..C]:..47N:T # 5 | T:..47B:[..G]G-*---U[..U]:..47M:T # 6 | T:..47C:[..G]G-----C[..C]:..47L:T # 7 | T:..47D:[..G]G-----C[..C]:..47K:T |
| Каноническая модель тРНК. |
|
|
| Предположительная, "спекулятивная" модель Ser-tRNA, представленная в файле 1SER.pdb |
restrict rna script col_helices_2.scr backbone 100 wireframe off # молекула тРНК представлена в тяжевой модели select g4.p color cyan cpk 400 # первый атом фосфора 4-ого нуклеотида (гуанин g) в шариковой модели, голубого цвета pick label set fontsize 14 set fontstroke 1 label %n %a %r # в названии указаны: имя нуклеотида (G, C, A, и тд), тип атома (фосфор), номер нуклеотида. select c71.p color magenta cpk 400 # последний атом фосфора 71-ого нуклеотида (цитидин С) в шариковой модели, розового цвета label %n %a %r # в названии указаны: имя нуклеотида (G, C, A, и тд), тип атома (фосфор), номер нуклеотида. zoom 150 |
|
|
|
|
|
|
restrict rna and not backbone zoom 180 select (oxygen,nitrogen) and rna and not backbone spacefill 200 select rna and not (oxygen, nitrogen) script col_helices_2.scr select (oxygen,nitrogen) and rna and not backbone color cpk select rna and not (backbone,oxygen,nitrogen) wireframe 60 # Итого получено изображение только нуклеотидных оснований с шариковой моделью атомов кислорода и азота, а углеродный остов этих оснований покрашен в цвета, указывающие на их принадлежность определенным спиралям или/и петельным участкам |
|
Интеркалярный стэкинг - виден сэндвич из двух зеленых (спирализованными) и между ними - белым (неспирализованным) нуклеотидами. |
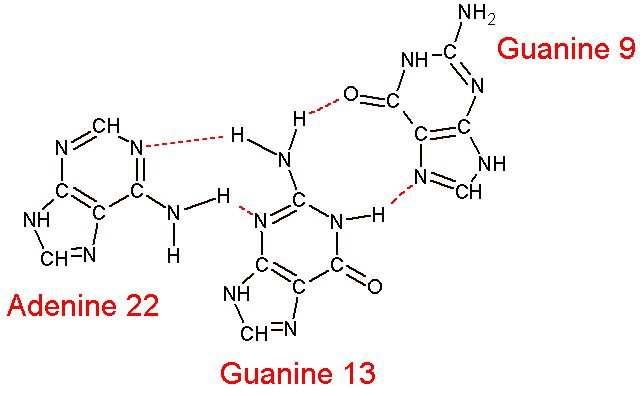
|
|
|
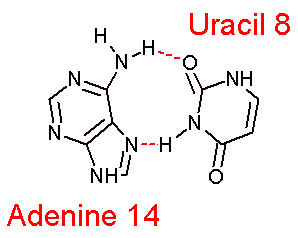
|
find_pair 1SER.pdb -t stdout | analyze
|
|
einverted -verbose
SEQUENCE: Score 22: 9/9 (100%) matches, 1 gaps
1 ggagaggtgc 10
||||||| ||
90 cctctcc-cg 82
|
mfold SEQ=1SER.fasta P=15
|
|