Бархатов В.
Ген pdxH кодирует монофункциональный белок, являющийся флавопротеином. Он может использовать в качестве субстрата пиридоксин-5'-фосфат (PNP) или пиридоксамин-5'-фосфат (PMP), поэтому его также называют PNP/PMP оксидазой. Белок играет ключевую роль в синтезе кофермента пиридоксаль-5'-фосфата, также участвует в "реутилизационном" пути. При окислении PNP и PMP используется молекулярный кислород, однако известен и другой путь при анаэробном росте ECOLI.
Реакция:
O2 + pyridoxine-5'-phosphate <=> H2O2 + pyridoxal 5'-phosphate(по данным БД EcoCyc)
Общие сведения о белке PDXH_ECOLI
Описание 3D-структуры
Описание области контакта с функциональным лигандом
Доменная структура PDXH_ECOLI
БД PROSITE, Составление паттерна
Гомологичные белки — белки, имеющие общее эволюционное происхождение.
В одно семейство гомологов обычно объединяют белки с одинаковыми функциями. Кроме того, белки из одного семейства должны быть структурно близки, иметь одинаковые домены и похожие аминокислотные последовательности (чем дальше эволюционное расстояние — тем больше они будут различаться).
Самый простой способ определить гомологичные белки — найти среди имеющих одинаковую функцию белков те, для которых можно построить хорошее множественное выравнивание. Однако для настоящих гомологов правильное выравнивание должно быть таким, чтобы функционально значимые остатки в разных последовательностях оказались в одних столбцах выравнивания, чтобы в спиралях и тяжах белка не оказалось гэпов, чтобы участки, имеющие одинаковую структуру и пространственное положение, выровнялись друг с другом, и наоборот, например, петли между спиралями не обязательно должны выровняться. Стандартные программы при построении выравнивания ищут максимальное число совпадающих/похожих аминокислот, штраф за гэп не зависит от его положения, пространственная структура не учитывается, в результате полученное выравнивание оказывается далеко не идеальным.
Для улучшения полученного выравнивания можно отредактировать его вручную, основываясь на пространственной структуре одного из гомологов и на пространственном выравнивании нескольких из них.
Примеры.
1. Это — выравнивание последовательностей гомеодоменов, построенное программой ClustalW: ![]()
На картинке изображено пространственное выравнивание пяти из этих последовательностей.
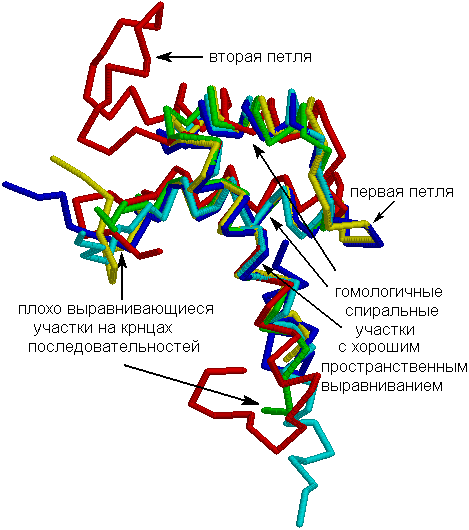
Как видим, они имеют три хорошо совпадающих α-спирали, между которых у некоторых последовательностей располагаются петли. Отредактируем выравнивание, полученное в ClustalW, в соответствии с нашими наблюдениями (в поле number указана нумерация по PDB-структуре; закрашены хорошо выравнивающиеся в пространстве участки): (выравнивание в формате msf) ![]()
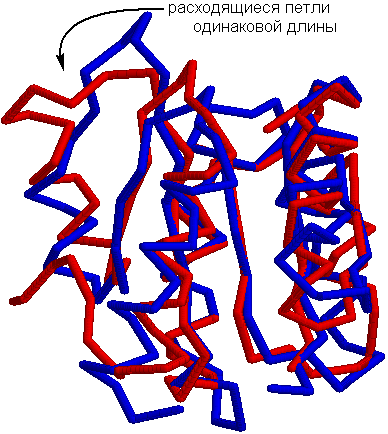
2. В пространственном выравнивании CheY-like доменов из E.coli и T.maritima петля между 79 и 85 остатками занимает разное положение, но имеет одинаковую длину. Это позволяет предположить, что петли гомологичны, но при проведении РСА находились в разных конформациях. Однако возможен и другой вариант: когда-то эти участки имели одинаковую структуру, но после замены в процессе эволюции одной или нескольких аминокислот структура изменилась, не повлияв на функцию белка.
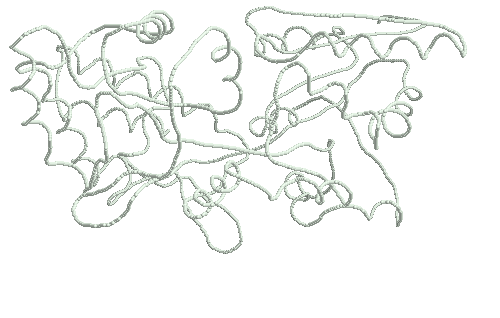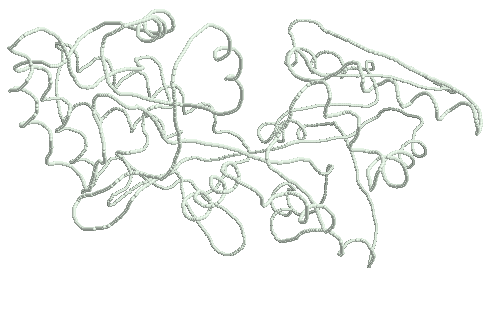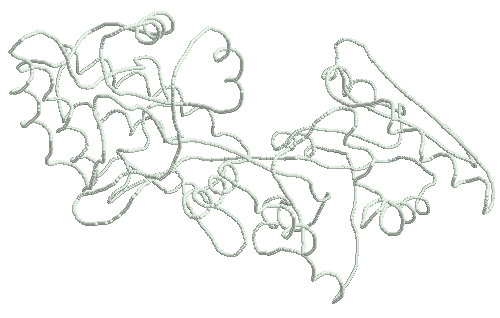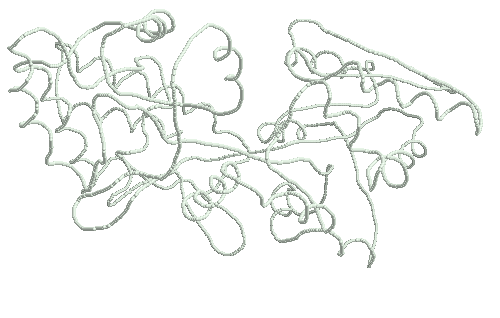
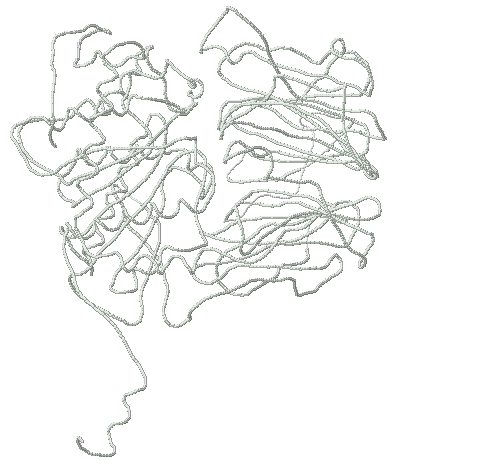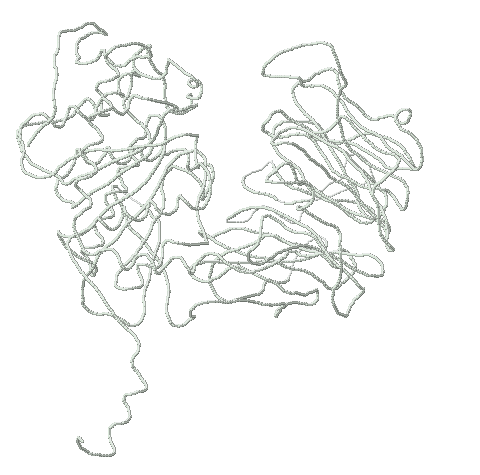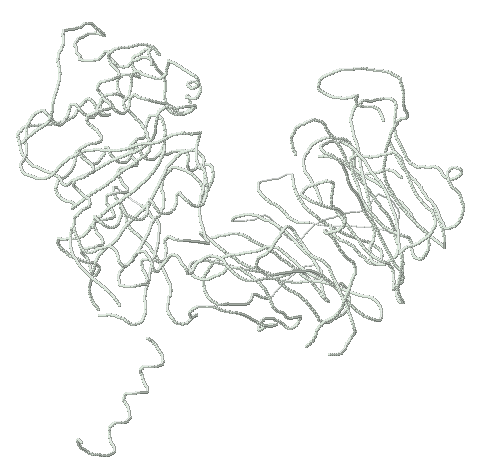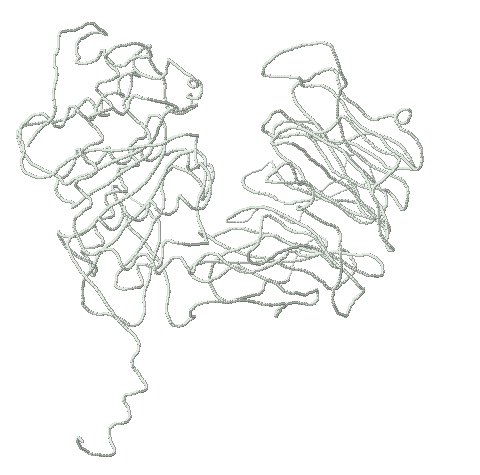
3. Существуют белки, существенно меняющие свою конформацию. Такие белки собраны в базе данных MolMov. Например, на картинках, приведённых ниже, глобулярные домены шарнирно движутся друг относительно друга.
Мальтодекстрин-связывающий белок (коды сривниваемых PDB-структур 1jw5 и 1anf, максимальное отклонение по Cα-атому 1.16208):
Пролилолигопептидаза (1yr2 и 2bkl, максимальное отклонение по Cα-атому 7.03443):
Изучаемое семейство состоит из белков, содержащих домен Pyridox_oxidase (PF01243). Функция домена - связывание FMN ( GO:0010181 ). Пиридоксамин-5'-фосфатоксидаза (PNPOx) — это FMN флавопротеин, он катализирует реакцию окисления пиридоксин-5'-фосфата (PNP) или пиридоксамин-5'-фосфата (PMP) до пиридоксаль-5'-фосфата (PLP). Эта реакция — последний этап биосинтеза PLP у E.coli и часть "реутилизационного" пути этого кофермента у E.coli и млекопитающих. Сайты связывания FNM и субстрата очень консервативны на протяжении эволюции. FMN-связывающий домен представлен у пиридоксамин-5'-фосфатоксидаз и некоторых белков, не проявляющих ферментативной активности. поэтому его также называют PNP/PMP оксидазой. Белок играет ключевую роль в синтезе кофермента пиридоксаль-5'-фосфата, также участвует в "реутилизационном" пути.
В банке Pfam к этому семейству отнесено 935 последовательностей. Белки семейства встречаются у: эукариот — 91, бактерий — 756, архей — 51. Примечательно, что у бактерий известно больше всего белков этого типа.
По данным Pfam, белки бывают 16 различных доменных архитектур, при этом 856 белков (из 935) имеют архитектуру только с Pyridox_oxidase доменом.
Взято выравнивание из 147 фрагментов последовательностей ("просеянные" (seed) последовательности, содержащие данный домен, из Pfam), оставлена 31 (отбирались наиболее отличающиеся последовательности, из близких видов по возможности отбрасывались), для удобства приведено выравнивание из 11 последовательностей (выравнивание в формате msf) (посмотреть все 31) ![]()
Для исследования составлена выборка этих же последовательностей целиком из SwissProt (запрос: Y2074_MYCTU| Q9ADN2_STRCO| Q89MF3_BRAJA| O07754_MYCTU| Q9L4E6_BACFR| Q8GJ54_BACVU| Q97FS1_CLOAB| Q8PA06_XANCP| PDXH_DEIRA| PDXH_HAEIN| PDXH1_RALSO| PDXH_CAUCR| PDXH_RHIME| PDXH_MYXXA| PDXH_XANCP| PDXH_CAEEL| PDXH_SHIFL| O69755_PSEAE| Q8ELS6_OCEIH| Q8PSH5_METMA| Q8G4E3_BIFLO| Q06608_YEAST| Q93ST0_PLESH| Q88N58_PSEPK| Q890I2_LACPL| Q89FM0_BRAJA| Q92XF2_RHIME| Q89CG2_BRAJA| Q88NP4_PSEPK| Q8PZT4_METMA| Q88SE1_LACPL , фрагментов найдено не было, из 31 запрошенной нашлось только 10 последовательностей, которые присутствуют в SwissProt)
Затем в выборку были добавлены ещё 5 последовательностей (полученных по ссылке на описание PDB-структуры в Pfam), для которых известны PDB:
pdb id 1cio PDX3_YEST (P38075) 1flm FMNB_DESVM (Q46604) 1nrg PNPO_HUMAN (Q9NVS9) 1rfe O5240_MUCTU 1tgm O69775_PSEAE
Множественное выравнивание, полученное в ClustalW: (выравнивание в формате msf) ![]()
Последовательности на концах не обязательно должны выравниваться, поэтому убираем длинные гэпы в начале. Некоторые последовательности сильно сдвинуты по сравнению с выравниванием из Pfam. Поэтому ниже приведено корректированное выравнивание (в строке 2D_struct α-спирали обозначены как "H", β-тяжи — как "S" (использовалась структура PDX3_YEST); в строке Aligment — стоит A, если есть сопоставление остатков всех последовательностей (такие участки могут содержать несколько гэпов), L, когда есть сопоставление участков всех последовательностей, но внутри них нет выравнивания (связывающие участки); в строке Function звёздочкой отмечены функционально важные остатки; "D>>>" и "<<<D" — начало и конец домена): (выравнивание в формате msf) ![]()
По полученному выравниванию был составлен паттерн, учитывались значимые и хорошо выровненные аминокислоты:
[RVT]-X-[VLITM]-[LIVWG]-X(0,3)-[LIFY]-[KART]-X(3,6)-[GI]-X(2)-[FLVIW]-[YVLIAF]-[TSVE]-[NPTH]-X(2,3)-[SM]-[QRAH]-K
По паттерну производился поиск в PROSITE ( ScanProsite).
Найдено 85 последовательностей, из них 6 оксоацилредуктаз, остальные -- FMN-связывающие или пиридоксамин-5'-фосфатоксидазы (т. е. найдено 79 из 123 аннотированных в SwissProt последовательностей семейства и 6 лишних), при этом не нашлось два белка из выборки (O5240_MUCTU и O69775_PSEAE), так как они отсутствуют в БД SwissProt.
Использовался профиль PFTOOLS, построенный программой MyHits Полученный профиль: pf01243.prf По нему найдено 136 последовательностей, две из выборки не найдены (O5240_MUCTU и O69775_PSEAE)
По результату программы SVETKA семейства делились не по таксономическому признаку.
Выдача SVETKA:
+-87-+-71-*O69775_PSEAE
| |
| +-46-+-61-*PDX3_YEST
| |
| +-16-+-51-*PDXH_SHIFL_
| |
| +-6--+-8--+-70-*PDXH_XANCP_
| | |
| | +-10-+-34-*PDXH_MYXXA_
| | |
| | +-17-+-31-*PDXH_CAUCR_
| | |
| | +-35-*PDXH_RHIME_
| |
| +-4--+-64-*PDXH_CAEEL_
| |
| +-4--+-47-*PDXH_HAEIN_
| |
| +-6--+-48-*PNPO_HUMAN
| |
| +-11-+-48-*PDXH_DEIRA_
| |
| +-26-*PDXH1_RALSO_
|
+-87-+-119*FMNB_DESVM
|
+-75-+-103*Y2074_MYCTU_
|
+-105*O5240_MUCTU
| Таксономическое дерево:
cellular organisms + Eukaryota + + Fungi/Metazoa group + + + Metazoa + + + + Bilateria + + + + + Pseudocoelomata + + + + + + Caenorhabditis elegans + + + + + Coelomata + + + + + + Homo sapiens + + + Fungi + + + + Saccharomyces cerevisiae + Bacteria + + Proteobacteria + + + Alphaproteobacteria + + + + Caulobacterales + + + + + Caulobacter vibrioides + + + + Rhizobiales + + + + + Sinorhizobium meliloti + + + delta/epsilon subdivisions + + + + Deltaproteobacteria + + + + + Desulfovibrionales + + + + + + Desulfovibrio vulgaris + + + + + Myxococcales + + + + + + Myxococcus xanthus + + + Gammaproteobacteria + + + + Pasteurellales + + + + + Haemophilus influenzae + + + + Pseudomonadales + + + + + Pseudomonas aeruginosa + + + + Enterobacteriales + + + + + Shigella flexneri + + + + Xanthomonadales + + + + + Xanthomonas campestris + + + Betaproteobacteria + + + + Ralstonia solanacearum + + Deinococcus-Thermus + + + Deinococcus radiodurans + + Actinobacteria + + + Mycobacterium tuberculosis |
Первое подсемейство:
Второе подсемейство:
Поиск диагностических признаков, программа SDPpred: предсказано 17 SDP.
![]()
Предсказанные различия между группами:
| Позиция по выравниванию | 89 | 103 | 105 | 108 | 126 | 147 | 176 | 181 | 184 | 186 | 187 | 188 | 236 | 240 | 241 | 248 | 250 |
| АК в первой группе | LTV | H | TV | GW | TP | QEM | DLG | YVG | RWA | R | TY | PTD | IA | IRF | ESK | DGV | LPT |
| АК во второй группе | A | SNDY | R | LV | NH | FL | YQ | PA | ST | LIVP | AGM | AS | VPL | E | F | R | H |
| Значимость АК в этой позиции | + | + | ++ | ++ | + | + | + | + | + | + | + | + | + | + | + | +++ | + |
Kalinina OV, Mironov AA, Gelfand MS, Rakhmaninova AB. (2004) Automated selection of positions determining functional specificity of proteins by comparative analysis of orthologous groups in protein families. Protein Sci 13(2): 443-56.