Задача — построить и исследовать филогенетическое дерево белков.
Аминокислотные последовательности были найдены с помощью SRS в БД UniProt, если попадались последователиности из двух подвидов одного вида, то выбирался один (для первой группы). Искались белки:
Ниже приведены последовательности этих белков: ![]()
|
Список организмов, изкоторых эти последовательности получены: |
Таксономическое дерево (получено со страницы таксономии сайта NCBI): |
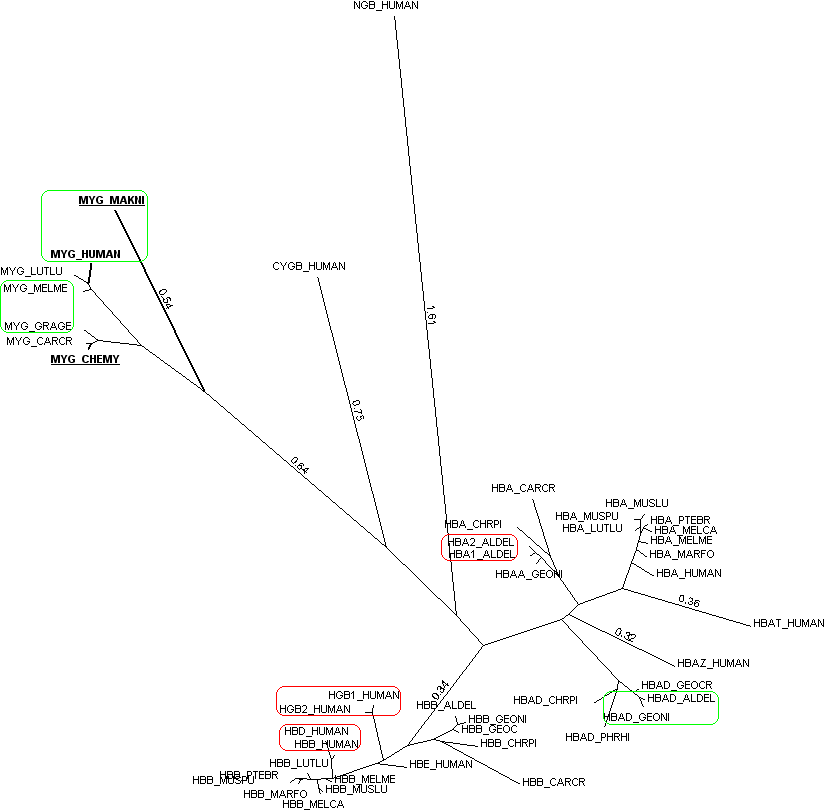
С помощью emma было построено выравнивание, а затем программой eprotdist получена матрица расстояний и реконструировано филогенетическое дерево по алгоритму Neighbour-Joining с помощью программы eneighbor. Изображение дерева получено в GeneMaster. Последовательности из внешней группы выделены жирным шрифтом. Ортологи отмечены зелёным, паралоги красным.