ФББ МГУ, 2 курс, весна 2006
ecoli02.txt – фрагмент последовательности ДНК Escherichia coli в формате EMBL (с аннотацией);
human02.txt – фрагмент последовательности ДНК человека в формате FASTA.
Мне дан фрагмент последовательности ДНК Escherichia coli и аннотация к нему в формате EMBL (см. файл ecoli02.txt). Надо идентифицировать гены в этом фрагменте при помощи программ ORF Finder и GeneMark и сравнить полученные результаты с аннотацией.
| CDS из аннотации ecoli02 | |||
| начало | конец | длина | рамка |
| <1* | 250 | ||
| 247 | 969 | 723 | +1 |
* CDS продолжается за пределами аннотированной последовательности
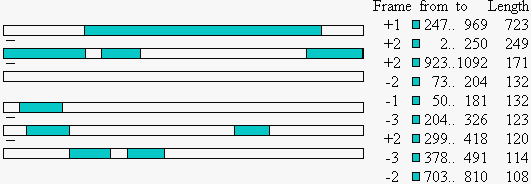
Программа ORF Finder (Open Reading Frame Finder) идентифицирует открытые рамки (ORFs) на обеих цепях ДНК. Доступ к ней можно получить по ссылке
| Обнаруженные открытые рамки: |
 |
Самые длинные не пересекающиеся предсказанные рамки приведены в таблице; рамки, совпадающие с аннотированными CDS, выделены зелёным (как видно, они обе совпадают с аннотацией):
| начало | конец | длина | рамка |
| 247 | 969 | 723 | +1 |
| *2 | 250 | 249 | *+2 |
С помощью BLAST был проведён поиск белков, похожих на транслированную ORF, в GenBank. Нашлись гомологи только для трёх открытых рамок, причём для двух рамок, совпадающих с аннотированными, есть гомологи со 100% сходством. Вот их выравнивания: ![]()
GeneMark – программа статистического предсказания генов. Доступ к ней можно получить по ссылке
Результаты работы программы GeneMark 2.4:
| Ген | Цепь | Левый Конец | Правый Конец | Длина гена | Класс |
| 1 | + | <2 | 250 | 249 | 1 |
| 2 | + | 247 | 969 | 723 | 1 |
Графики распределения кодирующего потенциала для каждой рамки считывания: ![]()
Дан фрагмент ДНК человека, содержащий альтернативно сплайсируемый ген (файл human2.txt). Задача – найти две различные изоформы этого гена и некодирующие экзоны используя программы GENSCAN, BlastX и Human Genome Browser (HGB)
GENSCAN – программа статистического распознавания генов, она предсказывает границы экзонов и интронов, промоторы, сайты полиаденилирования. Доступ к программе можно получить по ссылке
Таблица выдачи Genscan:
| Начало | Конец | Длина | Тип | Цепь | Рамка |
| 613 | 1118 | 506 | начальный экзон | + | 0 |
| 1223 | 1427 | 205 | внутренний экзон | + | 2 |
| 1517 | 1711 | 195 | внутренний экзон | + | 1 |
| 1945 | 1977 | 33 | внутренний экзон | + | 0 |
| 2173 | 2308 | 136 | внутренний экзон | + | 0 |
| 2462 | 2531 | 70 | внутренний экзон | + | 0 |
| 3225 | 3320 | 96 | внутренний экзон | + | 0 |
| 3426 | 3591 | 166 | конечный экзон | + | 0 |
| 3762 | 3767 | 6 | сайт полиаденирования |
Программа BlastX производит поиск формального транслята входной последовательности в базе данных известных белков. Доступ к программе можно получить по ссылке
Поиск проводился без фильтра фрагментов малой сложности и только среди белков позвоночных. Результат поиска: ![]()
Выравнивания двух изоформ с разным числом экзонов:
Сравнение двух изоформ с разным числом экзонов.
Альтернативные экзоны выделены цветом: оранжевым – имеющие сильно различающиеся границы, красным – отсутствующие в другой изоформе:
| Человек Сериновая протеаза Htra2 | Рыба-зебра Предположительно сериновая протеаза 11 | ||||
| рамка | координаты по ДНК | координаты по белку | рамка | координаты по ДНК | координаты по белку |
| +1 | 613-1119 | 1-169 | +1 | 1030-1110 | 161-187 |
| +3 | 1221-1427 | 169-237 | +3 | 1206-1418 | 185-255 |
| +2 | 1517-1711 | 237-302 | +2 | 1505-1711 | 255-323 |
| +1 | 2203-2307 | 314-348 | +1 | 2203-2289 | 335-363 |
| +3 | 2464-2529 | 349-371 | |||
| +1 | 3223-3588 | 372-458 | |||
Сравнение найденных BlastX экзонов с предсказанием GENSCAN. Красным выделены строки, соответствующие лишним GENSCAN-экзонам, не пересекающимся ни с одним BlastX-экзоном. BlastX-экзонов, не пересекающихся с GENSCAN-экзонами, не найдено. Перекрытие QQ - мера близости GENSCAN и BlastX-экзонов, вычисляется как отношение длины пересечения к длине объединения отрезков; GENSCAN-экзоны, хорошо совпадающие с каким-либо BlastX-экзоном (QQ>0.9), выделены зелёным цветом:
| Экзоны по GENSCAN | BlastX Человек | BlastX Рыба-зебра | ||||
| Координаты по ДНК | Рамка | Координаты по ДНК | Рамка | Координаты по ДНК | Рамка | |
| 613-1118 | 0 | 613-1119 | +1 | 1030-1110 | +1 | 1 |
| 1223-1427 | 2 | 1221-1427 | +3 | 1206-1418 | +3 | 0,99 |
| 1517-1711 | 1 | 1517-1711 | +2 | 1505-1711 | +2 | 1 |
| 1945-1977 | 0 | 0 | ||||
| 2173-2308 | 0 | 2203-2307 | +1 | 2203-2289 | +1 | 0,77 |
| 2462-2531 | 0 | 2464-2529 | +3 | 0,94 | ||
| 3225-3320 | 0 | 3223-3588 | +1 | 0,26 | ||
| 3426-3591 | 0 | 0 | ||||
База Human Genome Browser (HGB) содержит гены, белки, мРНК, EST и другие объекты, картированные на геном человека (и не только). Браузер позволяет просмотреть разнообразную информацию, относящуюся к заданному фрагменту ДНК. Программа BLAT аналогично BLAST позволяет искать последовательности в геноме с учетом возможной фрагментированности генома.
В программе Human BLAT Search был проведён поиск похожих фрагментов генома для последовательности человеческой ДНК.
Результаты для лучшего (совпадающего на 100%) из семи найденных фрагментов:
| Цепь ДНК | Геномные координаты | Хромосома | Координаты по последовательности | Offset |
| Прямая (+) | 74668187 - 74672461 | 2 | 11 - 4285 | 74668176 |
Изображение выравнивания последовательности с геномной ДНК и сплайсированными EST и мРНК из базы: ![]()
Информация о mRNA AF141305:
| Участки нетранслируемых экзонов: |
| 11 - 612 |
| 3592 - 3784 |
| Внутренний не предсказанный BlastX кодирующий экзон: |
| 1945 - 1977 |
Все найденные в Human Genome Browser экзоны:
| МРНК AF141305 | ||
| начало экзона | конец экзона | тип экзона |
| 11 | 1118 | частично кодирующий |
| 1223 | 1427 | кодирующий |
| 1517 | 1711 | кодирующий |
| 1945 | 1977 | кодирующий |
| 2203 | 2308 | кодирующий |
| 2462 | 2531 | кодирующий |
| 3225 | 3320 | кодирующий |
| 3426 | 3784 | частично кодирующий |
Нетранслируемые участки экзонов выделены красным, транслируемые – синим: ![]()