Автор: Струкова Вера, студентка 1 курса Факультета биоинженерии и биоинформатики МГУ. им Ломоносова
РЕЗЮМЕ
Ключевые слова: бактерия, геном, протеом, Chlorobaculum tepidum TLS, ген, белок.
1. ВВЕДЕНИЕ
Chlorobaculum tepidum TLS – зелёная серная бактерия, входящая в семейство грамотрицательных Chlorobiaceae.[1] Впервые была получена из горячего источника в р. Роторуа, Новая Зеландия, предпочитает бескислородную среду с оптимальным количеством света, например, глубокие озёра, глубоководные источники термальных вод и т.д.[1] Эта бактерия является фотолитотрофом, окисляет сульфид до элементарной серы или элементарную серу и тиосульфат до сульфата.[1]Хорошо культивируется, является модельным организмом сем. Chlorobiaceae для изучения фототрофного окисления серы.[1] Геном Chlorobaculum tepidum представлен 2 154 946 парой нуклеотидов, заключённых в единственную хромосому.[2] В данной работе производился анализ генома и протеома бактерии Chlorobaculum tepidum.
2.МАТЕРИАЛЫ И МЕТОДЫ
Последовательность ДНК бактерии была получена из открытой базы данных NCBI [3], скопирована в файл и перенесена на kodomo[4]. При работе использовались различные методы работы с электронными таблицами (Google Sheets), такие как:
1.Импортирование данных;
2.Фильтр столбцов;
3.Сортировка столбцов;
4.Создание новых листов;
5.Использование функции ВПР для переноса данных между листами;
6.Создание плоских таблиц;
7.Копирование и вставка;
8.Использование значка $ для распространения формул;
9.Оформление таблицы;
10.Уничтожение формул;
11.Вставка примечаний;
12.Функции СЧЁТЕСЛИ и СЧЁТЕСЛИМН, которые позволяют подсчитать количество значений столбца данных, соответствующих одному или нескольким критериям;
13.Функция ОКРУГЛ для округления значений;
14.Создание сводных таблиц;
15.Построение гистограмм.
Также были применены программы Linux и методы работы с ним:
1.wordcount -wordsize 1 - подсчет количества нуклеотидов;
2.geecee - подсчет GC - пар;
3.wordcount -wordsize n - вычисление количества k - меров определённой длины n;
4.cbcalc -s words-n -K - расчёт O/E (отношение наблюдаемой частоты последовательности длины n к ожидаемой);
5.cusp - вывод частоты использования кодонов, кодирующих одну и ту же аминокислоту;
6.Работы с файлами с помощью программы WinSCP (загрузка в credits на kodomo).
3.РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЯ
3.1 Размер генома Chlorobaculum tepidum TLS
Размер генома C. tepidum составляет 2154946 п.н.[2] и представлен одной хромосомой, что указано на листе genome_size сопроводительных материалов. В целом, это довольно маленький геном, который, однако, не выходит за пределы известных нам геномов[5].
3.2 Типы генов
В таблице 1 представлены типы генов, встречающиеся в геноме бактерии (лист genes_per_types сопроводительных материалов). Материалы были взяты из таблицы genes.
По результатам исследований можно заключить, что геном бактерии в большей степени представлен генами, кодирующими белки (2079). На втором месте по количеству находятся псевдогены (68). Псевдоген - “нефункционирующий гомолог работающего гена, потерявший способность кодировать белок или функциональную мРНК”[6]. Псевдогены могут служить значимым источником генетической изменчивости, поэтому имеют важное значение для жизненного цикла бактерии [6].
Кроме того, в составе генома C. tepidum встречаются гены, кодирующие транспортные РНК (тРНК), рибосомальные РНК (рРНК), транспортно - матричные РНК (тмРНК), сигнальные РНК и РНКазу P.
| Таблица 1:Типы генов | |||||
| Тип генов | Количество | ||||
|---|---|---|---|---|---|
| кодирующие белки | 2079 | ||||
| псевдогены | 68 | ||||
| тРНК | 50 | ||||
| рРНК | 6 | ||||
| тмРНК | 1 | ||||
| Сигнальная РНК | 1 | ||||
| РНКаза Р | 1 | ||||
3.3 Количество и соотношение различных нуклеотидов
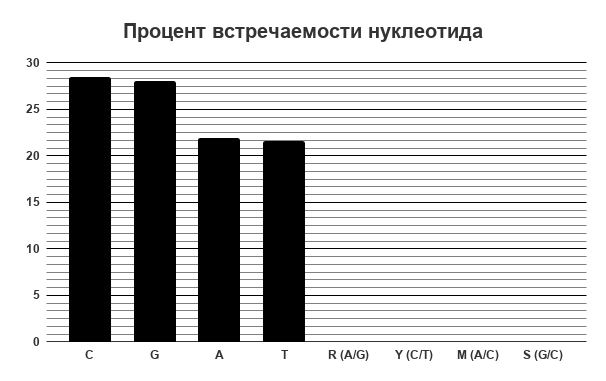
С помощью программы Linux wordcount -wordsize 1 было подсчитано количество аминокислот, результаты подсчетов представлены на рисунке 1. Дополнительно их можно посмотреть на листе nucleotide_composition. Нетрудно заметить, что в геноме помимо типичных A, T, G, C встречаются ещё буквы S, Y, M, R. Это специальные обозначения для нуклеотидов, которые не получилось определить при секвенировании, по коду IUPAC R - это A или G, S - G или C, M - A или C, Y - C или T [7.] В дальнейшем эти неопределенные нуклеотиды исключены из исследования в целях получения более простых и точных результатов. Данная гистограмма показывает примерно равное количественное соотношение G и C, A и T. Их стандартные отклонения (5417 и 4470) довольно маленькие, что говорит о выполнении правила Чаргаффа.

3.4 Соотношение GC/AT
Обратимся к листу frequency сопроводительных материалов. При помощи программы geecee было рассчитано количественное соотношение GC - пар (0,57 или 57%) к AT - парам (0,43 или 43%). Как мы видим, GC - пар больше, чем AT - пар. GC - состав является важной характеристикой бактерий. Поскольку в таких парах образуется не две, а три водородные связи между нуклеотидами, они гораздо прочнее, чем AT. Далеко не всегда количество GC можно соотнести с местом обитания бактерии[5]. Как было сказано выше, С. tepidum часто проживает в горячих источниках, поэтому возникает необходимость укрепления молекулы ДНК. Возможно, относительно большое количество GC - пар способствует такой прочности.
| Таблица 2:Количество и частоты кодонов, кодирующих аминокислоты | |||||
| Аминокислоты | Количество кодонов | Мин | Макс | ||
|---|---|---|---|---|---|
| A | 4 | 12,087 | 37,846 | ||
| C | 2 | 2,048 | 9,233 | ||
| D | 2 | 20,918 | 30,973 | ||
| E | 2 | 29,245 | 37,789 | ||
| F | 2 | 12,832 | 29,926 | ||
| G | 3 | 9,258 | 43,659 | ||
| Итого | 15 | 2,048 | 43,659 | ||
3.5 Частоты использования кодонов, кодирующих одни аминокислоты.
На листе codon_usage_table_test и codon_usage_table сопроводительных материалов представлены, соответственно, количество и частота кодонов, кодирующих аминокислоты в геноме бактерии, и сводная таблица с данными наиболее и наименее часто используемых кодонов. Сводная таблица приведена ниже в качестве таблицы 2.
Можно заметить, что наибольшее количество кодонов имеет A - аланин, что соответствует таблице генетического кода, знакомой всем нам[8]. Стоить отметить, что в таблице 2 используется однобуквенный код аминокислот, где A - аланин, C - цистеин и т.д. Однобуквенный код следует смотреть так же по ссылке[8]. В случае аланина наименее часто используемый кодон - GCT, самый часто используемый - GCC.
После аланина по количеству используемых кодонов для кодирования одной аминокислоты идёт G - глицин. Это три кодона. А вот аминокислоты C (цистеин), D (аспарагиновая кислота), E (глутаминовая кислота) и F (фенилаланин) зашифрованы в геноме только с помощью двух кодонов. В целом, аминокислотный код данной бактерии ничем не отличается от такового у подавляющего большинства других живых организмов, что в который раз подтверждает его универсальность[9].
В геноме довольно много кодонов, соответствующих отрицательно заряженным глутаминовой и аспарагиновой кислотам, глицину. Наибольшее количество составляют аланиновые триплеты (58562). Аланин является сильно гидрофобной кислотой, его боковая цепь насыщена углеводородными группами[10] Глутаминовая и аспарагиновая кислоты в растворах имеют отрицательный заряд[10].
Можно предположить, что такой состав аминокислот связан с окислительной способностью C. tepidum, участвующей в реакциях соединений серы.
3.6 Гистограмма длин белков
На рисунке 2 представлена гистограмма длин белков. Её создание можно посмотреть в сопроводительных материалах (лист proteins).
Протеом бактерии представлен в основном небольшими белками (100-300 аминокислот). Длина лежит в пределах 32 - 1534 аминокислот. Всего лишь три белка имеют длину более 1500 аминокислотных остатков. Самый длинный белок (1534) - это большая субъединица глутаматсинтазы, самый короткий (32) - Белок, содержащий сортирующий домен PEP-CTERM. Все данные были взяты из таблицы genes сопроводительных материалов.
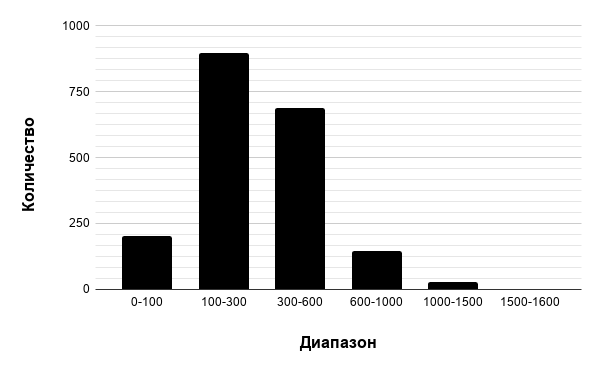
По горизонтальной оси указаны диапазоны длины белков, по вертикальной - количество белков, подходящих под данный диапазон.
4. ВЫВОДЫ
На рисунке 2 представлена гистограмма длин белков. Её создание можно посмотреть в сопроводительных материалах (лист proteins).
В данном мини - обзоре был определён размер генома, который укладывается в стандартные рамки размеров геномов бактерий. Подсчитаны типы генов, кодирующих разные белки, количество и соотношение нуклеотидов, которое соответствует правилу Чаргаффа. Соотношение GC/AT, полученное в исследовании, соотносится с местом обитания C. tepidum. Частоты кодонов, кодирующих разные аминокислоты, соответствует сложившимся представлениям об аминокислотном коде. Было уточнено возможное значение аминокислот в жизнедеятельности бактерии. Гистограмма длин белков показала, что в протеоме C.terpidum больше всего относительно коротких белков.
СОПРОВОДИТЕЛЬНЫЕ МАТЕРИАЛЫ
Хромосомная таблица с материалами исследования Strukova_supple_fin:
БЛАГОДАРНОСТИ
Выражаю благодарность преподавателям практической биоинформатики, которые помогали мне писать этот мини - обзор. А также своей семье и друзьям, поддерживающим меня в любых начинаниях.
СПИСОК ЛИТЕРАТУРЫ