Обзор генома и протеома бактерии Legionella waltersii
Резюме
В данной работе рассматривается распределение длин белков Legionella waltersii, вероятность случайного распределения генов в геноме данного организма, особенности генома, связанные с нуклеотидным составом, а также расположение рибосомальных РНК и белков.
1. Введение
Legionella waltersii – вид грамотрицательных бактерий из семейства Legionellaceae, который впервые был обнаружен в системе распределения питьевой воды в городе Аделаида в Австралии [1]. Данный род бактерий получил своё название после вспышки на то время неизвестной болезни среди людей, посетивших съезд Американского легиона – ассоциации ветеранов вооружённых сил США. Оказалось, что болезнь была вызвана бактерией, живущей в вентиляции отеля, куда поселили участников съезда [2].
Описан один случай легионеллёза, вызванного Legionella waltersii. При этом у пациента, кем являлась девочка 5 лет, отмечалась повышенная температура, лихорадка, затруднённое и учащённое дыхание, сухой кашель [3]. До этого случая считалось, что Legionella waltersii не является патогенной.
В данной работе с помощью метода электронных таблиц исследуется геном и протеом штамма NCTC13017 Legionella waltersii.
2. Методы
Информация о протеоме и геноме штамма NCTC13017 Le-gionella waltersii была взята из открытой базы данных NCBI Genome. Для обработки данных была использована програм-ма MS Excel, позволяющая работать с данными в виде электронных таблиц. В работе с данными были использованы следующие возможности: преобразование файла TXT в электронную таблицу формата XLCX, применение таких спецсимволов, как “=” для написания формул, “&” для соединения значений в одну строку, “$” для закрепления строк и столбцов, “;” для разделения аргументов формул и прочие; были использованы специальная вставка значений без соответствующих формул, комментирование ячеек. Также с помощью горячих клавиш Ctrl + C, Ctrl + V, Ctrl + F, Ctrl + A, Ctrl + X осуществлялось копирование и вставка данных, поиск и замена значений, выделение всех значений листа и удаление выделенного диапазона соответственно. С помощью функции ВПР была осуществлена связь значений между двумя таблицами, а именно значениями из одной таблицы дополнялась другая. Приведение таблицы к удобному для восприятия виду осуществлялось с помощью изменения ширины столбцов и их перестановки. Для поиска необходимого типа значений применялись сортировка и фильтрация строк по значениям.
Для получения данных из других таблиц и листов ЭТ применялись функции СЧЁТЕСЛИ и СЧЁТЕСЛИМН, позволяющие подсчитать количество данных, подходящих под указанные условия. Также использовалась специальная вставка значений, для использования значений отдельно от их формул. С помощью этих методов была получена таблица различных типов генов и их расположения на разных цепях ДНК.
Вероятность распределения различных типов генов в хромосоме рассчитывалась с помощью функции БИНОМ.РАСП следующим образом: в связи с необходимостью учитывать обе ситуации, когда минимум генов наблюдается на либо на «+»-цепи, либо на «-»-цепи, находится минимальное значение для БИНОМ.РАСП и умножается на два (в силу симметричности биномиального распределения).
Для нахождения количества нуклеотидов и k-меров в геноме использовалась программа EMBOSS wordcount. Для нахождения GC-состава использовалась программа EMBOSS geecee
Для нахождения доли аминокислот с гидрофобными остатками в белках были использованы данные о кодирующих последовательностях ДНК Legionella waltersii [5], которые были переведены в соответствующие последовательности аминокислот с помощью программы EMBOSS transeq и специального скрипта на python (Сопроводительные материалы: hydrophobic.py).
3. Результаты и обсуждение
3.1 Длины ДНК и состав генома
Геном Legionella waltersii представлен единственной хромосомой, чья длина составляет 3.735.697 пар оснований [4].
3.2 Распределение генов в хромосоме
В Таблице 1 представлены данные о количестве генов разных классов, их распределение на «+»- и «-»-цепях, а также рассчитанные для каждого класса генов вероятности их случайного распределения. Из представленных данных видно, что для транслируемых областей, псевдогенов и тРНК распределения по цепям ДНК вероятнее всего не является случайным, причём тРНК и псевдогены в основном сосредоточены на «-»-цепи, в то время как более половины всех транслируемых генов расположены на «+»-цепи. Обсуждение этих результатов приводится в пункте 3.4.
Табл.1 Распределение генов в хромосоме
| Класс гена | "+"-Цепь | "-"-Цепь | Всего | Вероятность | Случайно |
|---|---|---|---|---|---|
| транслируемые | 1676 | 1593 | 3269 | 0,16 | нет |
| псевдогены | 40 | 50 | 90 | 0,34 | нет |
| всего (белки) | 1716 | 1643 | 3359 | ||
| tRNA | 16 | 27 | 43 | 0,12 | нет |
| rRNA | 6 | 3 | 9 | 0,5 | да |
| ncRNA | 1 | 0 | 1 | 1 | -* |
| tmRNA | 1 | 0 | 1 | 1 | - |
| RNase_P_RNA | 0 | 1 | 1 | 1 | - |
| SRP_RNA | 1 | 0 | 1 | 1 | - |
| всего (РНК) | 25 | 31 | 56 | ||
| всего | 1741 | 1674 | 3415 |
*прочерк означает, что в данном случае недостаточно данных для того, чтобы судить о случайности
3.3 GC-состав
Геном Legionella waltersii на 39% состоит из нуклеотидов G и C, что соответствует диапазону GC-составов от 36,7% до 51,1% для видов рода Legionella [6].
3.4 Нуклеотидный состав генома
Частоты встречаемости нуклеотидов представлены в Таблице 2. Несложно заметить, что представленные данные удовлетворяют второму правилу Чаргаффа, так как число аденинов равно числу тиминов, то же верно и для цитозина с гуанином. Предполагается, что причиной этому служат транслокации и инверсии, влияющие на геном организмов в процессе их эволюции [7].
Табл.2 Частоты встречаемости нуклеотидов в геноме Legionella waltersii
| Нуклеотид | Частота встречаемости |
|---|---|
| A | 0,305 |
| T | 0,303 |
| G | 0,196 |
| C | 0,196 |
3.5 Анализ k-меров длины 2
Распределение k-меров длины 2 представлено на Рисунке 2, где мы можем отчётливо видеть, что k-мер GC перепредставлен в геноме Legionella waltersii, в то же время в значительной степени недопредставлен k-мер CG.
3.6 Расположение рибосомальных белков и РНК
Как можно видеть из представленных данных (Сопроводительные материалы: Лист “ribosomal_table”) рибосомальные белки и РНК образуют два крупных кластера и 4 небольши обособленных группы, причём самый крупный кластер и две из четырёх групп расположены на «+»-цепи, что может отчасти объяснять неслучайность большего количеста генов на «+»-цепи. Также, если рассматривать только рибосомальные РНК (Сопроводительные материалы: Лист “ribosomal_table”), то можно заметить, что гены рРНК расположены в три группы одинакового состава из трёх генов: 5S-РНК, 23S-РНК и 16S-РНК, причём две из этих групп находятся на «+»- цепи.
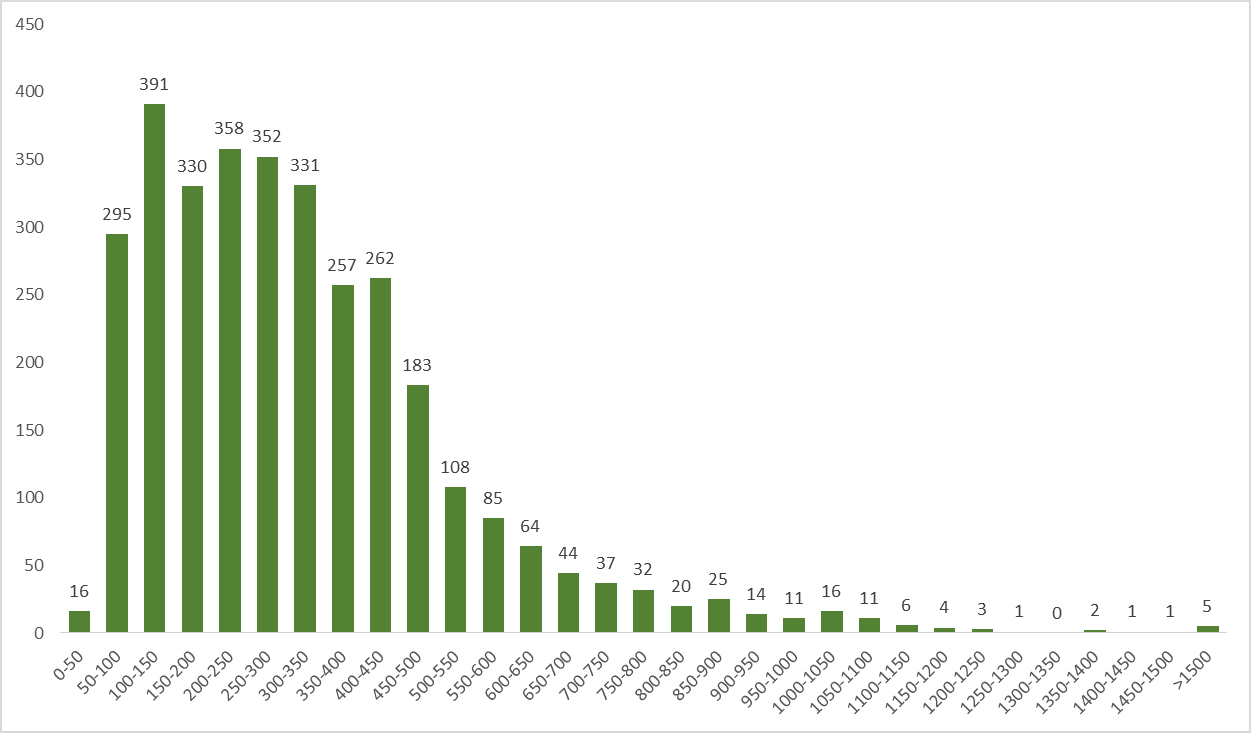
3.7 Распределение длин белков
В гистограмме (Рисунок 2) можно выделить два пика, соответствующие диапазонам 100-150 аминокислотных остатков и 200-250 а.о. В эти диапазоны попадет большое количество разнообразных белков, выполняющие множество разных функций, кроме этого в них содержится много гипотетических белков (Сопро-водительные материалы: Лист “prot_length”). Поэтому не представляется возможным однозначно установить связь между функцией белков и их длиной в геноме Legionella waltersii.
3.8 Доля аминокислот с гидрофобными остатками в белках
Доля аминокислот с гидрофобными остатками в белках Для рассмотрения доли аминокислот с гидрофобными остатками в белках в качестве таковых рассматривались валин, изолейцин, лейцин, метионин, фенилаланин, пролин, аланин и триптофан. Согласно полученным данным (Сопроводительные материалы: Лист “hydrophobic”), в среднем в белках Legionella waltersii содкрится 0,433 аминокислот с гидрофобными остатками, при этом максимальное и минимальное значения – 0,703 и 0,2 соответственно – принадлежат гипотетическим белкам. Среди белков, где доля искомых аминокислот в белке превышает 0,6, значительно преобладают мембранные белки, например субъединица C АТФ-синтазы и цитохром c (Сопроводительные материалы: Лист “hydrophobic”). Таких белков 44. Это можно объяснить тем, что для мембранных белков необходимо, чтобы большая часть их поверхности состояла из гидрофобных остатков, что позволяет им встраиваться в мембрану.
Заключение
Таким образом, подтверждается предположение, что большинство генов в геноме Legionella waltersii распределены неслучайным образом, что, возможно, объясняется расположением большой группы рибосомальных генов на одной из цепей, однако строгое доказательсво этого факта требует дальнейших исследований. Помимо этого, длины преобладающего большинства белков Legionella waltersii лежат в диапазоне 50-500 а.о., около 44 белков имеют высокое содержание аминокислот с гидрофобными остатками. В геноме Legionella waltersii также недпредставлен динуклеотид CG и перепредставлен динуклеотид GC. Несмотря на это, Legionella waltersii характеризуется относительно низким GC-составом, что, вероятно, связано с отсутствием необходимости защищать ДНК от денатурации в условиях обитания данной бактерии.
Сопроводительные материалы
1. Таблица с сопроводительными материалами.
Список литературы
1. Benson, RF.; Thacker, WL.; Daneshvar, MI.; Brenner, DJ. (Jul 1996). "Legionella waltersii sp. nov. and an unnamed Legionella ge-nomospecies isolated from water in Australia". International Journal of Systematic Bacteriology. 46 (3): 631–4. doi:10.1099/00207713-46-3-631. PMID 8782669.
2. Lawrence K. Altman. In Philadelphia 30 Years Ago, an Eruption of Illness and Fear. New York Times (1 августа 2006).
3. König, C.; Hebestreit, H.; Valenza, G.; Abele-Horn, M.; Speer, CP. (Oct 2005). "Legionella waltersii--a novel cause of pneumonia?". Ac-ta Paediatr. 94 (10): 1505–7. doi:10.1080/080352505100. PMID 16299887.
4. Legionella waltersii strain NCTC13017 genome assembly, chromo-some: 1. NCBI. URL: https://www.ncbi.nlm.nih.gov/nuccore/LT906442.1
5. Данные о геноме Legionella waltersii NCTC13017. URL: ftp://ftp.ncbi.nlm.nih.gov/genomes/all//GCF900/187/095/GCF_900187095.1_51699_A01
6. Burstein, D., Amaro, F., Zusman, T. et al. Genomic analysis of 38 Legionella species identifies large and diverse effector reper-toires. Nat Genet 48, 167–175 (2016). https://doi.org/10.1038/ng.3481
7. Albrecht-Buehler G (2006). "Asymptotically increasing compliance of genomes with Chargaff's second parity rules through inversions and inverted transpositions". Proc Natl Acad Sci USA. 103 (47): 17828–17833.