Работа с UniProt
Halothiobacillus neapolitanus - хемолитоафтотрофные Грам-отрицательные бактерии, принадлежащие к семейству Halothiobacillaceae внутри класса Gammaproteobacteria. Эти организмы могут фиксировать CO2 за счёт окисления серы. В этом ей помогают специализированные органеллы - карбоксисомы, которые содержат фиксирующий углерод фермент - рибулозодифосфаткарбоксилазу (сокращённо - Рубиско), что позволяет использовать Halothiobacillus neapolitanus как модельный объект в изучении карбоксисом и их роли в фиксации углерода [1]. Рубиско катализирует реакцию карбоксилирования рибулозо-1,5-бисфосфата, в результате которой получается две молекулы 3-фосфоглицерата, и состоит из двух различых субъединиц - малой и большой, и именно о последней пойдёт речь в этом отчёте.
Информация о белке из базы данных
Для поиска белка используем доступный нам идентификатор AAC32549.1. Для на сайте UniProt в форме "Retrieve/ID mapping" выберем в поле "from" опцию "EMBL/GenBank/DDBJ CDS" и опцию "UniProtKB" в поле "to". В резултате заапроса получаем одну запись об искомом белке, где находим информацию о его расположении в различныз базах данных (Табл.1).
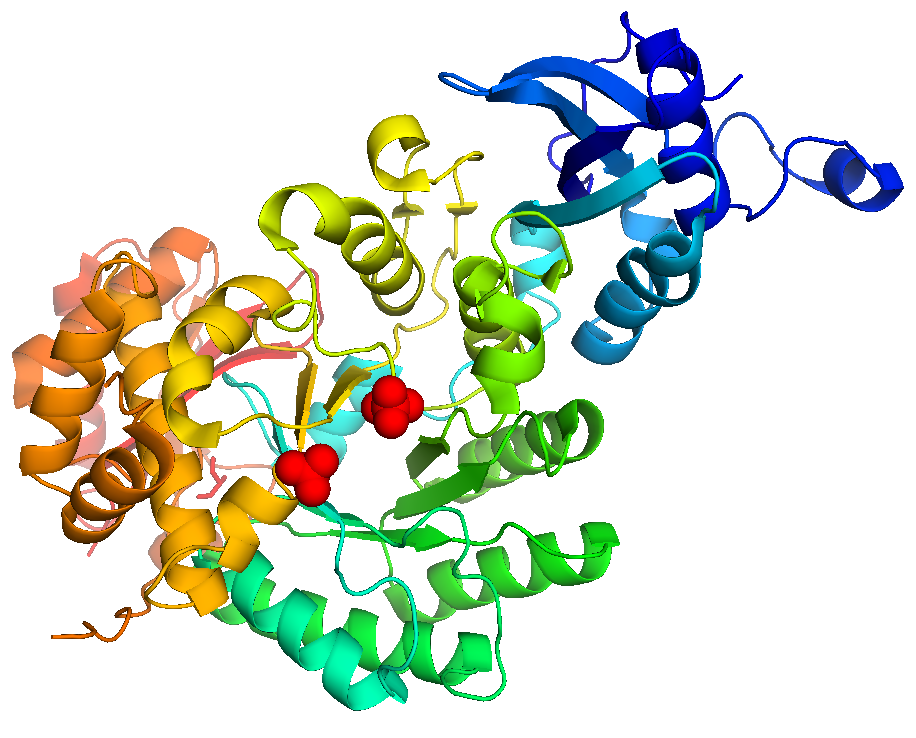
Таблица 1. Основная информация о белке Pseudoazurin из UniProt
| Раздел UniProtKB | Swiss-Prot |
| UniProt ID | RBL1_HALNC |
| UniProt AC | O85040; D0KZ92 |
| EMBL AC (ENA/GenBank/DDBJ) | AAC32549.1; ACX95765.1 |
| PDB ID | 1SVD; 6UEW |
| Длина, а.о. | 473 |
| Молекулярная масса, Да | 52636 |
| Рекомендуемое название | Ribulose bisphosphate carboxylase large chain |
| Альтернативное название | Form 1 RuBisCO |
Структура и функции белка
Молекула Рубиско состоит из 8 молекул малых субъединиц и 8 молекул больших субъединиц, последние из которых объединены в димеры, которые выполняют каталитическую функцию, то есть содержат каталититческий центр [2]. В этом центре содержится ион магния, который помогает связывать молекулу углекислого газа в активном центре, направляя её на остаток лизина Lys210, который образует карбамат с молекулой CO2[3]. Рубиско катализаирует реакцию карбоксилирования рибулозо-1,5-бисфосфата с образованием двух молекул 3-фосфоглицерата, однако также может протекать реакция между кислородом как очень похожим на углекислый газ и рибулозо-1,5-бисфосфатом с образованием фосфогликолата и 3-фосфоглицерата, что обуславливает процессы фотодыхания.
Запросы в UniProt
Выполним различные запросы в продвинутом режиме поисковой строки (Табл.2)
Таблица 2. Запросы в UniProt и их результаты
| Tекст запроса | Swiss-Prot | TrEMBL | Результат поиска |
| name:"rubisco large subunit" existence:"Evidence at protein level [1]" NOT name:binding | 44 | 4 | Записи о всех молекулах больших субъединиц Рубиско, чье существование подтверждено на уровне белка (мы исключили специально записи со словом binding, так как иначе помимо искомых записей повляются записи о шаперонах) |
| name:"rubisco large subunit" existence:"Evidence at protein level [1]" database:(type:pdb) NOT name:binding | 17 | 2 | То же, что и в предыдущем запросе, только дополнительно отбираются те записи, для которых существует структура PDB. |
| name:"rubisco large subunit" existence:"Evidence at protein level [1]" NOT database:(type:pdb) NOT name:binding | 27 | 2 | Записи о всех молекулах больших субъединиц Рубиско, чье существование подтверждено на уровне белка, но для которых не существует соответсвующей структуры PDB |
| name:"rubisco large subunit" taxonomy:proteobacteria pdb | 3 | 0 | Записи о всех молекулах больших субъединиц Рубиско организмов в группе Proteobacteria, для которых существует структура PDB. |
| name:"rubisco large subunit" NOT taxonomy:viridiplantae NOT taxonomy:bacteria pdb | 1 | 1 | Записи о всех молекулах больших субъединиц Рубиско тех организмов, что не принадлежат к группе Proteobacteria или Viridiplantae (т.е. разнообразные водоросли), для которых существует структура PDB. |
| "ribulose bisphosphate carboxylase large chain" existence:"Inferred from homology [3]" AND reviewed:yes | 597 | 0 | Все записи в базе Swiss-Prot (то есть те, что проверены вручную),но данные о белке получены исходя из информации о гомологичных белках. |
| taxonomy:"halothiobacillus neapolitanus" goa:(carboxysome) | 10 | 0 | Все записи о белках, связанных с карбоксисомой в Halothiobacillus neapolitanus |
| taxonomy:chromatiales gene:cbbl AND reviewed:yes | 7 | 0 | Все записи о белках гена cbbL (кодирующего большую субъединицу Рубиско) в разных организмах из порядка chromatiales, проверенные вручную. |
| gene:cbbl pdb | 9 | 1 | Теперь найдем все такие зариси о белке, кодируемым геном ccbL, для которых существует структура PDB. (интересно, что для одной записи есть PDB, но нет ручной проверки, при том, что существование белка подтверждено.) |
Изучение кластеров UniRef
Кластер UniRef100
Колическтво белков в кластере: 1
ID кластера: UniRef100_O85040
Изучаемый белок в данном кластере является единственным и при этои референсным.
Посмотреть кластерКластер UniRef90
Колическтво белков в кластере: 131
ID кластера: UniRef90_O85040
В данном кластере наш белок явлется референсным, однако не является сидом, так как присутствуют белки большей длины. Помимо изучаемого белка в кластере присутствуют ещё четыре записи из Swiss-Prot, принадлежащие организмам из разных семейств.
Посмотреть кластерКластер UniRef50
Колическтво белков в кластере: 14814
ID кластера: UniRef50_O85040
Этот кластер также включает изучаемый белок в качестве референсного, но опять же он не является сидом, помимо этого среди ограомного числа белков, включенных в кластер, присутствуют 110 записей из Swiss-Prot, принадлежащие не только прокриотам, но и эукариотам, например, среди них встречается много растений и водорослей.
Посмотреть кластерСравнение протеомов
Для сравнения были взяты протеомы бактерии Halothiobacillus neapolitanus и состоящей с ней в одном порядке Thioalkalivibrio sp. (strain K90mix), выбор которой обусловлен тем, что для неё существует почти полный геном, в котором можно найти ген, кодирующий изучаемый белок. Результаты сравнения представленны в Табл.3. В протеомах обеих бактерий производился поиск белков связанных с карбоксисомой, которая участвует у них в фиксации углерода.
Таблица 3. Сравнение протеомов Halothiobacillus neapolitanus и Thioalkalivibrio sp. (strain K90mix)
| Организм | Halothiobacillus neapolitanus | Thioalkalivibrio sp. |
|---|---|---|
| ID протеома | up000009102 | up000009099 |
| Общее количество белков | 2353 | 2823 |
| Количество записей в Swiss-Prot | 15 | 3 |
| Запрос в UniProt (трансмембранные белки) | annotation:(type:transmem) AND organism:"Halothiobacillus neapolitanus (strain ATCC 23641 / c2) (Thiobacillus neapolitanus) [555778]" AND proteome:up000009102 | annotation:(type:transmem) AND organism:"Thioalkalivibrio sp. (strain K90mix) [396595]" AND proteome:up000009099 |
| Количество трансммбранных белков | 426 | 541 |
| Количество трансммбранных белков в Swiss-Prot | 2 | 2 |
| Запрос в UniProt (ферменты) | ec:* AND organism:"Halothiobacillus neapolitanus (strain ATCC 23641 / c2) (Thiobacillus neapolitanus) [555778]" AND proteome:up000009102 | ec:* AND organism:"Thioalkalivibrio sp. (strain K90mix) [396595]" AND proteome:up000009099 |
| Количество ферментов | 642 | 541 |
| Количество ферментов в Swiss-Prot | 6 | 2 |
| Запрос в UniProt (белки карбоксисомы) | name:carboxysome AND organism:"Halothiobacillus neapolitanus (strain ATCC 23641 / c2) (Thiobacillus neapolitanus) [555778]" AND proteome:up000009102 | name:carboxysome AND organism:"Thioalkalivibrio sp. (strain K90mix) [396595]" AND proteome:up000009099 |
| Количество белков карбоксисомы | 8 | 4 |
| Количество записей о белках карбоксисомы в Swiss-Prot | 8 | 0 |
Литература
[1]Reclassification of Halothiobacillus hydrothermalis and
Halothiobacillus halophilus to Guyparkeria gen. nov. in the
Thioalkalibacteraceae fam. nov., with emended descriptions of
the genus Halothiobacillus and family Halothiobacillaceae, Boden, Int J Syst Evol Microbiol 2017;67:3919–3928
DOI 10.1099/ijsem.0.002222
[2]Oltrogge, L. M., Chaijarasphong, T., Chen, A. W., Bolin, E. R., Marqusee, S., & Savage, D. F. (2020). Multivalent interactions between CsoS2 and Rubisco mediate α-carboxysome formation. Nature Structural & Molecular Biology. doi:10.1038/s41594-020-0387-7
[3]Stec B. Structural mechanism of RuBisCO activation by carbamylation of the active site lysine. Proc Natl Acad Sci U S A. 2012;109(46):18785-18790. doi:10.1073/pnas.1210754109