Комплексы ДНК-белок и вторичная структура РНК¶
Задание 1¶
Упражнение 1¶
Для работы с программой einverted из базы данных PDB была взята последовательность тРНК, входящей в структуру PDB ID: 1o0b. На вход программе был дан текстовый файл с последовательностью нуклеотидов (первый нуклеотид - урацил - был удален из последовательности, так как его нет в pdb-файле). Параметр treshold был выбран равным 0, так как при значени этого параметра по умолчанию программа не находит инвертированных участков. В результате был получен следующий файл, в котором содержится один комплементарный участок, относящийся к акцепторному стеблю тРНК. Программа find_pair по сравнению с einverted смогла найти большее количество водородных связей и комплементарных учатков, в то время как einverted нашла только один компементарный участок (см. Табл.1).
Упражнение 2¶
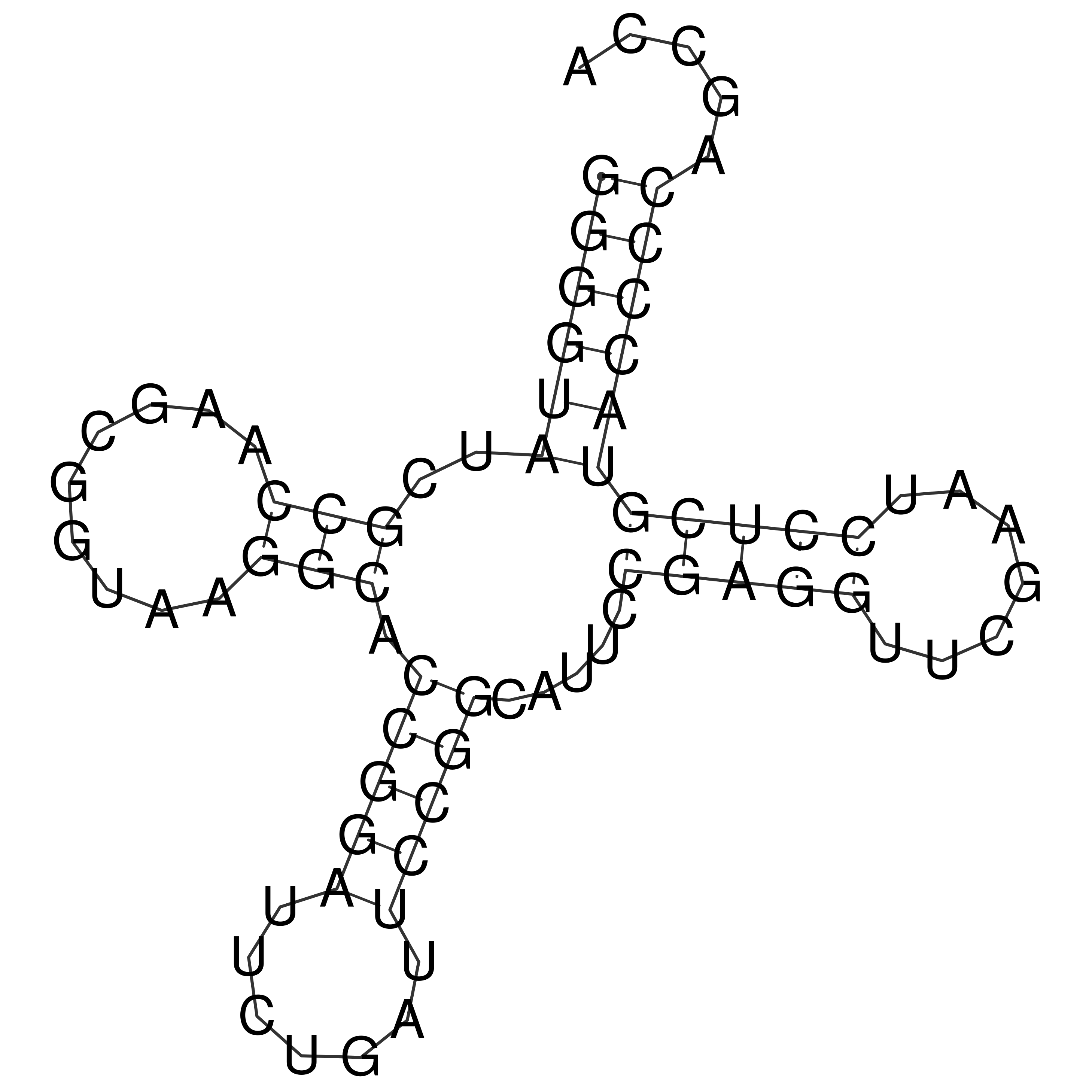
С помощью следущей команды было выполнено предсказание вторчной структуры РНК по алгоритму Зукера.
export PATH=${PATH}:/home/preps/golovin/progs/bin
cat 1o0b_trna.fasta | RNAfold --MEAРезультаты работы алгоритма были получены в виде картинки (Рис.1), на которой изображена вторичная структура данной тРНК.
Сравнение всех подходов к предсказанию вторичной структуры РНК представлено в Таблице 1.
|
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
|
предсказано 6 пар из 6 реальных |
предсказано 6 пар из 6 реальных
|
D-стебель |
5'-910-912-3' |
- |
предсказано 3 пар из 3 реальных |
T-стебель |
5'-949-953-3' |
- |
предсказано 5 пар из 7 реальных |
Антикодоновый стебель |
5'-926-933-3' |
- |
предсказано 5 пар из 5 реальных |
Общее число представленных в стеблях канонических пар нуклеотидов |
18 |
6 |
19 |
Задание 2¶
Упражнение 1¶
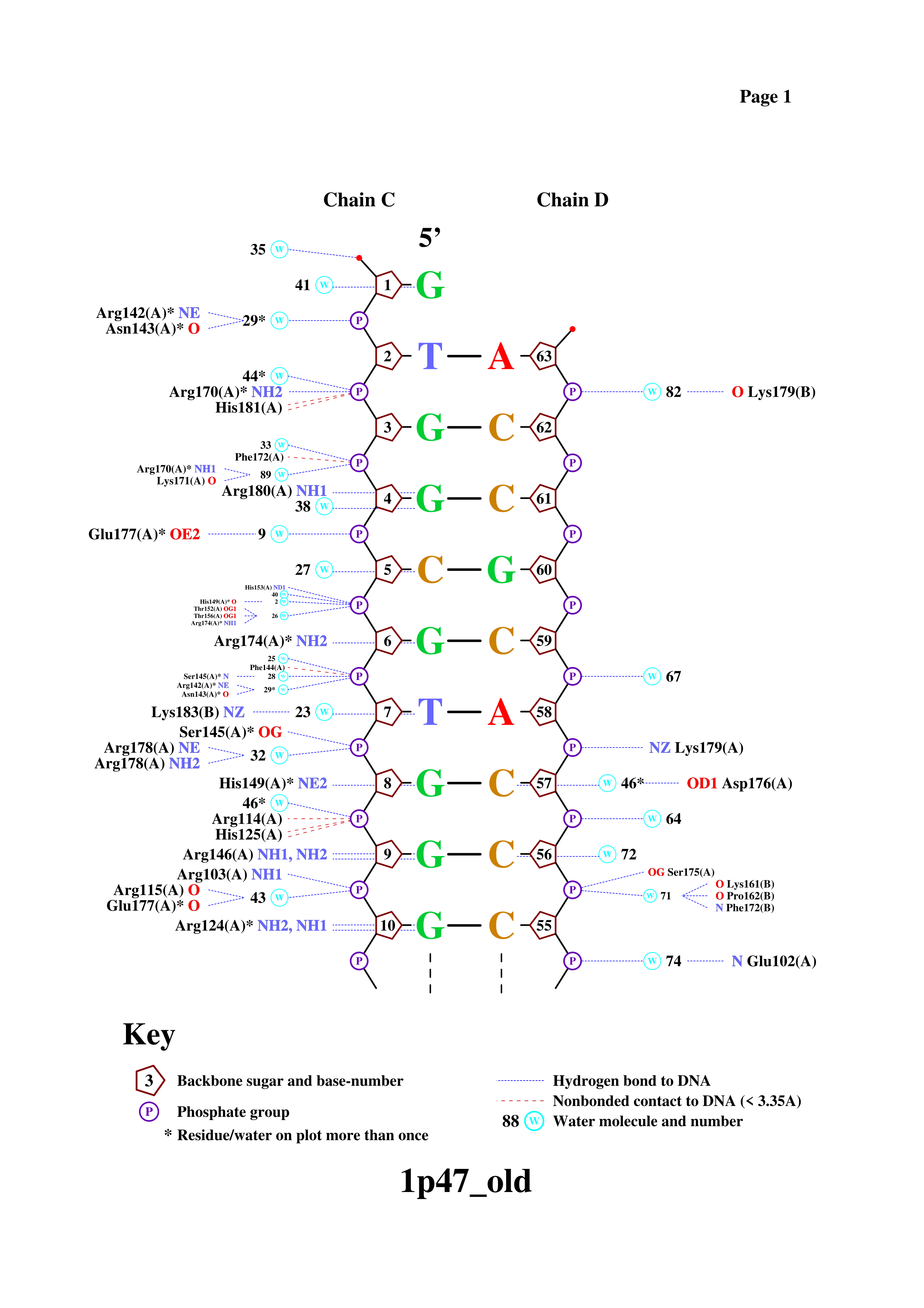
В данном упражнении было необходимо найти и посчитать различые виды связей между атомами белка и ДНК в структуре PDB ID: 1P47. Сперва для дальнйших расчетов нужно было определить множества атомов, удоволетворяющих определенным условиям. По данной ссылке можно ознакомиться со скриптом, орпеделяющим нужные меожества атомов. Также необходимо было написать скрипт, демонстрирующий ДНК и обозначенные атомы.
Упражнение 2¶
В Таблице 2 представленны подсчитанные с помощью скрипта различные виды связей ДНК-белок. Из представленных данных можно сделать вывод, что больше всего взаимодействий ДНК-белок в структуре 1P47 возникает в области большой бороздки, что можно было бы предположить, вглянув на форму белка, который "ложится" вдоль большой бороздки (представленный белок как раз имеет мотив так называемых "Цинковых пальцев", который ответственнен за взаимодейтсвие с молекулами нуклеиновых кислот).
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 2 | 34 | 36 |
| остатками фосфорной кислоты | 19 | 21 | 40 |
| остатками азотистых оснований со стороны большой бороздки | 25 | 83 | 108 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 1 | 1 |