Трансмембранные белки¶
Задание 1. Знакомство с базой данных OPM¶
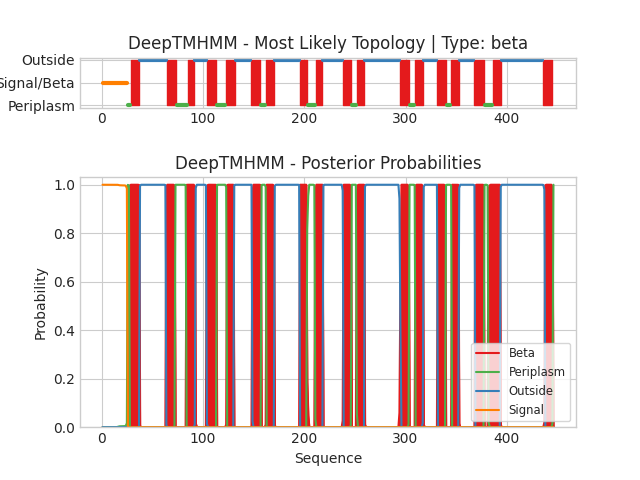
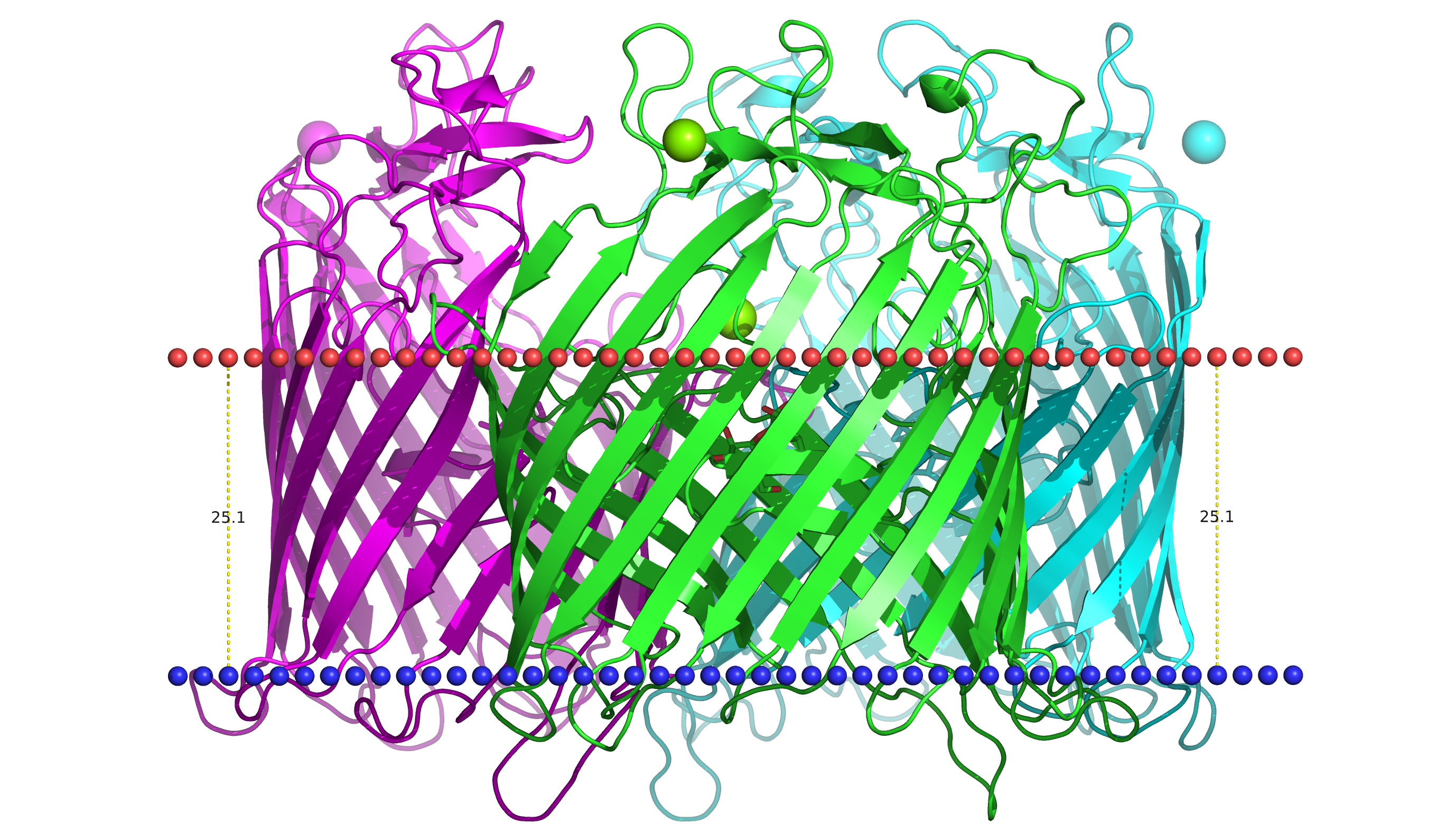
Для выполнения этого задания из базы данных OPM был выбран мальтопорин, принадлежащий Escherichia coli. Данный тримерный трансмембранный белок относится к классу белков, имеющих структуру β-бочонка (Рис.1) и участвует в транспорте мальтозы и мальтодекстринов. Также известно, что он может играть роль рецептора для некоторых бактериофагов, в том числе и для λ.
Мальтопорин Escherichia coli локализован во внешней мембране. Согласно информации из баз данных, в толщину гидрофобная часть белка составляет 25.1 Å, что подтверждается измерением растояния с помощью инструмента Measure distance в программе Pymol.(Рис.1)
| Характеристика | Данные |
|---|---|
| Тип белка | Трансмембранный белок, бета-бочонок |
| Суперсемейство | Порины, транспортрующие сахара |
| Семейсво | Мальтопорин-подобные белки |
| Организм | Escherichia coli |
| Локализация | Внешняя мембрана Грам-отрицательной бактерии |
| Uniprot ID | LAMB_ECOLI |
| PDB | 1AF6 |
| Толщина гидрофобного слоя | 25.1 Å |
| Число трансмембранных структур | 54 (в тримере) |
| Средняя длина трансмембранных структур | 9-11 |
| Топология | N-конец белка в периплазме |
Согласно базе данных, белок имеет 18 гидрофобных участков длиной примерно 9-12 остатков: 1(2-13),2(39-48),3(58-68),4(75-88),5(98-103),6(125-132),7(138-146),8(170-179),9(185-194),10(213-221),11(227-235),12(269-278),13(284-293),14(305-314),15(320-329),16(343-352),17(361-370),18(411-420).