

Работа с RasMol и 3DNA: анализ структур A- и B-форм ДНК
| A-форма | B-форма | Файл DNA32.pdb | |
| Тип спирали (правая или левая) | Правый | Правый | Правый |
| Шаг спирали (Å) | 28,02 | 33,75 | 31,29 |
| Число оснований на виток | 11 | 10 | 10 |
| Ширина большой бороздки | 7,98 (Гуанозин) | 17,21 (Гуанозин) | 15,7 (Аденозин) |
| Ширина малой бороздки | 16,80 (Аденозин) | 11,69 (Цитидин) | 11,92 (Тимидин) |
Исследуемая нуклеиновая кислота файла DNA32.pdb представляет собой B - форму ДНК.
Торсионные углы ДНК
A - форма ДНК
Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2
2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2
7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2
9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2
13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
16 C -51.7 174.8 41.7 79.1 --- --- -157.2
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -51.7 174.8 41.7 79.0 --- --- -157.2
2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2
15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2
16 G --- 174.8 41.7 79.1 -147.7 -75.1 -157.2
B - форма ДНК
Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0
2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0
14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
16 C -29.9 136.4 31.1 143.4 --- --- -98.0
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C -29.9 136.4 31.1 143.4 --- --- -98.0
2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0
5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0
14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9
15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0
16 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0
Исследуемая ДНК 32
Main chain and chi torsion angles:
Note: alpha: O3'(i-1)-P-O5'-C5'
beta: P-O5'-C5'-C4'
gamma: O5'-C5'-C4'-C3'
delta: C5'-C4'-C3'-O3'
epsilon: C4'-C3'-O3'-P(i+1)
zeta: C3'-O3'-P(i+1)-O5'(i+1)
chi for pyrimidines(Y): O4'-C1'-N1-C2
chi for purines(R): O4'-C1'-N9-C4
Strand I
base alpha beta gamma delta epsilon zeta chi
1 C --- --- -133.5 119.1 -154.8 -117.7 -136.3
2 G -38.1 -171.5 22.8 145.4 164.0 -63.3 -96.9
3 C -56.4 -127.5 20.2 167.6 -176.5 -144.5 -82.9
4 A -44.8 134.7 80.8 94.0 -177.1 -105.6 31.1
5 A 145.5 -129.9 178.1 106.2 -143.4 -76.6 -153.9
6 G -21.8 -170.4 -23.1 172.1 -147.6 -133.6 -85.3
7 C -52.5 145.7 44.0 123.1 -100.7 178.6 -120.5
8 T 62.4 -175.3 -131.5 167.3 -120.2 -166.3 -104.9
9 G -56.0 146.7 30.3 131.3 150.8 -53.6 -110.1
10 G -58.0 -133.2 32.1 165.6 -175.2 -145.7 -82.3
11 C -66.3 152.4 72.8 153.4 -157.4 -112.6 -109.2
12 G 41.9 -177.3 -72.5 155.1 --- --- -91.2
Strand II
base alpha beta gamma delta epsilon zeta chi
1 G 14.4 -170.6 -26.1 124.9 --- --- -110.7
2 C 0.8 122.3 -2.8 157.3 -178.1 -143.7 -81.9
3 G -69.6 -159.7 52.8 154.8 -127.6 159.3 -94.0
4 G -129.8 147.6 81.8 130.2 166.5 -83.7 -106.8
5 T 39.9 114.7 -24.6 168.3 -90.4 170.8 -74.0
6 C -77.6 142.8 83.0 90.3 160.7 -83.7 -132.4
7 G -2.9 165.8 -39.2 173.6 -161.3 -133.6 -81.0
8 A -36.5 140.0 33.4 162.1 -112.7 -176.8 -96.4
9 A 9.3 162.8 -11.3 158.3 -153.5 -145.4 93.0
10 C -45.0 120.5 69.3 74.3 -152.8 -115.6 -149.2
11 G -1.5 173.3 -3.9 163.2 -154.7 -128.8 -99.7
12 C --- --- 158.0 101.5 -167.8 -124.5 -145.9
Classification of each dinucleotide step in a right-handed nucleic acid
structure: A-like; B-like; TA-like; intermediate of A and B, or other cases
step Xp Yp Zp XpH YpH ZpH Form
1 CG/CG -1.98 8.82 0.22 -2.06 8.77 0.98 B
2 GC/GC -2.78 8.81 -0.24 -1.30 8.74 -1.13 B
3 CA/GG -1.78 0.34 8.30 1.79 0.78 -2.99
4 AA/TG -1.91 -4.12 6.02 -1.24 -5.21 3.44
5 AG/CT -3.49 9.05 -0.06 -5.75 7.90 4.38
6 GC/GC -3.15 9.01 -0.73 -2.70 8.82 -1.88 B
7 CT/AG -3.19 8.80 -0.56 -3.95 8.74 1.12 B
8 TG/AA -2.54 8.22 -0.23 -2.45 8.20 -0.67
9 GG/CA -2.24 8.53 0.29 -13.06 7.93 -2.85
10 GC/GC -2.13 8.83 -0.14 0.38 8.38 -2.79 B
11 CG/CG -2.83 9.01 0.17 -2.67 9.01 -0.20 B
Углы дельта достаточно стабильны, и исследуемая ДНК 32 по этим углам соответствует B - форме. Другие углы менее стабильны, определить форму ДНК по ним сложнее. Подтверждением предположения о форме ДНК может быть таблица, где указаны формы для каждого участка ДНК.
Стопочные модели ДНК
Посредством программы pdb2img были построены стопочные модели ДНК структур.
A - форма ДНК


B - форма ДНК

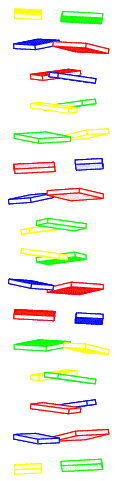
Исследуемая ДНК 32

Азотистые основания ДНК 32 расположены относительно параллельно и не образуют канала между собой. Такие свойства не характерны для азотистых оснований A - формы ДНК и характерны для азотистых оснований B - формы ДНК.
На главную страницу третьего семестра