Структура РНК
1. Полученный PDB код 1i9v.
Структура PHENYLALANINE TRANSFER RNA тРНК и из SACCHAROMYCES CEREVISIAE (YEAST) организма описана в записи PDB, идентификаторы цепей тРНК MOL_ID: 1, ещё макромолекулы есть в структурах: WYBUTOSINE, NEOMYCIN, 5-METHYLURIDINE 5'-MONOPHOSPHATE (главная цепь).
2. Последовательность тРНК:
SEQRES 1 A 76 G C G G A U U U A G C U C
SEQRES 2 A 76 A G U U G G G A G A G C G
SEQRES 3 A 76 C C A G A C U G A A YG A U
SEQRES 4 A 76 C U G G A G G U C C U G U
SEQRES 5 A 76 G 5MU U C G A U C C A C A G
SEQRES 6 A 76 A A U U C G C A C C A
Модифицированные основания тРНК:
YG A 37 WYBUTOSINE
5MU A 54 5-METHYLURIDINE 5'-MONOPHOSPHATE
Цепь РНК (номера начального и конечного нуклеотидов):
1 – 76
"Insertion codes" нет.
3. Анализ структуры программой find_pair.
Было найдено 3 спирали:
Helix #1 (13): 1 – 13
Helix #2 (13): 14 – 26
Helix #3 (1): 27
Длина соответственно равна 13, 13 и 1.
GCGGAUU U A GCUCAG UUG G GA GAGCGCCAG A CU GA A YG A U CUGGA GGU C CUGUG5MU U C GAUCCACAGAAUUCGC ACCA
Нуклеотидные пары, найденные программой:
GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAYGAUCUGGAGGUCCUGUG5MUUCGAUCCACAGAAUUCGCACCA
4. Исследуемая тРНК.
define helix1 1-7, 49-54, 58, 61-72 define helix2 8, 10-15, 22-30, 32, 33, 36, 38, 40-44, 48 define helix3 19, 56 select helix1 color red select helix2 color green select helix3 color magenta select all wireframe off backbone 100 select G1A.O5.* label 1 select A76A.P label 76 set fontsize 17 colour label purple set fontstroke 1
Анализ структуры РНК:
Пример внеспирального стекинг - взаимодействия между основаниями.
Примеры водородных связей между основаниями, не сводящийся к Уотсон-Криковскому спариванию комплементарных оснований:
G4 U69
5MU54 A58
A38 C32
A44 G26.
Спирали исследуемой РНК похожи на спирали A - формы ДНК.
Ширина большой бороздки: 11,32 Å (Аденозин);
Ширина малой бороздки: 15,17 Å (Аденозин);
Число оснований на виток 11.
На рисунке представлены A - форма ДНК, B - форма ДНК и исследуемая РНК.
5. Посредством программы einverted были найдены инвертированные последовательности РНК.
RNA: Score 16: 8/10 ( 80%) matches, 0 gaps
22 gagcgccaga 31
|| | |||||
48 ctggaggtct 39
RNA: Score 15: 5/5 (100%) matches, 0 gaps
49 ctgtg 53
|||||
65 gacac 61
GCGGAUUUAGCUCAGUUGGGAGAGCGCCAGACUGAAYGAUCUGGAGGUCCUGUG5MUUCGAUCCACAGAAUUCGCACCA
Сравнение находок.
Программа einverted находит последовательности относительно точно, но не полностью. Это объясняется тем, что искомые последовательности фрагментированы. Поэтому выравнивания строятся неполноразмерные.
GCGGAUU U A GCUCAG UUG G GA GAGCGCCAG A CU GA A YG A U CUGGA GGU C CUGUG5MU U C GAUCCACAGAAUUCGC ACCA
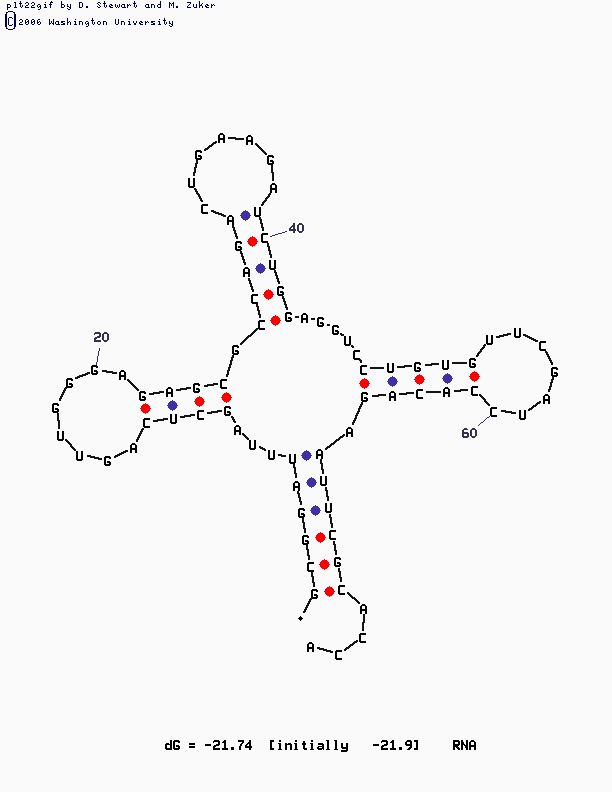
6. Посредством программы Mfold был произведён анализ первичной последовательности тРНК. Программа работала с параметром P равным 12, т. е. рассматривались вторичные структуры с энергиями, отличными от оптимальной не более, чем на 12%.
Результат работы программы, файл № 2.

На главную страницу третьего семестра