Занятие 8. Структура тРНК
-
Полученный PDB-код - 1qu2.
-
В записи содержится структура изолейциновой тРНК (цепь Т), а также
изолейциновой тРНК-синтетазы (цепь А) из STAPHYLOCOCCUS AUREUS, экспрессирующихся в ESCHERICHIA COLI.
-
Последовательность данной тРНК:
GGGCUUGUAGCUCAGGUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCAC
Номер начального нуклеотида -1, конечного - 74. Insertion code "А" имеется у 21-го нуклеотида.
Структура была проанализирована программой find_pair. В результате было обнаружено
две спирали длиной 14 bp, и две длиной 1 bp. На последовательностbи тРНК отмечены разными цветами участки,
входящие в разные спирали.
GGGCUUG UA GCUC AGGUGGUU AGAG CGCACCC CUGAU AAGGGUG AGGU CGGUG GUUCAAG UCCAC UCAGGCC CAC
При помощи Rasmol была создана картинка в формате GIF, на которой
изображена только данная цепь тРНК в остовной модели,
найденные спирали покрашены в разные цвета, при атомах фосфора
5'-концевого и 3'-концевого нуклеотидов подписаны номера остатков(первый нуклеотид последовательности
фосфора не имеет, поэтому подписан второй).

Картинка была получена после использования следующего набора команд:
restrict rna
center selected
backbone 200
wireframe off
select 1-7,66-72
color red
select 49-53,61-65
color purple
select 38-44,26-32
color blue
select 10-13,22-25
color green
background [121,255,255]
set fontsize 14
select rna and 2 and *.p
label <-------------%r
select rna and 74 and *.p
label <-------------%r
Пользуясь средствами RasMol, была проанализирована данная структура.
-
На рисунке приведен пример внеспирального стекинг-взаимодействия между 21-м и 46-м основаниями.
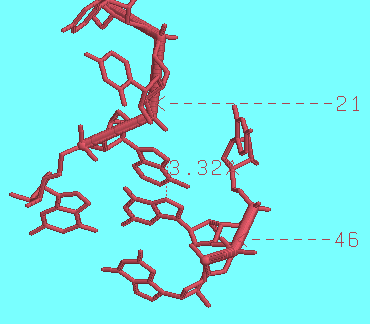
-
Ниже приведены два рисунка иллюстрирующие возможность Уотсон-Криковского спаривания
комплементарных оснований в РНК (пары U-G и U-U):
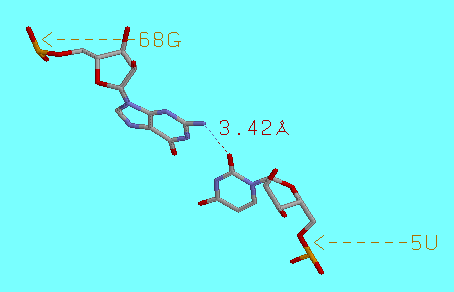
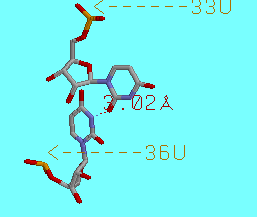
Cпирали РНК более всего напоминают А-форму ДНК, т.к.:
- Имеется отверстие при визуальном наблюдении дуплекса с торца.
- Имеется ~одиннадцать оснований на виток.
Гипотеза подтверждается программой analyze.
Программа einverted из пакета EMBOSS позволяет найти инвертированные
последовательности в НК. C ее помощью были найдены возможные двуспиральные участки в
последовательности исследуемой РНК. Порог Minimum score threshold был установлен равным 12. В случае, если он больше программа
не находила ничего.
Выдача программы выглядела следующим образом:
: Score 16: 8/10 ( 80%) matches, 0 gaps
23 gagcgcaccc 32
|| | |||||
49 ctggagtggg 40
: Score 14: 6/7 ( 85%) matches, 0 gaps
1 gggcttg 7
|| ||||
63 cctgaac 57
А вот, что на этот счет выдает find_pair:
T:...1_:[..G]G-----C[..C]:..72_:T
T:...2_:[..G]G-----C[..C]:..71_:T
T:...3_:[..G]G-----C[..C]:..70_:T
T:...4_:[..C]C-----G[..G]:..69_:T
T:...5_:[..U]U-*---G[..G]:..68_:T
T:...6_:[..U]U-----A[..A]:..67_:T
T:...7_:[..G]G-----C[..C]:..66_:T
T:..49_:[..G]G-*---U[..U]:..65_:T
T:..50_:[..G]G-----C[..C]:..64_:T
T:..51_:[..U]U-----A[..A]:..63_:T
T:..52_:[..G]G-----C[..C]:..62_:T
T:..53_:[..G]G-----C[..C]:..61_:T
T:..54_:[..U]U-**--A[..A]:..58_:T
T:..55_:[..U]U-**+-G[..G]:..18_:T
T:..36_:[..U]U-**+-U[..U]:..33_:T
T:..38_:[..A]A-*---C[..C]:..32_:T
T:..39_:[..G]G-----C[..C]:..31_:T
T:..40_:[..G]G-----C[..C]:..30_:T
T:..41_:[..G]G-----C[..C]:..29_:T
T:..42_:[..U]U-----A[..A]:..28_:T
T:..43_:[..G]G-----C[..C]:..27_:T
T:..44_:[..A]A-*---G[..G]:..26_:T
T:..10_:[..G]G-----C[..C]:..25_:T
T:..11_:[..C]C-----G[..G]:..24_:T
T:..12_:[..U]U-----A[..A]:..23_:T
T:..13_:[..C]C-----G[..G]:..22_:T
T:..14_:[..A]A-**--U[..U]:...8_:T
T:..15_:[..G]G-**+-C[..C]:..48_:T
T:..16_:[..G]G-**--U[..U]:..17_:T
T:..19_:[..G]G-----C[..C]:..56_:T
Находки совсем не совпадают с действительностью (обнаруженные участки действительно принимают участие в формироании спиралей,
но не в таком порядке).
Возможно, это связано с тем, что программа программа создана для того чтобы искать двойные спирали ДНК
("Finds DNA inverted repeats", как сказано в описании к ней), т.е. не учитывает внеспиральные стекинг взаимодействия
и неканонические вододродные связи между парами оснований, которые так важны для формирования пространственной структуры РНК.
Последовательность тРНК была проанализирована программой
mfold. Структура наиболее похожая на реальную приведена ниже (получена второй по счету с параметром P=10)
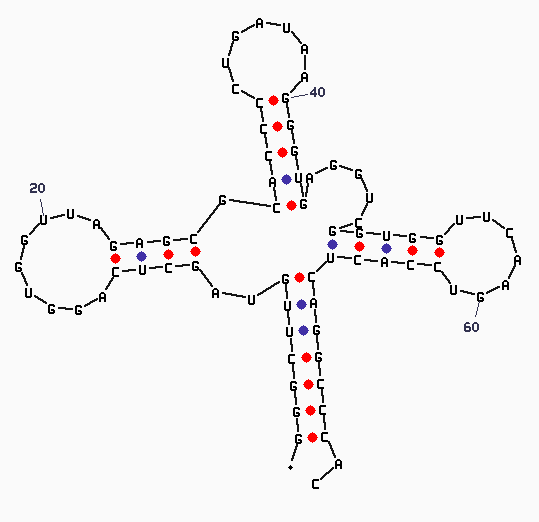
Параметр P указывает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального.
Как видно, отличие от оптимума энергии составляет 10%, что само по себе величина достаточно большая. Этот факт
позволяет понять какую важную роль играют взаимодействие белка с РНК, внеспиральные стекинг взаимодействия между основаниями РНК
для стабилизации ее пространственной структуры.
Вернуться к списку протоколов.
©:Сорокин Максим







