Исследование структуры тРНК
- Краткое описание структуры в файле 2fmt.pdb В файле приведены координаты атомов следующих молекул:
Formyl-methionyl-trnafmet2, цепи C, D
Methionyl-trna fmet formyltransferase, цепи A, B
молекулы из организма ESCHERICHIA COLIДля исследования была выбрана цепь C, представляющая метионил-тРНК со следующей последовательностью:
[1] 5’ CGCGGGG4SUGGAGCAGCCUGGH2UAGCUCGUCGGGOMCUCAUAACCCGAA GAUCGUCGG5MUPSUCAAAUCCGGCCCCCGCAACCA 3’ [76]Как можно заметить, на 3’-конце есть триплет CCA, к которому присоединяется аминокислота (метионин, судя по названию), координаты его атомов приведены. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (выходной файл – rna_old.out).
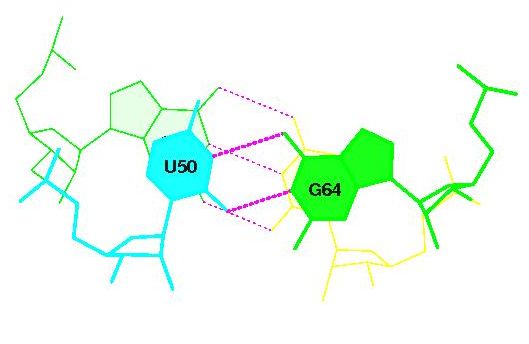
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change) Strand I Strand II Helix 1 (0.017) C:...2_:[..G]G-----C[..C]:..71_:C (0.004) | 2 (0.006) C:...3_:[..C]C-----G[..G]:..70_:C (0.006) | 3 (0.008) C:...4_:[..G]G-----C[..C]:..69_:C (0.005) | 4 (0.004) C:...5_:[..G]G-----C[..C]:..68_:C (0.014) | 5 (0.009) C:...6_:[..G]G-----C[..C]:..67_:C (0.009) | 6 (0.006) C:...7_:[..G]Gx----C[..C]:..66_:C (0.003) | 7 (0.005) C:..49_:[..G]G-----C[..C]:..65_:C (0.006) | 8 (0.009) C:..50_:[..U]U-*---G[..G]:..64_:C (0.007) | 9 (0.006) C:..51_:[..C]C-----G[..G]:..63_:C (0.006) | 10 (0.015) C:..52_:[..G]G-----C[..C]:..62_:C (0.002) | 11 (0.009) C:..53_:[..G]G----xC[..C]:..61_:C (0.010) | 12 (0.013) C:..54_:[5MU]u-**-xA[..A]:..58_:C (0.005) | 13 (0.043) C:..55_:[PSU]Px**+xG[..G]:..18_:C (0.007) x 14 (0.004) C:..22_:[..G]G-----C[..C]:..13_:C (0.009) | 15 (0.005) C:..23_:[..C]C-----G[..G]:..12_:C (0.007) | 16 (0.008) C:..24_:[..U]U-----A[..A]:..11_:C (0.005) | 17 (0.007) C:..25_:[..C]C----xG[..G]:..10_:C (0.015) | 18 (0.010) C:..26_:[..G]G-*---A[..A]:..44_:C (0.012) | 19 (0.006) C:..27_:[..U]U-----A[..A]:..43_:C (0.004) | 20 (0.005) C:..28_:[..C]C-----G[..G]:..42_:C (0.007) | 21 (0.006) C:..29_:[..G]G-----C[..C]:..41_:C (0.004) | 22 (0.008) C:..30_:[..G]G-----C[..C]:..40_:C (0.006) | 23 (0.013) C:..31_:[..G]Gx---xC[..C]:..39_:C (0.005) x 24 (0.007) C:..15_:[..G]Gx**+xC[..C]:..48_:C (0.006) + 25 (0.019) C:..19_:[..G]Gx---xC[..C]:..56_:C (0.004) + 26 (0.005) C:..33_:[..U]Ux*--xA[..A]:..35_:C (0.005) + 27 (0.012) D:...2_:[..G]G-----C[..C]:..71_:D (0.007) | 28 (0.007) D:...3_:[..C]C-----G[..G]:..70_:D (0.011) | 29 (0.014) D:...4_:[..G]G-----C[..C]:..69_:D (0.007) | 30 (0.005) D:...5_:[..G]G-----C[..C]:..68_:D (0.010) | 31 (0.005) D:...6_:[..G]G-----C[..C]:..67_:D (0.005) | 32 (0.006) D:...7_:[..G]Gx----C[..C]:..66_:D (0.005) | 33 (0.007) D:..49_:[..G]G-----C[..C]:..65_:D (0.007) | 34 (0.010) D:..50_:[..U]U-*---G[..G]:..64_:D (0.003) | 35 (0.004) D:..51_:[..C]C-----G[..G]:..63_:D (0.008) | 36 (0.009) D:..52_:[..G]G-----C[..C]:..62_:D (0.007) | 37 (0.012) D:..53_:[..G]G----xC[..C]:..61_:D (0.009) | 38 (0.011) D:..54_:[5MU]u-**-xA[..A]:..58_:D (0.005) | 39 (0.043) D:..55_:[PSU]Px**+xG[..G]:..18_:D (0.006) x 40 (0.007) D:..22_:[..G]G-----C[..C]:..13_:D (0.006) | 41 (0.005) D:..23_:[..C]C-----G[..G]:..12_:D (0.008) | 42 (0.012) D:..24_:[..U]U-----A[..A]:..11_:D (0.009) | 43 (0.013) D:..25_:[..C]C----xG[..G]:..10_:D (0.006) | 44 (0.009) D:..26_:[..G]G-*---A[..A]:..44_:D (0.007) | 45 (0.008) D:..27_:[..U]U-----A[..A]:..43_:D (0.005) | 46 (0.009) D:..28_:[..C]C-----G[..G]:..42_:D (0.008) | 47 (0.005) D:..29_:[..G]G-----C[..C]:..41_:D (0.009) | 48 (0.015) D:..30_:[..G]G-----C[..C]:..40_:D (0.006) | 49 (0.011) D:..31_:[..G]Gx---xC[..C]:..39_:D (0.008) x 50 (0.010) D:..15_:[..G]G-**+xC[..C]:..48_:D (0.006) | 51 (0.008) D:..16_:[..C]Cx**+xA[..A]:..59_:D (0.004) x 52 (0.008) D:..19_:[..G]Gx---xC[..C]:..56_:D (0.004) + 53 (0.010) D:..33_:[..U]U-*---A[..A]:..35_:D (0.009) +В соответствии с полученными данными, а также руководствуясь статьей, определили:Акцепторный стебель состоит из участка 2-6 и комплементарного ему участка 67-71 (красный)
Т-стебель – 49-53 и комплементарного ему участка 61-65(зеленый)
D-стебель – 10-13 и комплементарного ему участка 22-25(синий)
Антикодоновый стебель – 26-30 и комплементарного ему участка 40-44(оранжевый)
В файле rna_old.out показаны взаимодействия не с первого нуклеотида (цитозин), потому что цепь заканчивается аденином, а, значит, нет комплементарного взаимодействия.
Рис.1. Вторичная структура тРНК Скрипт для получения изображения 
restrict none background white select *:c backbone 150 color grey select (2-6, 67-71) and *:c color red select (49-53, 61-65) and *:c color green select (10-13, 22-25) and *:c color blue select (26-30, 40-44) and *:c color orange select (45-48) and *:c color yellow select (14-21) and *:c color pink select (54-60) and *:c color cyan select (31-39) and *:c color magenta select (34-36) and *:c cpk 150 wireframe 100 color brown
Структуру стеблевых дуплексов поддерживают 45 канонических и 8 неканонических пар оснований.
Изображение неканонической пары G-U
Следует отметить:
а) Вероятно, в цепи есть вариабильная петля(45-48 нуклеотиды), окрашено желтым;
б) В Т-петле отсутствует тимидин, но есть 5’-метилуридин-5’-монофосфат и псевдоуридин-5’-монофосфат(54 и 55 нуклеотиды соответственно);
в) В D-петле присутствует дигидроуридин(20 нуклеотид).
Антикодон в антикодоновой петле
Метионин кодируется единственным триплетом ATG, значит, антикодом для него будет CAU, который в антикодоновой петле идет под номерами 34…36 соответственно. На рис.1 антикодон изображен коричневым в шарнирной модели.
- Исследование третичной структуры
Стекинг-взаимодействия
В файле rna_old.out находим информацию о перекрывании азотистых оснований, она имеет вид:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 6 GG/CC 2.54( 1.05) 0.00( 0.00) 0.02( 0.00) 0.06( 0.00) 2.62( 1.05) 7 GU/GC 5.43( 2.45) 0.00( 0.00) 0.00( 0.00) 7.29( 3.77) 12.72( 6.22) 8 UC/GG 0.61( 0.00) 0.00( 0.00) 0.25( 0.00) 1.84( 0.52) 2.70( 0.52)Выбираем из имеющихся взаимодействий пар оснований ту, перекрывание которой наибольшее, например GU/GC .
Далее находим данные о таком взаимодействии в файле stacking.pdb, в данном случае Section #0007 GU/GC.
Вырезаем эту структуру в отдельный файл командойex_str -7 stacking.pdb step7.pdb
Строим изображение с помощью команды
stack2img -cdolt step7.pdb step7.ps
Далее пользуемся программой Ghost и получаем картинку в формате JPEG:
А теперь для сравнения приведем еще одно взаимодействие с одной из самых маленьких ненулевых площадей перекрывания(0, 49): CG/CA
На рисунках заметна разница в площадях перекрывания.
Дополнительные водородные связи
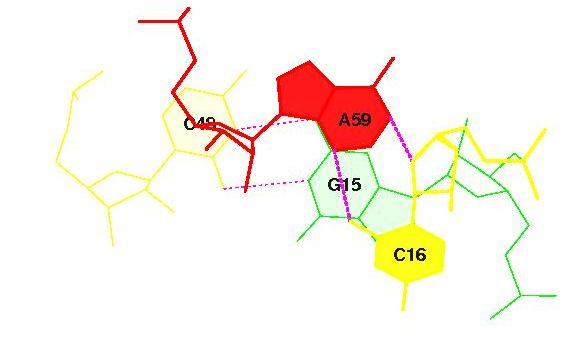
Рассмотрим комплементарные пары, не имеющие отношение к стеблям и стабилизирующие третичную структуру тРНК:
12 (0.013) C:..54_:[5MU]u-**-xA[..A]:..58_:C (0.005) | 13 (0.043) C:..55_:[PSU]Px**+xG[..G]:..18_:C (0.007) x 24 (0.007) C:..15_:[..G]Gx**+xC[..C]:..48_:C (0.006) + 25 (0.019) C:..19_:[..G]Gx---xC[..C]:..56_:C (0.004) + 26 (0.005) C:..33_:[..U]Ux*--xA[..A]:..35_:C (0.005) +
взаимодействия между 54 и 58 - взаимодействия в Т-петле
водородные связи между 55 и 18, 19 и 56 - дополнительные связи между D- и Т-петелями, стабилизирующие третичную структуру тРНК,
при этом пара 55-18 - неканоническая, 19-56 - каноническая.
33-35 - взаимодействия в V-петле.Дополнительное каноническое взаимодействие между D- и Т-петелями
- Предсказание вторичной структуры тРНК Приведите результаты выполнения задания 9: таблицу, рисунок, полученный с помощью mfold, не забудьте привести параметры использованных программ, а также краткие выводы. Желательно описать процедуру подбора параметров.