выравнивание последовательностей
алгоритмы построения - кластерный и топологический
матрицу расстояний(меньшее количество значащих цифр)
Получили филогенетическое дерево, воспользовавшись сервисом TreeTop
Для этого в поле Alignment ввели выравненные с помощью muscle последовательности белков.
В качестве результата TreeTop предоставляет следующие данные:
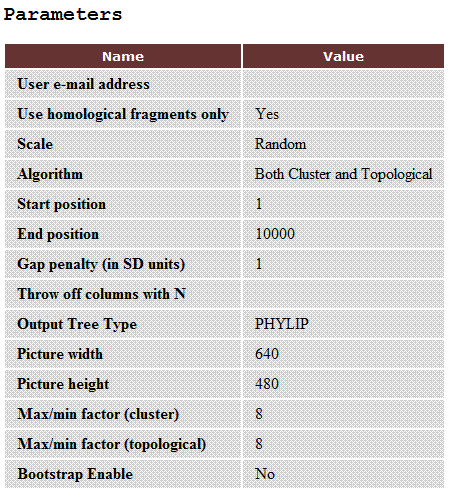
заданные пользователем параметры

выравнивание последовательностей
алгоритмы построения - кластерный и топологический
матрицу расстояний(меньшее количество значащих цифр)
графическое изображение филогенетического дереваDistance Matrix
1 2 3 4 5 6 7 8 1 RPOZ_AGRRK 0.000 0.470 0.837 0.839 0.839 0.827 0.942 0.912 2 RPOZ_RHOS4 0.470 0.000 0.768 0.765 0.765 0.766 0.865 0.844 3 RPOZ_ERWCT 0.837 0.768 0.000 0.103 0.103 0.540 0.698 0.748 4 RPOZ_ECOLI 0.839 0.765 0.103 0.000 0.063 0.545 0.701 0.746 5 RPOZ_SALTY 0.839 0.765 0.103 0.063 0.000 0.545 0.701 0.746 6 RPOZ_PSEAE 0.827 0.766 0.540 0.545 0.545 0.000 0.702 0.663 7 RPOZ_NEIMA 0.942 0.865 0.698 0.701 0.701 0.702 0.000 0.427 8 RPOZ_RALPJ 0.912 0.844 0.748 0.746 0.746 0.663 0.427 0.000
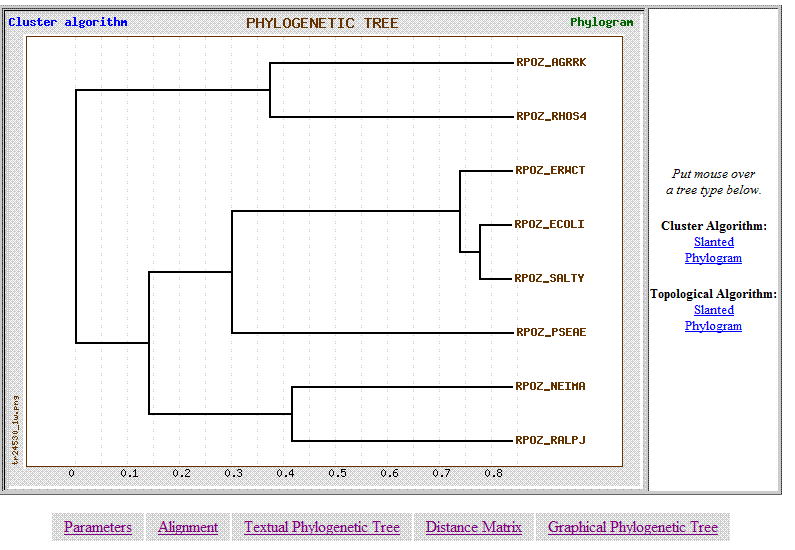
Тип представления изображения можно менять, пользуясь панелью справа
Результат, выданный TreeTop, полностью соответствует
правильному филогенетическому дереву в отличие от результатов, полученных ранее с помощью других программ.
Очевидным преимуществом TreeTop также является яркое графическое изображение.