- Описание доменной структуры белка по данным Pfam
БД Pfam.
Доменная структура белка ASPA_Ecoli по данным Pfam
Cхема из Pfam: 
Пояснения к схеме № Pfam AC Pfam ID Полное название семейства домена Положение в последовательности белка ASPA_Ecoli Клан 1. PF00206 Lyase_1 Лиаза_1, фермент, принадлежащий классу лиаз, для которых субстратом является фумарат. Было показано, что в этих ферментах есть короткий консервативный участок последовательности рядом с метионином, который, возможно, участвует в каталитической активности такого типа ферментов. 13-345 - 2. PF10415 FumaraseC_C Фумарат-гидратаза С катализирует стерео-специфичные взаимные превращения фумарата в L-малат в цикле Кребса. Целый белок формирует тетрамер заметно глобулярной формы. Фумарат-гидратаза С_С состоит из 65 С-концевых аминокислотных остатков , рассматриваемых как домен 3. Ядро молекулы состоит из пучка из 20 альфа-спиралей из пучка из 5 спиралей домена 2. Считают, что этот домен не является частью активного центра или активационного сайта, при этом домен, спиральный по структуре, формирует небольшой пучок 411-465 - - Экспортирован файл в формате msf выравнивание-затравка (seed) для N-концевого домена заданного белка.
PF00206_seed.msf
- Сравнение описания доменной структуры в Pfam с описанием в других БД
В БД InterPro найдено описание всех подписей, интегрированных в InterPro:

На мой взгляд, наиболее отличается от подписей Pfam подпись PROSITE pattern, на рисунке - последняя из интегрированных подписей(желтый цвет).
Его InterPro ID IPR020557
Эта подпись характеризует консервативный участок фермента фумаратлиаза длиной 10 аминокислотных остатков.
Паттерн G-S-x(2)-M-x-{RS}-K-x-N
Такой участок представлен во всех интегрированных подписях из других БД(всего указано 10) и несет мало информации о первичной структуре заданного белка. - Сопоставление доменной структуры с 3D структурой заданного белка
Изображение белка:

PDB ID 1JSW
Белок состоит из 4 цепей, на каждой из которых выделены 2 домена, рассмотренные выше.
Зеленым выделены Lyase_1 на всех цепях, желтым - FumaraseC_C, синим - то, что не входит в эти домены.Рассмотрим отдельно цепь В:
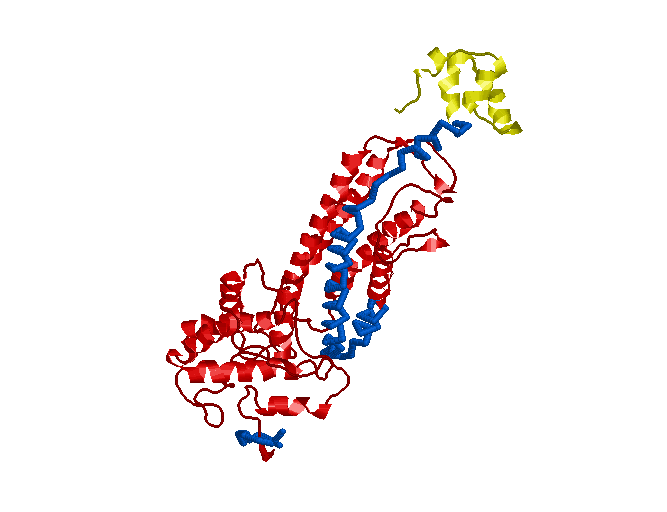
Можно утверждать, что домен FumaraseC_C соответствует структурному домену.
Домен Lyase_1 не полностью совпадает со струтурным доменом, есть участок белка в виде альфа-спирали, который входит в структурный домен, но не является частью домена Lyase_1(выделен синим).В целом, можно даже сказать, что оба домена составляют один структурный домен, но не полностью с ним совпадают - не содержат "синего" участка последовательности.
- Описание: как часто и в каких организмах встречаются домены белка ASPA_Ecoli
Выбрали один домен заданного белка: Lyase_1(PF00206).
Представленность домена PF00206 в организмах разных видов
ТаксонКоличество белков с доменом PF00206Эукариоты Зеленые растения 90 Грибы 211 Животные 189 Остальные эукариоты 110 Бактерии 5220 Археи 168 - Определить, в скольких разных белках Escherichia coli K12 встречаются домены, представленные в заданном белке
Представленность изучаемых доменов в белках Escherichia coli (strain K12)
№ PFAM ID Количество белков в Escherichia coli (strain K12) 1. PF00206 4 2 PF10415 2
Все найденные белки принадлежат классу лиазы, отличаются С-терминальным доменом, кроме белка PUR8_ECOLI, первый домен в нем не идентичен PF00206.С точно такой же доменной организацией найден единственный белок в Escherichia coli (strain K12), но также найден белок FUMC_ECOLI, доменная организация которого отличается неполной идентичностью домена PF10415:

Таким образом, домен PF00206 не является характерным для единственного белка, домен PF10415 встречается в одном белке. - Разные доменные перестройки
Домен PF00206
а) сочетание с другими доменами, причем бывает как на N-, так и на C-конце последовательности.
ASPA_ECOLI:
 C0S5W7_PARBP:
C0S5W7_PARBP:
б) дупликация + перекрытие
ASPA_ECOLI:
 Q0DS22_ORYSJ:
Q0DS22_ORYSJ:
в) разрыв в последовательности домена
ASPA_ECOLI:
 Q4UB18_THEAN :
Q4UB18_THEAN :
Домен PF10415
а) может не сочетаться с другими доменами
ASPA_ECOLI:
 C2Y934_BACCE :
C2Y934_BACCE :
б) может сочетаться с другими доменами, притом перекрываясь с ними
ASPA_ECOLI:
 A5P9M3_9SPHN :
A5P9M3_9SPHN :
© Ксения Лежнина 2008-2010
Для заполнения таблицы были выбраны данные, содержащие число уникальных последовательностей, на которых был найден изучаемый домен.
Полученные данные показывают, что существенно большее распространение этот домен получил среди бактерий.