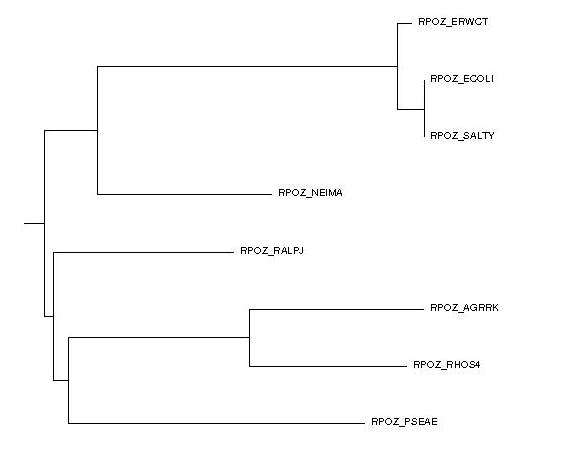
Укоренение произошло в ветвь {SALTY, ECOLI, ERWCT, NEIMA} против {AGRRK, RHOS4, PSEAE, RALPJ}
Общими с правильным деревом являются ветви:
Укоренять имеет смысл то дерево, в котором указаны длины ветвей и нет корня,
поэтому для данного задания подходит дерево,
полученное программой fneighbor методом neighbor-joining.
Для этого воспользуемся программой retreeпакета PHYLIP.
На вход подаем файл со скобочной формулой, выданный программой fneighbor.
Нужное действие - укоренение - получаем введением в поле буквы M("Midpoint root the tree").
Результат выдачи программы - файл outtree.
Изображение полученного укорененного дерева:

Укоренение произошло в ветвь {SALTY, ECOLI, ERWCT, NEIMA} против {AGRRK, RHOS4, PSEAE, RALPJ}
Общими с правильным деревом являются ветви:
Различия деревьев заключаются в представлении эволюции NEIMA, PSEAE, RALPJ.
Изображение полученного дерева c листом BACSU:

Отредактировав скобочную формулу, получили дерево без аутгруппы:
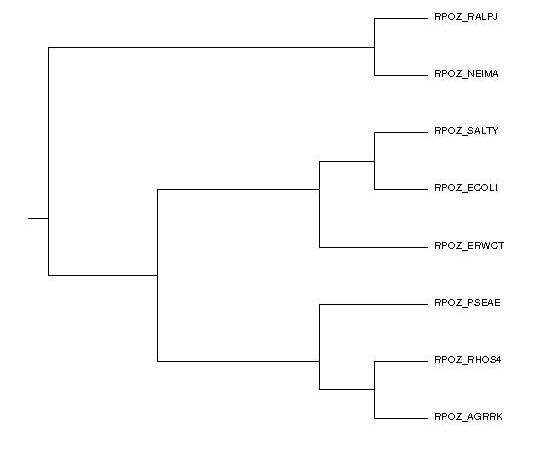
Укоренение - ветвь {NEIMA, RALPJ} против {SALTY, ECOLI, ERWCT, AGRRK, RHOS4, PSEAE}
Различие с правильным деревом - эволюция PSEAE.
+------RPOZ AGRRK
+--------100.0-|
| +------RPOZ RHOS4
+-73.0-|
| | +-------------RPOZ ERWCT
| +-100.0-|
+-100.0-| | +------RPOZ SALTY
| | +-90.6-|
| | +------RPOZ ECOLI
+------| |
| | +---------------------------RPOZ PSEAE
| |
| +----------------------------------RPOZ NEIMA
|
+-----------------------------------------RPOZ RALPJ
неукорененное дерево
В данном случае никаких изменений по сравнению с деревом, полученным fprotpars, не произошло.
Как видно, все представленные ветви поддерживаются большинством, но все же это дерево не совпадает с правильным.
Ветвь с поддержкой ветвей равной 73.0 не существует в правильном дереве.
Ветви, не получившие большинства:
1. RPOZ RALPJ 2. RPOZ NEIMA 3. RPOZ PSEAE 4. RPOZ SALTY 5. RPOZ ECOLI 6. RPOZ ERWCT 7. RPOZ RHOS4 8. RPOZ AGRRK Set (species in order) How many times out of 100.00 ..*...** 17.18 (1) ..****.. 9.83 (2) ...*.*.. 4.90 (3) ....**.. 4.45 (4)Верная ветвь (2): {SALTY, ECOLI, ERWCT, PSEAE} против {AGRRK, RHOS4, NEIMA, RALPJ}.
Несмотря на применение других алгоритмов к данной выборке организмов, ни одно построение полностью не совпало с правильным деревом.