Комплексы ДНК-белок
В этом практикуме необходимо было изучить и предсказать вторичную структуру РНК, а также исследовать ДНК-белковые взаимодействия.
Предсказание вторичной структуры заданной тРНК
В этой части практикума мы использовали несколько различных алгоритмов предсказания вторичной структуры РНК, а затем сравнивали их с реальной вторичной структурой РНК и смотрели, насколько предсказания сходятся с реальностью. Сначала мы воспользовались программой einverted - хорошего результата программа не дала даже при нулевом пороге Expected threshold, выдала всего один участок, приведённый ниже:
yablinkubovich@kodomo:~/public_html/term3/pr3$ cat emboss_001.inv
EMBOSS_001: Score 15: 5/5 (100%) matches, 0 gaps
3 cccca 7
|||||
69 ggggt 65
Затем вторичная структура РНК была предсказана по алгоритму Зукера (картинка полученного предсказания ниже, выдача программы - тоже):
yablinkubovich@kodomo:~/public_html/term3/pr3$ cat 1N78.fasta | RNAfold --MEA
>1N78_1|Chains
GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUUCCCCCUGGGGUCACCA
(((((((.(((((........)))))(((((.......)))))....(((((.......)))))))))))).... (-35.80)
(((((((.(((((........)))))(((((.......)))))....(((((.......)))))))))))).... [-36.21]
(((((((.(((((........)))))(((((.......)))))....(((((.......)))))))))))).... {-35.80 d=1.57}
(((((((.(((((........)))))(((((.......)))))....(((((.......)))))))))))).... {-35.80 MEA=72.59}
frequency of mfe structure in ensemble 0.511871; ensemble diversity 2.92
Исходя из полученных данных, мы сделали таблицу, сравнивающую результаты анализа с помощью программ find_pair, einverted и RNAFold.
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' -1-7- 3' 5'-66-72-3' Всего 7 пар |
предсказано 5 пар из 7 реальных | предсказано 7 пар из 7 реальных |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары |
предсказано 0 пар из 4 реальных | предсказано 4 пары из 4 реальных |
| T-стебель | 5'-48-52-3' 5'-60-64-3' Всего 5 пар |
предсказано 0 пар из 5 реальных | предсказано 5 пар из 5 реальных |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' Всего 7 пар (из них 2 неканонических взаимодействия, я решил включить их в описание стебля) |
предсказано 0 пар из 7 реальных | предсказано 5 пар из 7 реальных |
| Общее число канонических пар нуклеотидов | 23 | 5 | 21 |
По таблице видно, что алгоритм Зукера намного точнее предсказывает структуру РНК, чем программа einverted, однако он не учитывает неканонические взаимодействия азотистых оснований, что приводит к небольшим отклонениям модели от реальной структуры.
Поиск ДНК-белковых контактов в заданной структуре
Для нахождения контактов ДНК с белком был написан скрипт, который можно посмотреть здесь.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 12 | 21 | 33 |
| остатками фосфорной кислоты | 21 | 90 | 111 |
| остатками азотистых оснований со стороны большой бороздки | 1 | 16 | 17 |
| остатками азотистых оснований со стороны малой бороздки | 9 | 25 | 34 |
Как можно видеть, неполярных контактов больше, чем полярных во всех категориях. Больше всего контактов атомов углерода и серы с остатками фосфорной кислоты (их 90),
очень мало контактов с остатками азотистых оснований со стороны большой бороздки, что может быть объяснено труднодоступностью данного места (или я не знаю).
Файл pdb был переведён в старый формат, чтобы на его основе сделать карту ДНК-белковых взаимодействий с помощью утилиты nucplot. Результаты можно видеть на картинках,
представленных ниже.
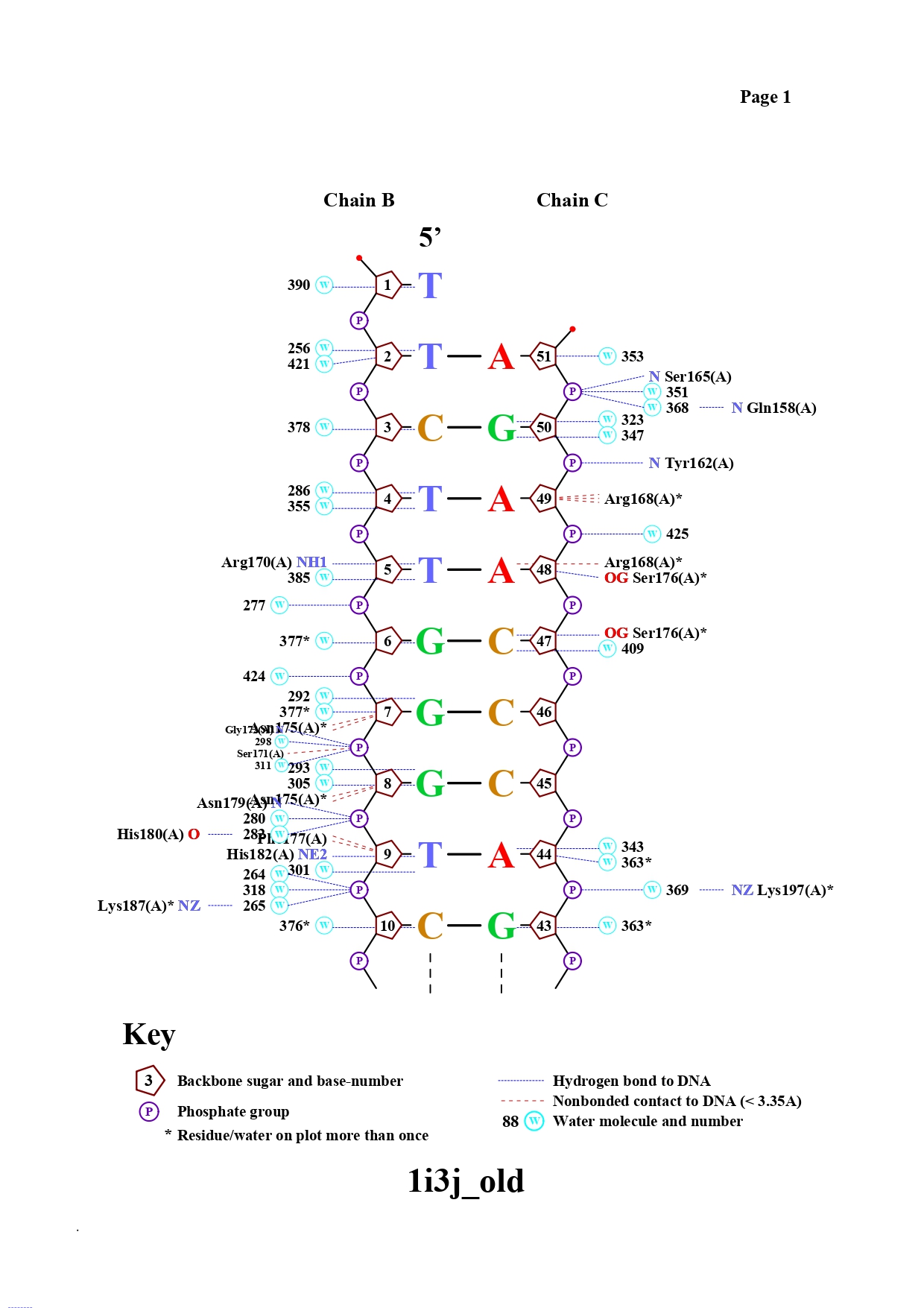
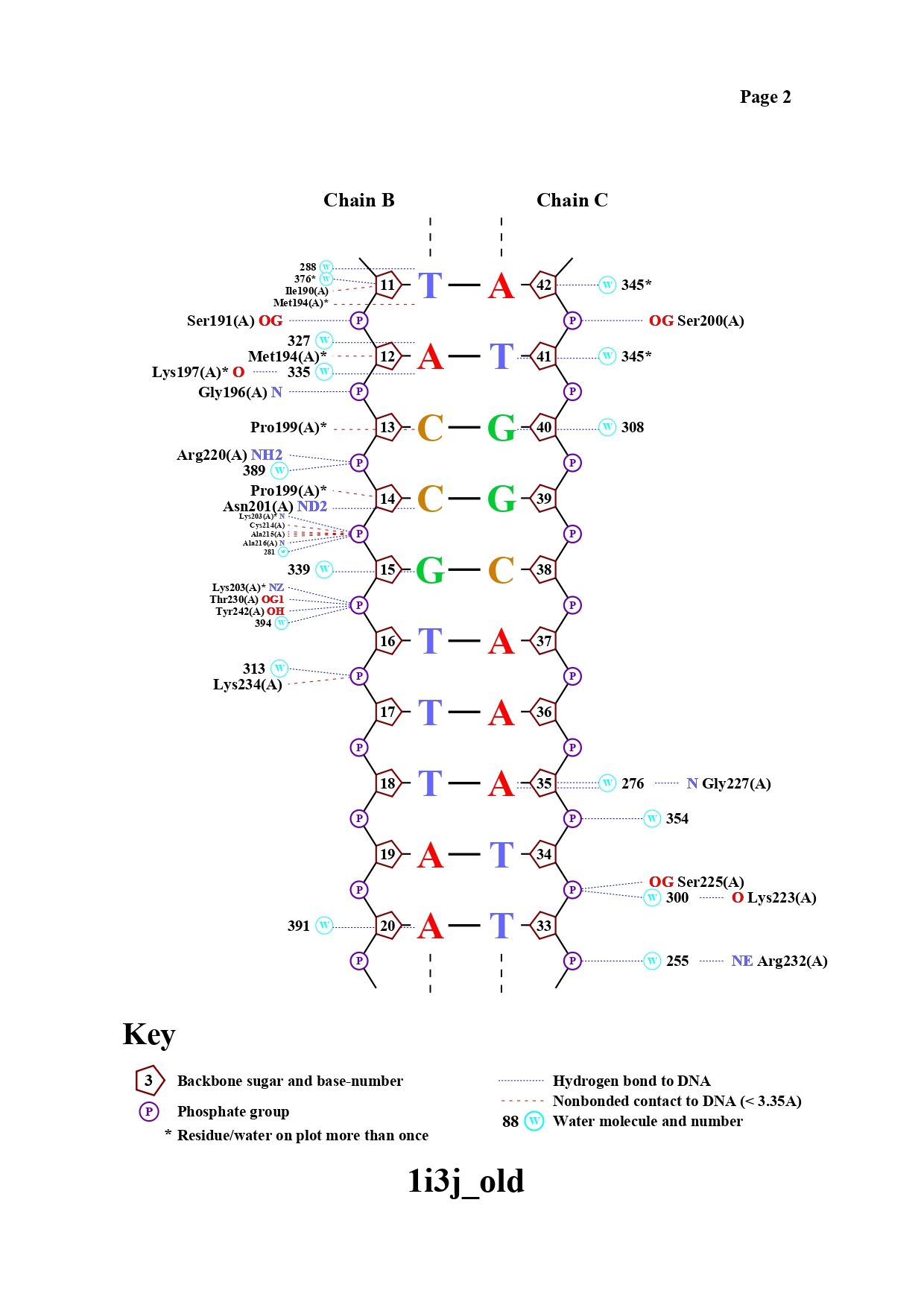
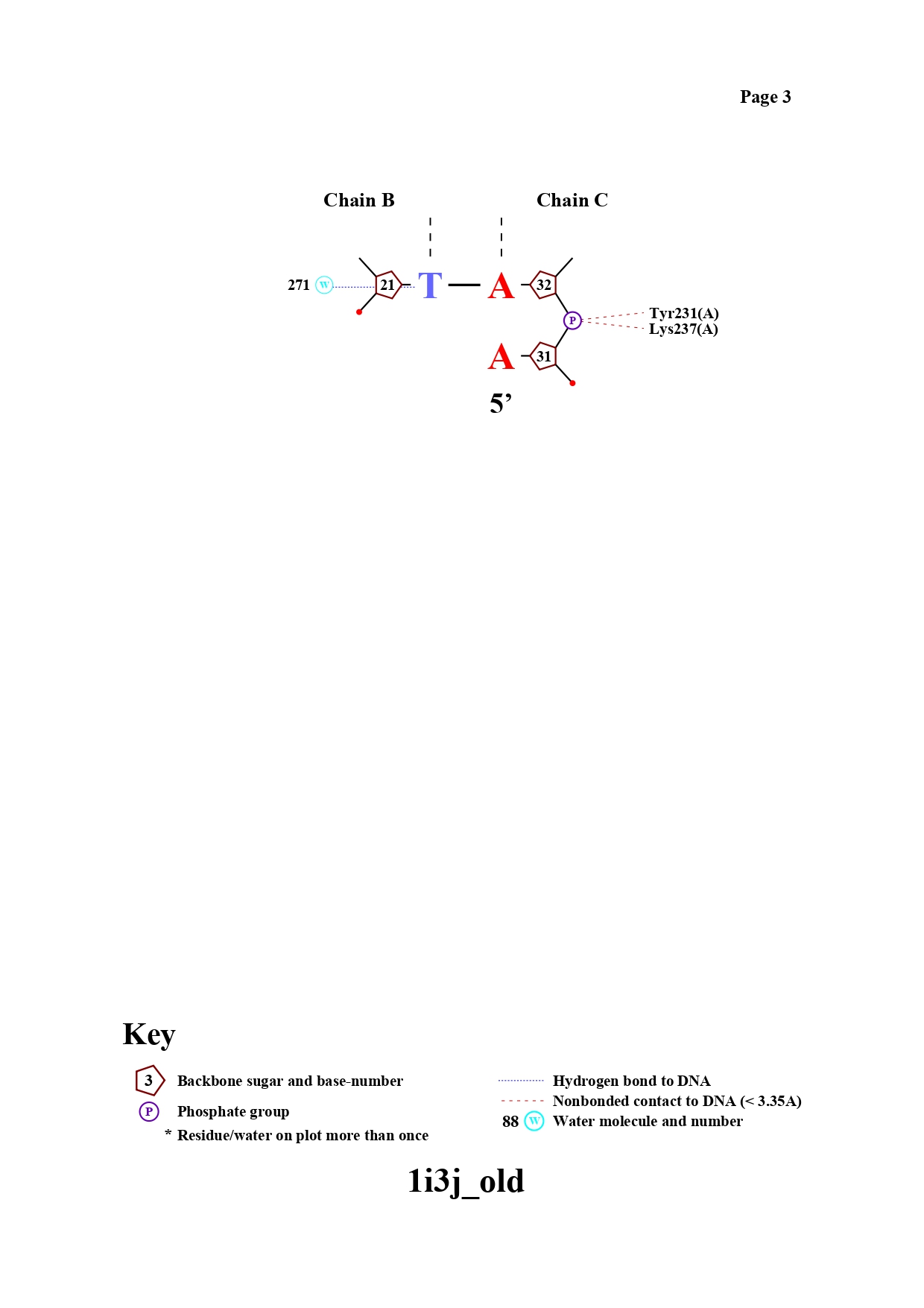
Больше всего контактов образуют серин 176 (с цитозином 47 и с остатком дезоксирибозы 48 нуклеотида в цепи С ДНК), пролин 199 (nonbonded contact с цитозином 13 и остатками дезоксирибозы следующего нуклеотида), а также аспарагин 175 ( по 2 странных взаимодействия с фосфатами 7 и 8 нуклеотида в цепи B ДНК).
Мне кажется, что важность аминокислотного остатка и количество его взаимодействий с ДНК должны каким-то образом коррелировать, поэтому выберу серин 175 и аспарагин 176 в качестве важных для опознавания последовательности остатков (связывают 2 последовательных нуклеотида и взаимодействуют прямо с азотистым основанием).