Сигналы и мотивы
Полученный в прошлом практикуме сигнал с наименьшим p-value(порядка 10e-4) был передан в программу FIMO.Полученные результаты можно видеть в Таблице 1, представленной ниже. Поиск
мотивов вёлся всё в том же коронавирусе летучей мыши HKU15. Среди находок есть находки в конце полипротеина, это можно объяснить перекрещиванием генов, из-за чего upstream область гена
S-белка расположена в конце гена полипротеина 1ab. Также есть ещё одна находка внутри полипротеина(7423-7433), это странно, как это объяснить, я, честно говоря, не знаю. В принципе, низкий
p-value для мотивов, найденных в upstream-областях поздних генов, может говорить о том, что мотив может быть сигналом.
| Motif ID | Alt ID | Sequence Name | Strand | Start | End | p-value | q-value | Matched Sequence |
|---|---|---|---|---|---|---|---|---|
| 1 | NC_018871.1 | + | 75 | 85 | 3.7e-07 | 0.0134 | AACTAAACGAA | |
| 1 | NC_018871.1 | + | 20629 | 20639 | 8.2e-07 | 0.0134 | AACTAAACAAA | |
| 1 | NC_018871.1 | + | 26297 | 26307 | 8.2e-07 | 0.0134 | AACTAAACAAA | |
| 1 | NC_018871.1 | + | 25349 | 25359 | 1.27e-06 | 0.0155 | AACTAAACTAA | |
| 1 | NC_018871.1 | + | 4148 | 4158 | 7.25e-06 | 0.057 | AACTAAACAAG | |
| 1 | NC_018871.1 | + | 25597 | 25607 | 1.3e-05 | 0.0792 | GTCTAAACGAA | |
| 1 | NC_018871.1 | + | 26292 | 26302 | 2.37e-05 | 0.129 | GTCTAAACTAA | |
| 1 | NC_018871.1 | + | 27519 | 27529 | 3.42e-05 | 0.167 | AACTAAACAGG | |
| 1 | NC_018871.1 | + | 7423 | 7433 | 4.4e-05 | 0.196 | TTCTTAACGAA | |
| 1 | NC_018871.1 | + | 21682 | 21692 | 7.04e-05 | 0.265 | AACGTAACAAG | |
| 1 | NC_018871.1 | + | 24656 | 24666 | 7.04e-05 | 0.265 | AACGTTACGAA |
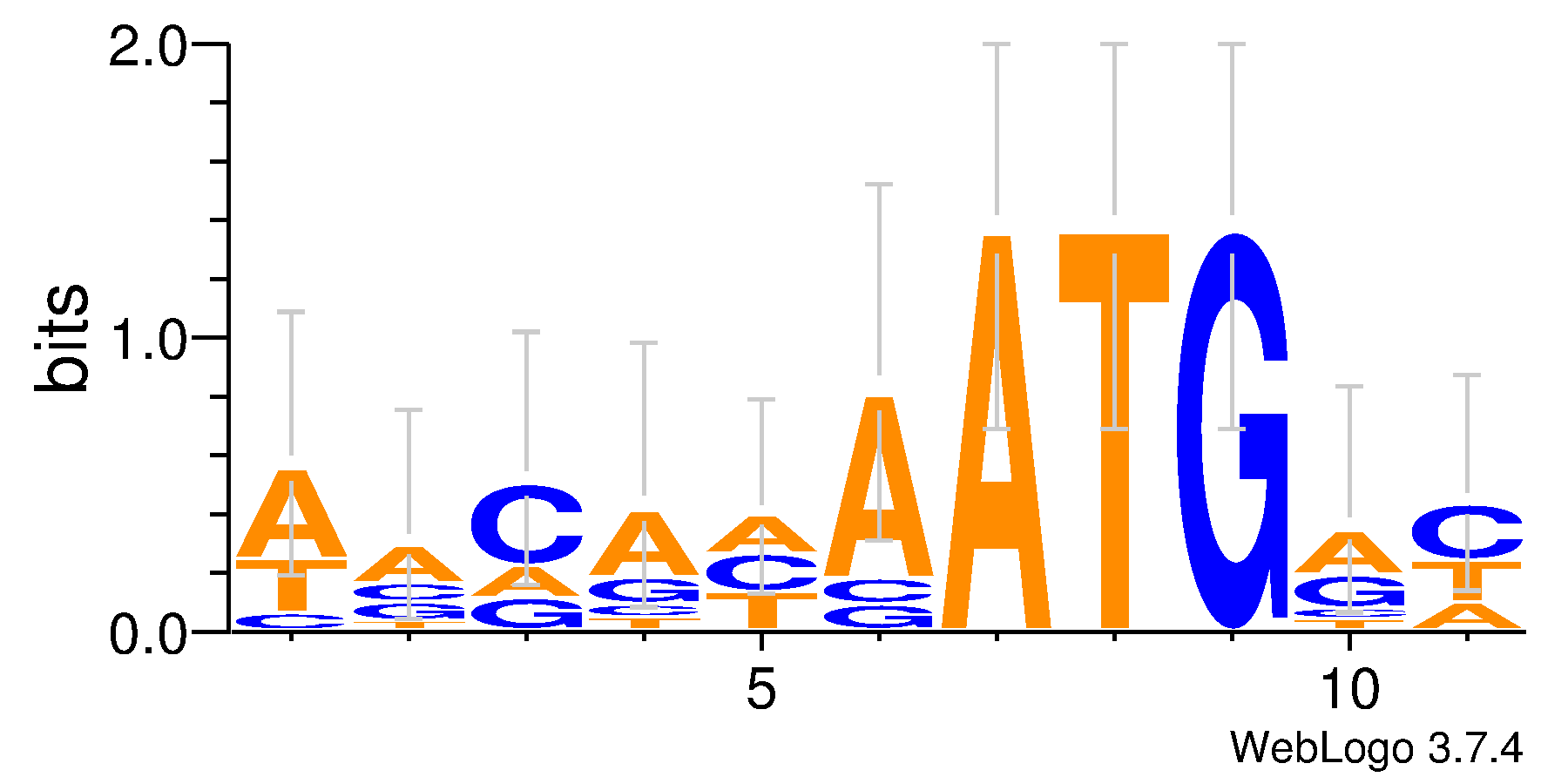
Для построения LOGO последовательности Козак в геноме коронавируса были взяты участки последовательности вокруг поздних генов (и полипротеина тоже) с координатами -6...+4. На Рисунке
1 представлен результат данных манипуляций. Как видно, каких-либо ярко выраженных консервативных последовательностей вокруг старт-кодона не наблюдается. Также было замечено, что
последовательность Козак весьма не похожа на последовательность Козак человека (5' - GCCRCCAUGG - 3' у млекопитающих).
С помощью программы FIMO был проведен поиск мотивов по геному коронавируса кролика HKU14 (NC_017083.1). Результаты поиска можно видеть на Рисунке 2 - их стало намного меньше, однако,
судя по координатам поздних генов, эти последовательности находятся внутри генов, а не перед ними, как можно было бы ожидать. Из полученных данных можно сделать вывод о специфичности данного
мотива для конкретного штамма коронавируса.
В итоге хочу сказать, что мы всё-таки нашли Core Sequence - по моему мнению, этой последовательностью является полученная в предыдущем практикуме последовательность 5'-CTAAAC-3'.