Работа в BLAST.
Поиск гомологов белка Q9S3U8 в SwissProt
Параметры запуска Blast
- Query subrange - С помощью этого параметра можно применить BLAST не ко всей последовательности, а только к ее участку. По умолчанию
- Database - Выбор базы данных. UniProtKB/Swiss-Prot(swissprot)
- Organism - Ограничить поиск только белками некоторых видов (или, наоборот, исключить некоторые виды - exclude). Кроме того, можно исключить белки с теми или иными идентификаторами RefSeq. По умолчанию
- Algorithm - Выбор алгоритма; я использовал blastp (по умолчанию). Другие варианты: PSI-BLAST (Position-Specific Iterative BLAST), PHI-BLAST (Pattern Hit Initiated BLAST), DELTA-BLAST (Domain Enhanced Lookup Time Accelerated BLAST). Так или иначе, все эти алгоритмы улучшают поиск гомологов, так как учитывают важность отдельных консервативных блоков. По умолчанию blastp
- Max target sequences - определяет максимальное количество искомых последовательностей. 20000
- Short queries(Automatically adjust parameters for short input sequences) - в случае поиска коротких последовательностей имеет смысл использовать другие параметры по умолчанию, нежели в случае длинных. Эта кнопка позволяет использовать эти параметры. По умолчанию
- Expect threshold - порог по E-value. Последовательности с большим в E-value не показываются. По умолчанию 10
- Word size - размер слова для поиска. Алгоритм бласта работает так, что индексирует слова определенной длинны. Чем меньше длинна слова, тем больше последовательностей будет сравниваться. По умолчанию 6
- Matrix - выбор матрицы аминокислотных замен. По умолчанию BLOSUM62
- Gap Costs - Штрафы за наличие и продолжение инделя. По умолчанию Existence: 11 Extension: 1
- Compositional adjustments - Корректировка матрицы замен для борьбы с участками малой сложности, в которых определенные аминокислоты повторяются много раз. По умолчанию
- Filter(Low complexity regions) - области малой сложности, в которых подсчёт гомологичности работает некорректно, например, в GR-богатых участках, забиваются "иксами". По умолчанию
- Mask for lookup table only - маскировка областей малой сложности "иксами" идёт только на шаге поиска возможно гомологичных последовательностей(см. word size), в выводы иксов нет. По умолчанию
- Mask Lower Case - вместо X замаскированные участки отмечаются нижним регистром. По умолчанию
Выдача BLAST доступна в Таблице.
Для построения множественного выравнивания были выбраны 10 последовательностей из различных организмов и с различными Similarity и E-value. Они были выровнены при помощи программы MuscleWS, встроенной в Jalview. Затем были удалены белки имеющие малую длину, а также белки с большим количествои инделей. Оставшиеся белки были выровнены для надежности.
Можно скачать Выравнивание

Был найден учаток с высокой долей гомологии. Он и представлен на приклепленной картинке.
Карта сходства белков M2R9A4_CERS8 и A0A1R4HTG4_9MICO
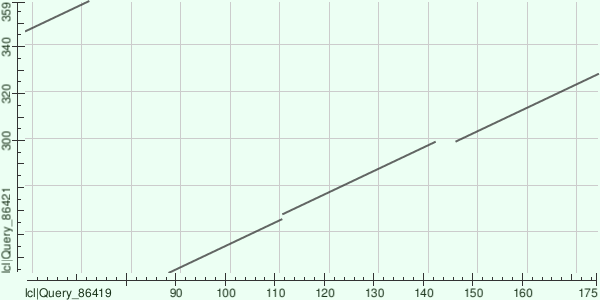
По карте видно, что у последовательности M2R9A4 произошла транслокация начального фрагмента в конец последовательности, а также делеция следовавшего за ним фрагмента.
Игры с BLAST
В качестве последовательности была взята: "Of Course I Still Love You". Если задать все параметры стандартными (кроме базы данных, которая по задания должна быть Swissprot, и количества выдаваемых последовательностей в 20000), то BLAST найдет все 20000 последовательностей. При этом E-value будет в диапозоне от 5.1 до 48576. При этом процент идентичности был высоким (около 70%). Но после снятия галочки Automatically adjust parameters for short input sequences все они исчезли из выдачи. После смены длины слова на 2, смены матрицы на BLOSUM90 появилась одна выдача с E-value 6.4. После смены алгоритма на PSI-BLAST, смены treshold на 1000 нашлось 48 последовательностей. С E-value от 6.4 до 984.
