Практикум 9. PFAM
Для работы были выбраны два белковых домена из бета-галактозидазы E.coli.
| ID | PF02837 | PF00703 |
|---|---|---|
| AC | Glyco_hydro_2_N | Glyco_hydro_2 |
| Название | Гликозил гидролаза семейства 2, сахар-связывающий домен | Гликозил гидролаза семейства 2 |
| Число находок в Uniprot | 47,246 | 48,004 |
 Ссылка с таблицей
Ссылка с таблицей
Построение HMM - профиля по выбранной архитектуре и проверка его работы
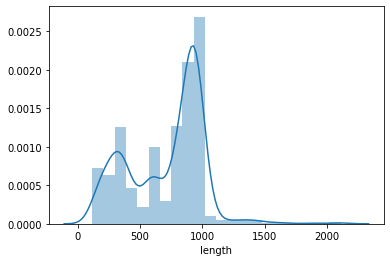
На рисунке 1 изображено распределение длин белков с выбранной доменной архитектурой. Распределение бимодальное (кроме основного пика на 900-100 а.о., есть второй пик на 300-400 а.о.). Это, возможно, отражает наличие двух подгрупп среди данных белков.
При помощи скрипта на питоне были выбраны около 300 белков с заданой архитектурой и длиной в районе пика, составлен запрос в uniprot. Последовательности белков были скачаны и выравнены в JalView программой Muscle. Из итогового выравнивания я удалил белки с крупными инсерциями, перевыравнил и повторил эту процедуру несколько раз. Затем по этим выравниваниям составлен HMM-профиль.

Команды для построения HMM профиля
hmm2build arch.hmm HMM_4.fasta
hmm2calibrate arch.hmm
Команда для проверки HMM профиля:
hmm2search -E 0.01 arch.hmm database.fasta.gz | cat > out.txt
Результат поиска был распаршен частично вручную, частично скриптом на питоне и экспортирован как EXCEL-документ. В документе были проведены все необходимые рассчеты, обработка в Google Sheets. Также была построена ROC-кривая. В результате специфичность профиля при E-value < 0.01 составила 0.958, а чувствительность всего 0.808.
