Практикум 11
Выбор семейства доменов из Pfam
Я скачал список всех семейств из Pfam себе на компьютер и установил фильтры по полям seed (30 <= seed <= 400) и full (full <= 400). Из получившегося списка я выбрал семейство белков PF00998.
Описание семейства белков
Name: Viral RNA dependent RNA polymerase
AC: PF00998
ID: RdRP_3
Seed: 32
Full: 218
Structures: 225
Species: 210
Domain Architectures: 162
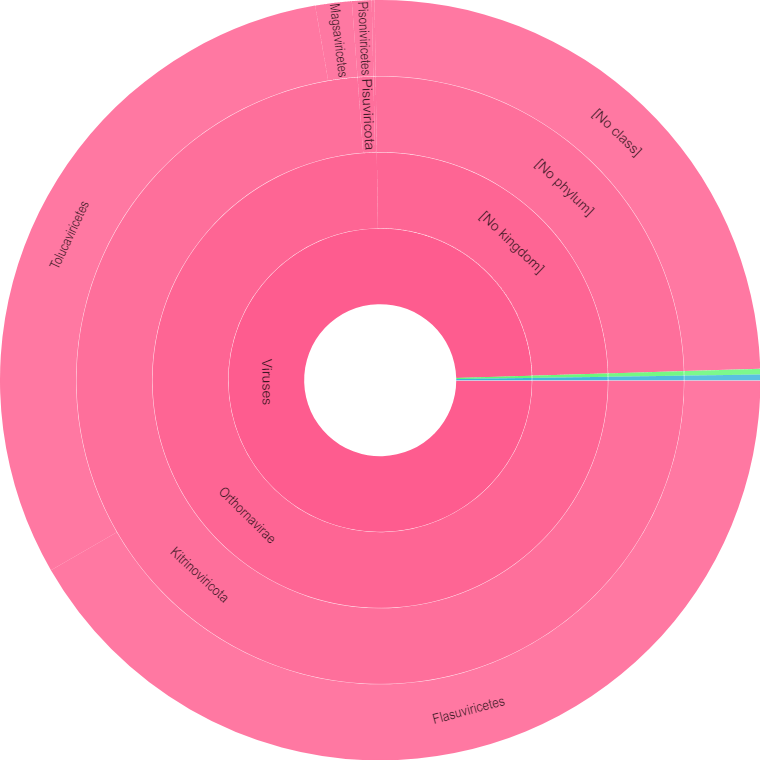
Как видно из диаграммы ниже, большинство обладателей данного семейства белков - вирусы. Большая часть из них относится к Orthornavirae, у остальных царственная принадлежность не установлена.
Словесное описание из Pfam
Это семейство включает в себя ферменты вирусных РНК-зависимых РНК-полимераз из вируса гепатита С и различных растительных вирусов.
Белки этого семейства - это РНК-зависимые РНК-полимеразы. Это ферменты, катализирующие репликацию РНК на основе РНК-шаблона. То есть синтез комплементарной РНК-цепи идёт по заданному РНК-шаблону. Это ключевое отличие от типичных РНК-полимераз, зависимых от ДНК, которые используются практически всеми организмами для транскрипции РНК на основе ДНК-шаблона.
Описание выравниваний seed с точки зрения гомологичности всех последовательностей
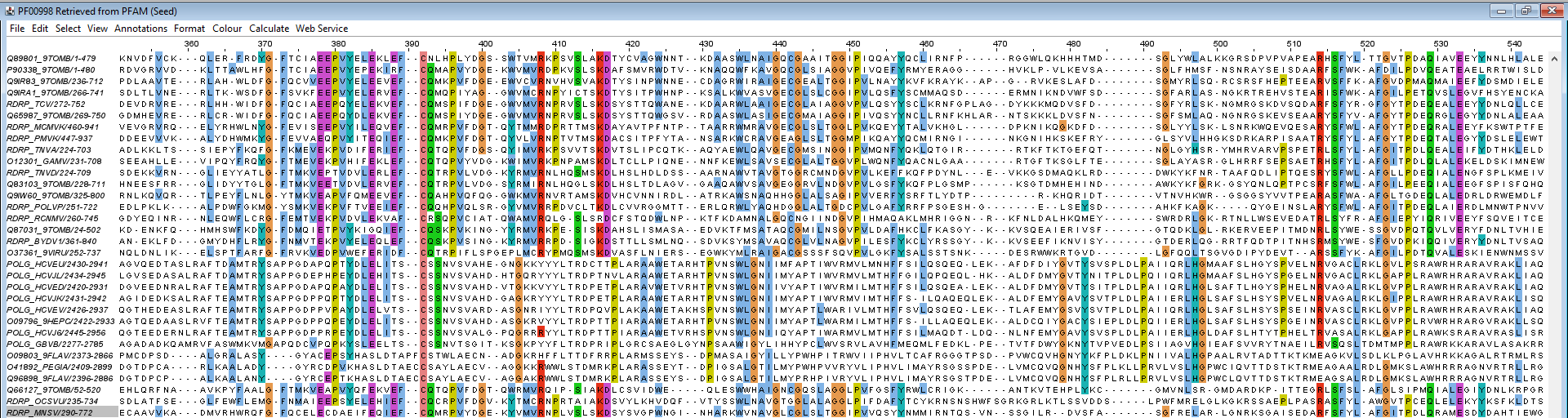
Максимально достоверные (консервативные) блоки
Единичные консервативные нуклеотиды встречаются на позициях 48, 240, 303, 306, 314, 392. Блоков, консервативных на 100 процентов, всего 2: с 244-245 и с 341-343

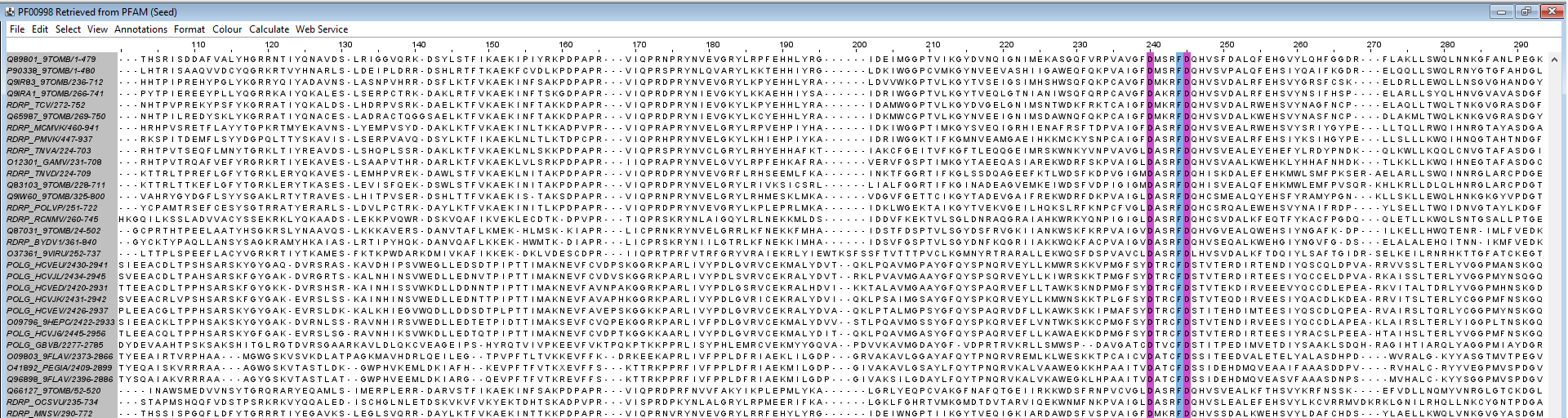
Максимально достоверные (консервативные) блоки в подгруппе белков
Максимально достоверный блок в выбранной подгруппе находися на позициях 48-56 и имеет длину 9 кодонов

Проект JalView с покраской 100% консервативных участков подгруппы белков
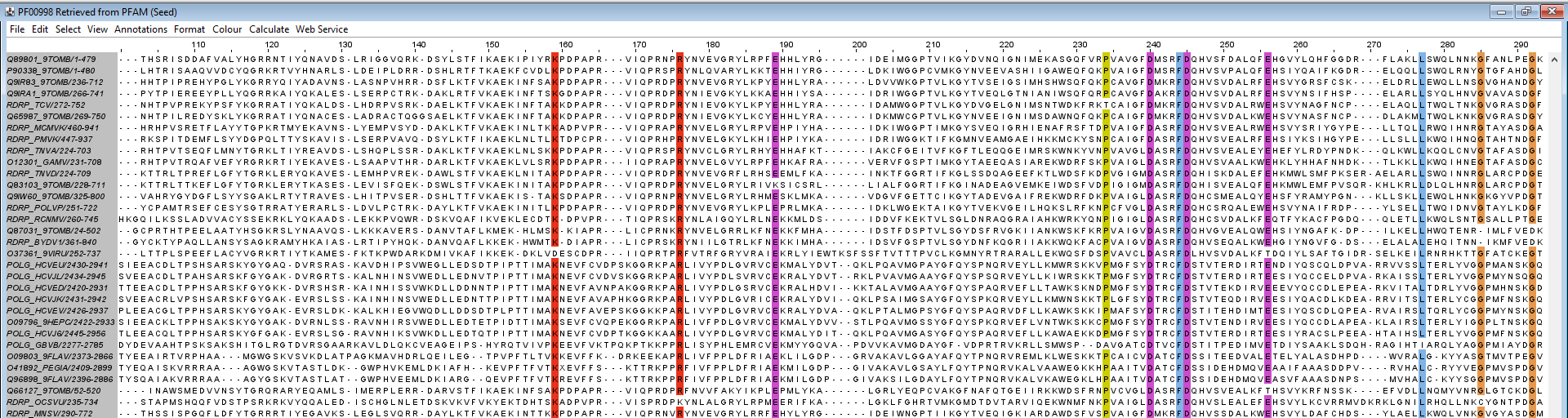
Максимально недостоверные блоки
Максимально недостоверный блок, вероятнее всего не отражающий реальный ход жволюции, находится на позициях 461-476
Выводы о гомологии последовательностей изходя из их выравниваний
Конечный результат показывает гомологию последовательностей достаточно достоверно. На основной части последовательностей можно наблюдать небольшие, но достоверные блоки (как показано на изображениях ниже). Маленькие участки с блоками также могут быть результатом ошибок в выравнивании или отсутствием важной функции в соответствующем участке, что приводит к накоплению мутаций. Однако, общая картина выравнивания (особенно с учетом 50% раскраски) показывает, что все они можно разделить на две группы: с 1 по 18 + с 29 по 32 и с 19 по 28. Внутри этих групп гомология последовательностей достаточно точно отражается.
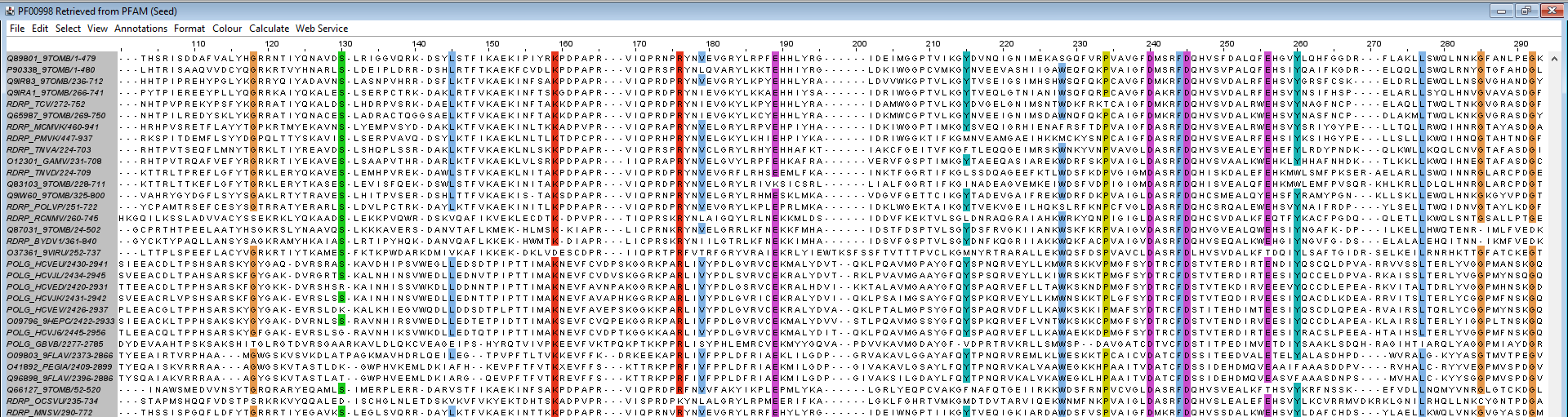
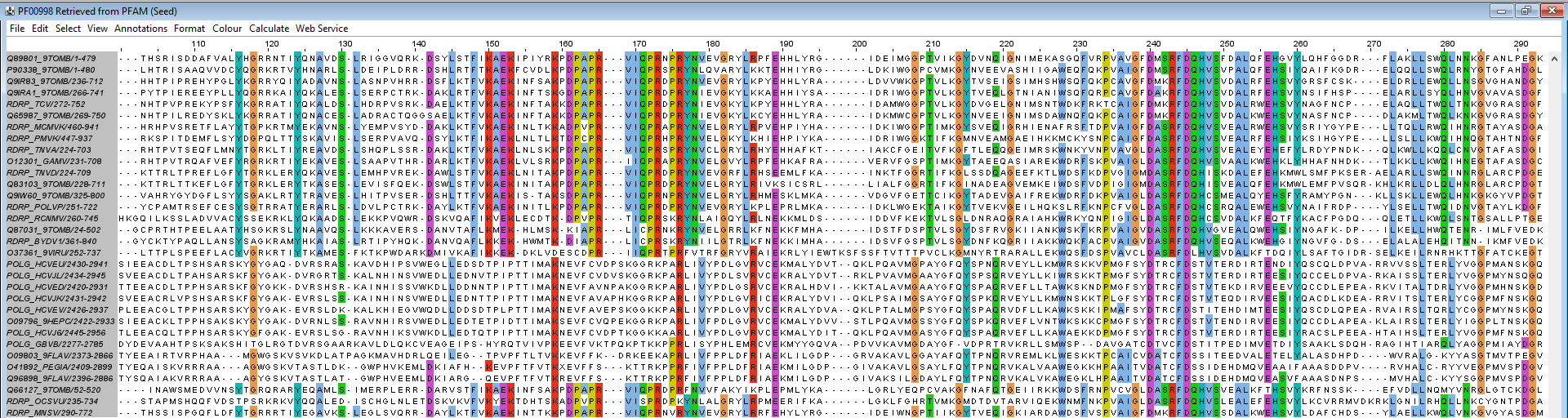
Соответствующих проекты JalVeiw: