Практикум 12
Сравнение выравниваний одних и тех же последовательностей разными программами
Как и в предыдущем практикуме, здесь я использую семейство белков PF00998. Для сравнения множественных выравниваний использовались нижние 5 белков.
Я использовал программу Лизы Плешко для сравнения выравниваний


Сравнение выравниваний
Сравнение дало следующие результаты (их можно посмотреть в файле):
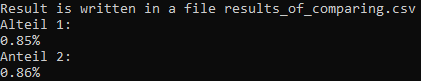
При помощи скрипта на питоне я получил результаты сравнения выравниваний. Выравнивания совпадают на позициях 1-104 и 105-442. Таким образом, выравнивания разбиваются на 2 гомологичных блока, а несколько триплетов между ними оказываются негомологичными. Эти результаты говорят о том, что разные алгоритмы гомологичные последовательности выравнивают одинаково.
Выравнивание по 3D структуре
С помощью UniProt я выбрал 3 белка
(2FM2,
2D3U,
3M5L)
из рассматриваемого семейства
со схожей доменной архитектурой и имеющих расшифрованную 3D структуру.
Я получил 3D выравнивания при помощи PDB,
которые затем перенёс проект в JalView
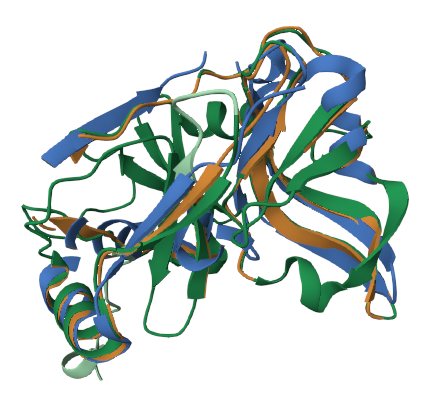
Предположительно, выравнивание структур белков часто является более достоверным способом определения гомологии, поскольку последовательность белка отражается в его пространственной структуре, которая определяет его свойства. Следовательно, для сохранения свойств и прохождения естественного отбора, белок должен сохранять наиболее значимые элементы структуры. Для этого необходимо поддерживать правильное расположение аминокислот или заменять их аминокислотами с схожими свойствами.
Описание работы программы MAFFT
MAFFT (Multiple Alignment using Fast Fourier Transform) - это программа для множественного выравнивания последовательностей. Алгоритм работы MAFFT можно описать следующим образом:
Входные данные: Программа принимает набор последовательностей в формате FASTA (или других поддерживаемых форматах)
Выбор метода выравнивания: MAFFT предлагает несколько методов выравнивания, таких как L-INS-i, FFT-NS-2, или G-INS-i
Построение графа: MAFFT строит граф на основе входных последовательностей, где каждая вершина представляет собой позицию в выравнивании, а ребра соединяют вершины, соответствующие одному и тому же фрагменту последовательности
Выравнивание: MAFFT использует FFT для вычисления автокорреляционной функции последовательностей ДНК или белка. Это позволяет искать оптимальные выравнивания между последовательностями. Алгоритм строится на предположении о том, что хорошие выравнивания имеют пики в своей автокорреляционной функции, и поэтому использует FFT для быстрого вычисления этой функции и поиска оптимальных выравниваний.
Итерации: MAFFT выполняет несколько итераций, в каждой из которых улучшается выравнивание.
Выходные данные: В результате работы MAFFT создает файл с выравниванием последовательностей в выбранном формате (например, FASTA)
Summary: В целом, алгоритм MAFFT основывается на принципе итеративного выравнивания и стремится достичь наилучшего выравнивания последовательностей, учитывая выбранный метод и оптимизируемую метрику