Практикум 8
Для работы используется сборка GRCm39 организма Mus MusculusУпражнение 1
Я выбрал фрагмент с координатами 4560507..4568271 длины 7765. Последовательность этого фрагмента можно скачать тут
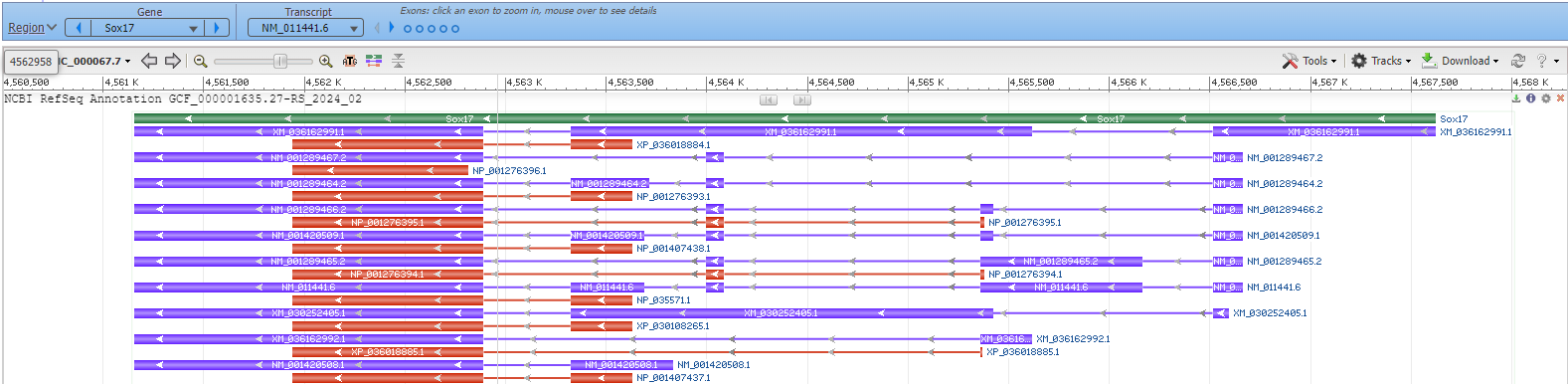
На рисунке ниже показана схема расположения генов на выбранном участке
В качестве таксона для поиска гомологов я выбрал группу Accipitriformes (taxid:2558200)
Результаты 4 бластов находятся в таблице ниже
| Алгоритм | Как используется | Word length | Количество находок |
| blastn | Используется для определения консервативности последовательности, видовой (таксономической) принадлежности. Можно изучать мобильные элементы (ДНК ретровирусов и подобное) |
15 | 65 |
| megablast | Используется для картирования на известный геном, либо поиска очень походих последовательностей | 16 | 46 |
| blastx | Используется для нахождения и исследования схожих белков | 6 | 178 |
| tblastx | Используется для предсказания CDS | 3 | Алгоритм не смог завершить работу, поскольку требуется слишком много вычислительной мощности (А у NCBI время ограничено примерно 3 минутами) |
Результаты выдачи blast
1. blastn проводит поиск по нуклеотидной БД. В моём случае ген Sox17 кодирует эукариотический белок, в котором присутствуют интроны, подверженные большему количеству мутаций. Поиск в целом имеет смысл, но не удивительно, что результатов не так много по сравнению с выравниваниями регуляторных/более консервативных последовательностей.
2. С megablast'ом ситуация в целом схожая, однако он менее чувствительный (требует большей схожести последовательностей). Неудивительно, что находок он выдал ещё меньше чем blastn.
3. blastx выдал гораздо больше находок, поскольку этот алгоритм больше подходит под задачу. Он транслирует нуклеотидную последовательность в белок и ищет уже по белковой БД, что соответствует реальным процессам (ген Sox17 как раз кодирует белок).
В целом, величины e-value стартуют со значения 0.0 (последовательности абсолютно точно гомологичны) и заканчиваются на значении около 0.007 (вероятна гоология, но послежовательности всё же далёкие) (у всех алгоритмов в выдаче приблизительно такие значения). Это похоже на распределение, которое получается в бласте обычно, никаких особых аномалий, несмотря на то, что blastn не очень подходит для выбранной задачи.
Упражнение 2
Была сделана небольшая датабаза на основе сборки GRCm39.fastaКоманда: "makeblastdb -in GRCm39.fasta -dbtype nucl"
Из файла были получены 2 рибосомальных РНК Ecoli, 16S (из малой субъединицы) и 23S (из большой субъединицы). Для поиска был использован blastn, так как рРНК не транслируются (значит искать по белковой послежовательности бессмысленно)
Команды для поиска:
- blastn -task blastn -query 16S.fasta -db GRCm39.fasta -out blastn_16S.txt -evalue 0.05 -outfmt 7
- blastn -task blastn -query 16S.fasta -db GRCm39.fasta -out blastn_16S.txt -evalue 0.05 -outfmt 7
Использовались стандартные параметры с сайта NCBI
Всего было найдено 1 совпадение для 16S RNA и 22 для 23S RNA.
Единственное совпадение для 16S найлено в 17 хромосоме, и его e-value неплохое (возможно там сидит ген 18S эукариотической рРНК)
Находки для 23S раскиданы по разным хромосомам (вероятно находки связаны с гомологичными 28S RNA эукариот).
Что примечательно, есть находки в митохондриальном геноме. Неожиданно её e-value оказался очень высоким (0.007), несмотря на прокариотическое прошлое митохондрий.