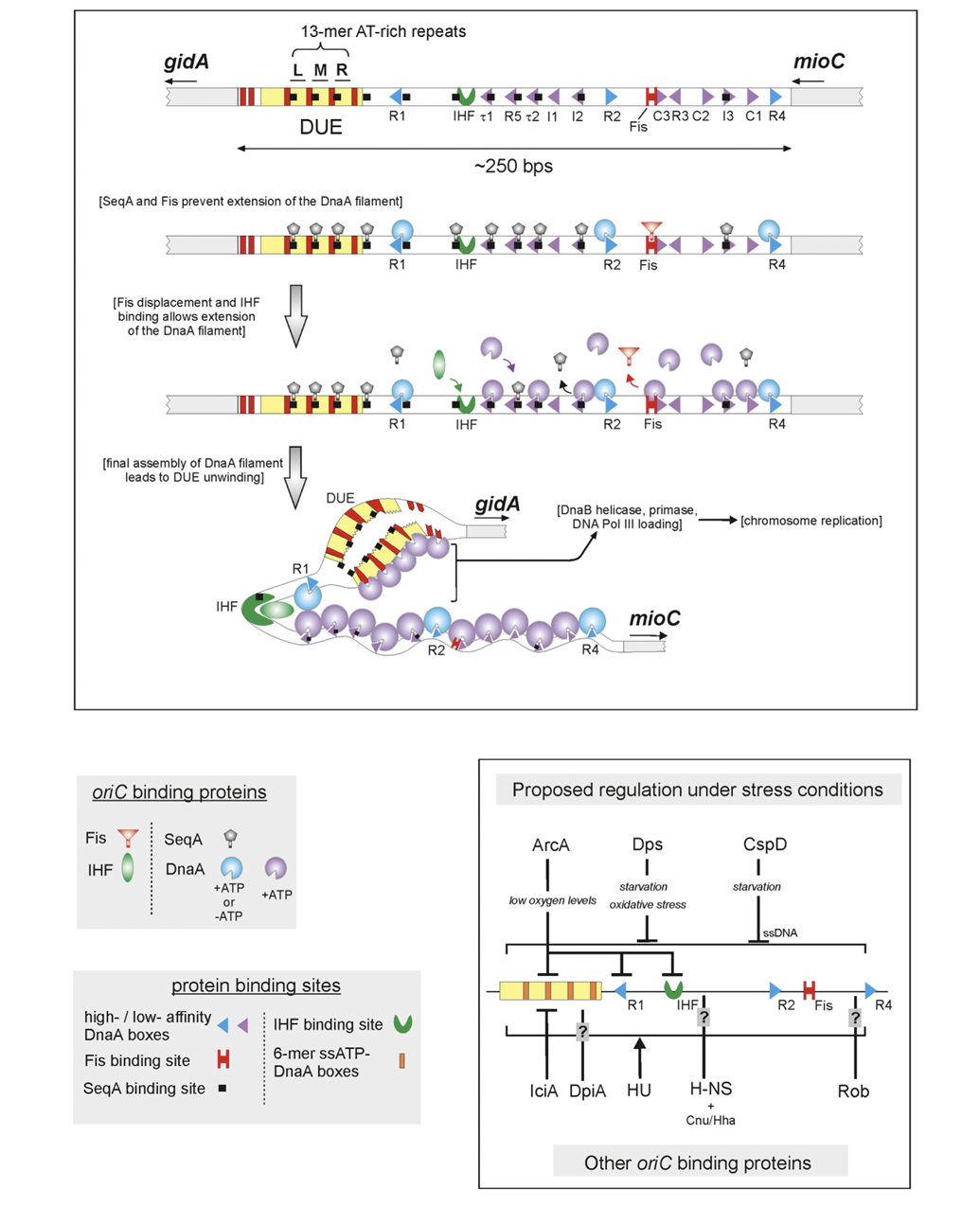
В данном практикуме исследовались множественные сигналы OriC - ориджина репликации бактерий. OriC - последовательность ДНК прокариот(~250 нуклеотидных пар), на которой содержится множество мотивов, служащих сигналом для связывания с разнообразными регуляторными белками(Fig. 1). Основным таким белком является DnaA: связывание множества DnaA способствует плавлению DUE(АТ-богатого сайта). Также есть мотивы узнаваемые белком SeqA, препятствующим связыванию DnaA. Репликация в основном регулируется на этапе инициации через OriC, в том числе в стрессовых условиях за счёт других регуляторных белков(Fig. 1 схема в правом нижнем углу). Носители сигналов OriC это не только мотивы, наличие или отсутствие SeqA, но ещё и пространственная структура DUE: в расплавленном виде в неё встраиваются белки репликативной вилки(хеликаза DnaB, DNA Pol III и др.). Таким образом OriC содержит множественные сигналы для начала репликации в целом и для участвующих в этом процессе белков в частности.

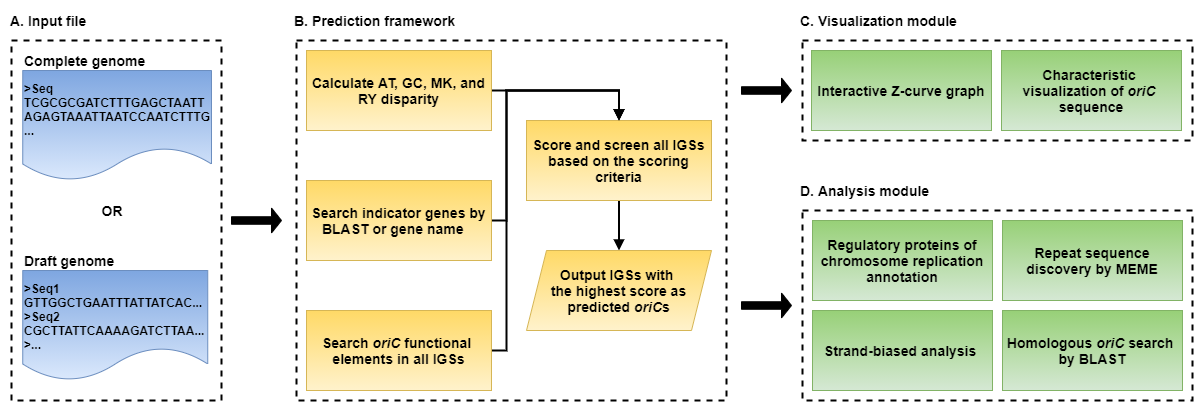
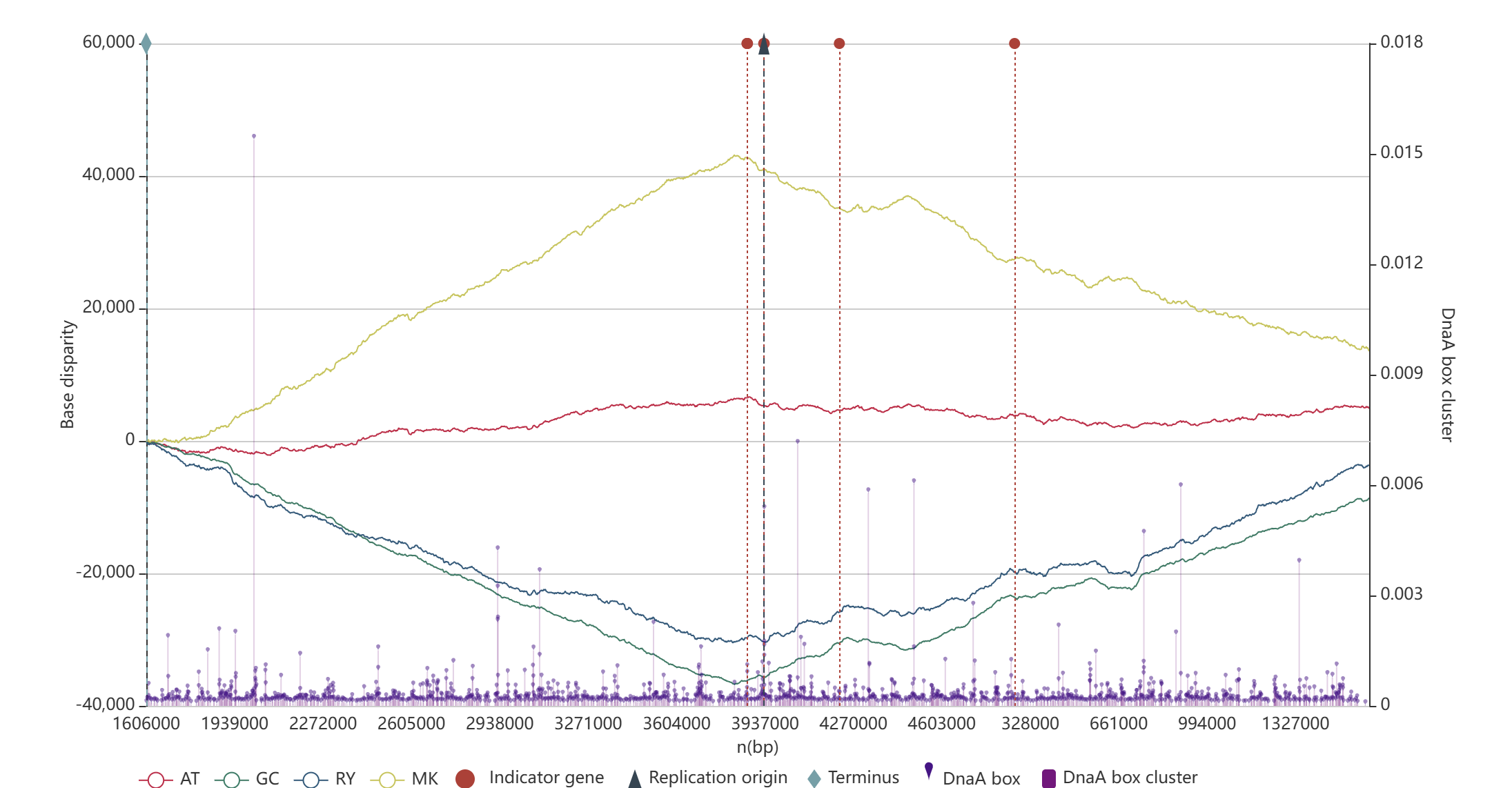
В основе работы Ori-Finder 2022 лежит теория Z-кривой[2]. Принцип изображён на Fig. 2. Программа принимает на вход последовательность генома или аннотированные геномные последовательности, затем считает кривую плотности CG и AT - состава, MK и RY соотношения; при помощи blast гены-маркеры и мотивы регуляторных белков в IGSs. Затем межгенным последовательностям присваивается вес, основываясь на всём, что посчитано заранее. Далее программа предсказвает OriC на основе веса.
Программу по поиску OriC было решено проверить подав в неё главную хромосому Shigella flexneri 2a str.301. Этот организм был выбран, так как он хорошо изучен и я с ним ранее уже работал. После завершения работы программа выдаёт две web страницы. На первой мы можем увидеть общую информацию о запуске(Fig. 3) и проекции Z-кривой, отражающие GC, AT, MK и RY соотношения, и отмеченные маркерные гены, мотивы регуляторных белков и предсказанные OriC(Fig. 4).
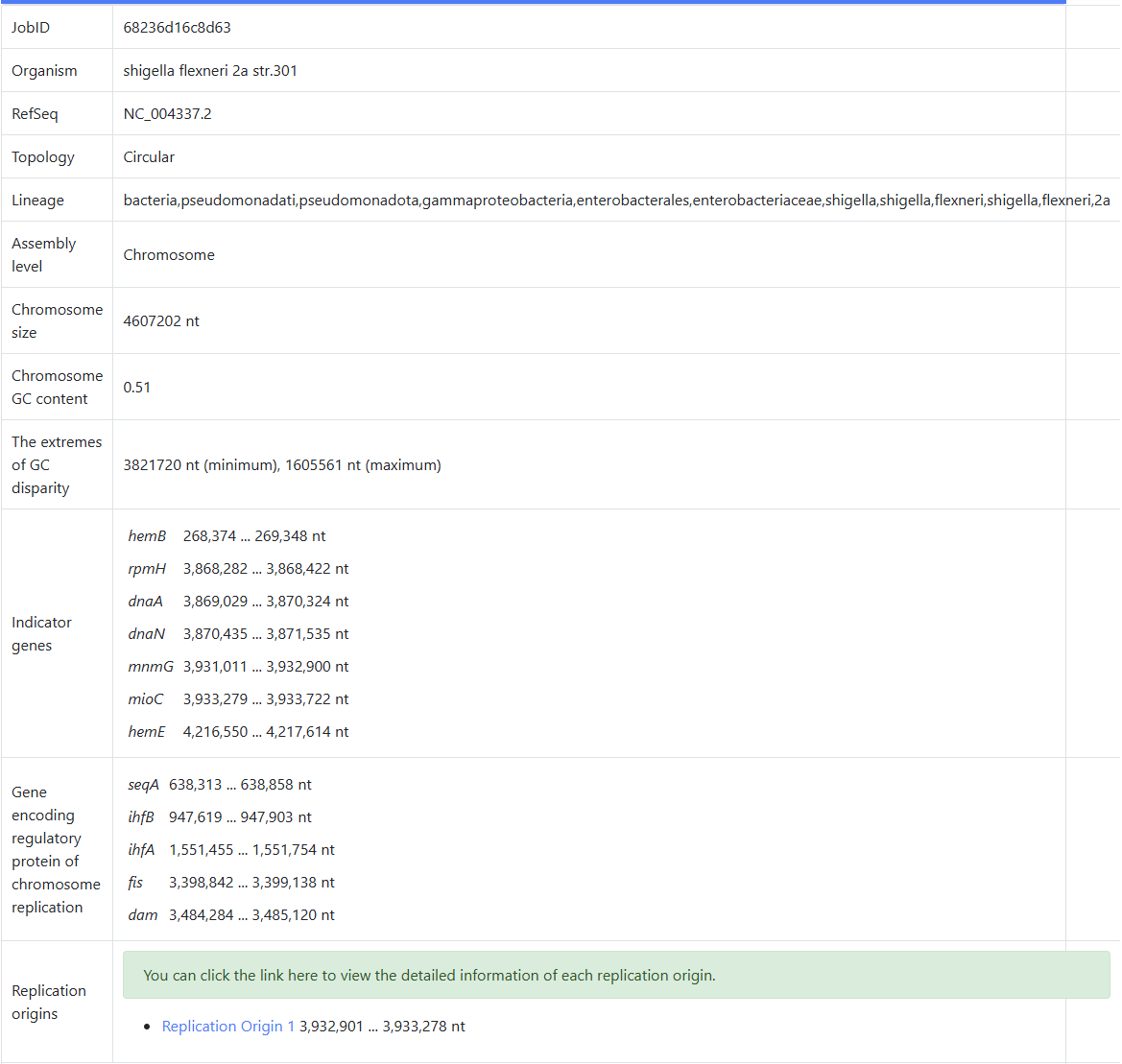
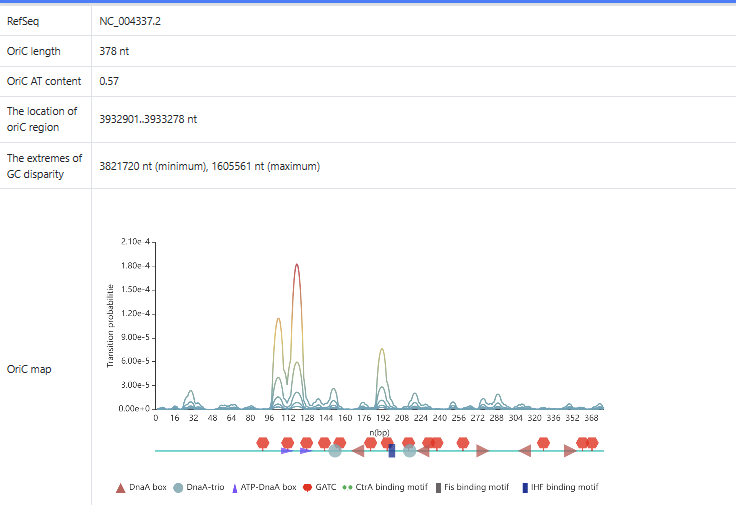
На второй web странице мы видим детальную информацию касательно предсказанного OriC: график зависимости
лёгкости плавления от позиции нуклеотида рассчитаных при 5 разных значениях закрученности ДНК при помощи SIST
и схема мотивов на OriC ниже(Fig. 5), последовательность OriC с отмеченными известными мотивами и возможные
новые мотивы среди повторяющихся участков, найденные MEME(Fig. 6).
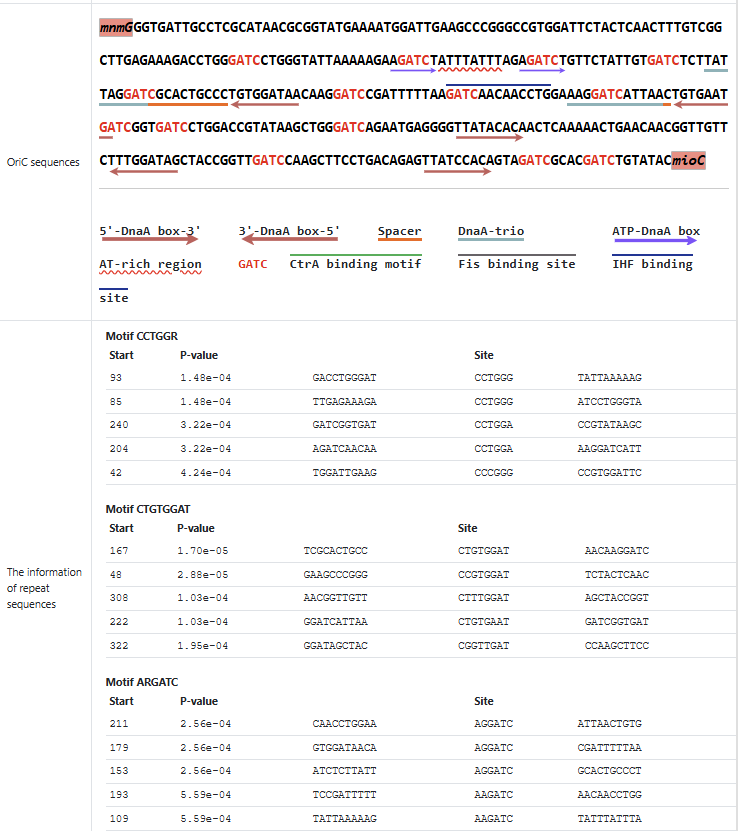
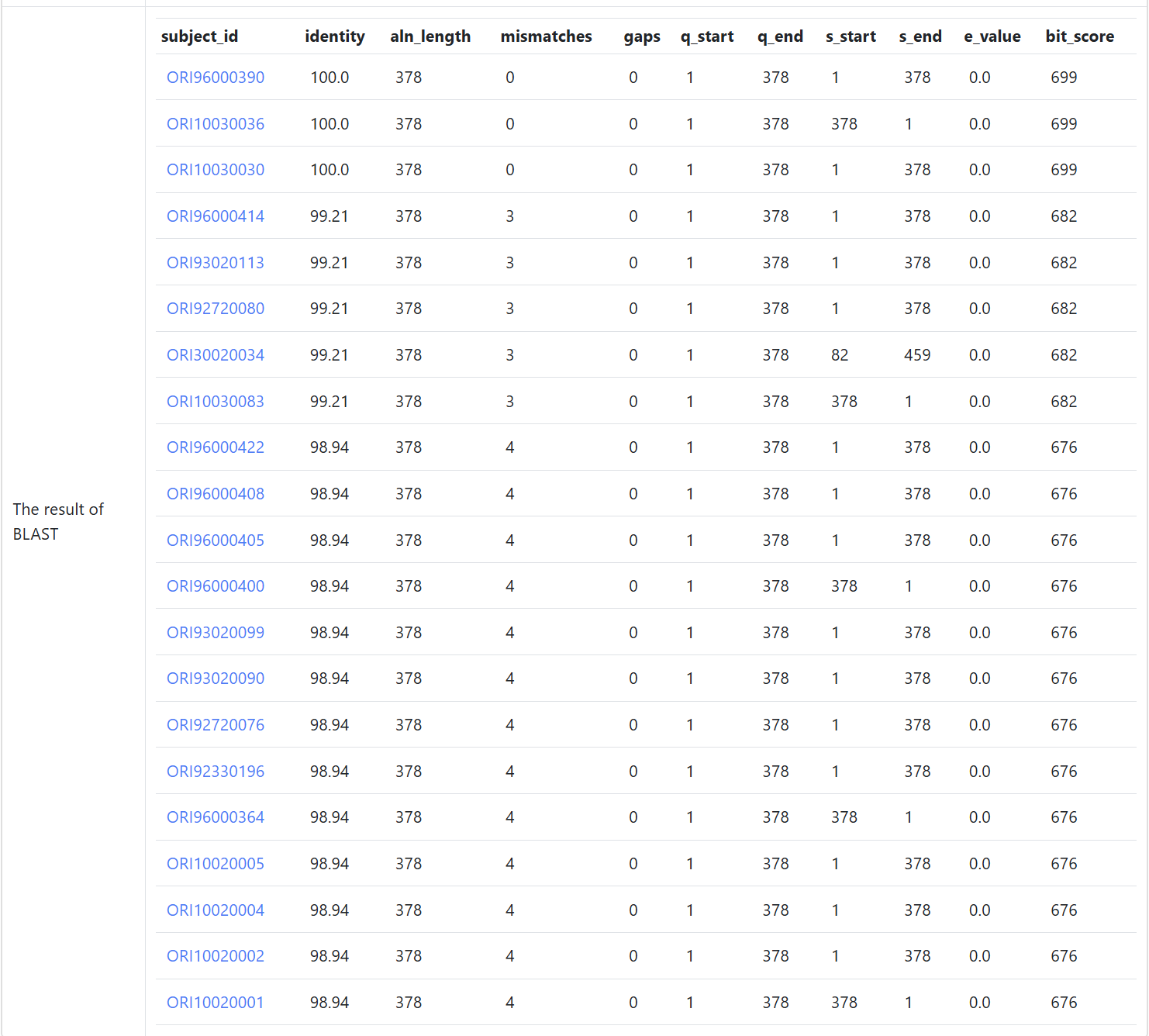
На второй странице мы также можем увидеть результаты работы бласта по выравниванию нашей предсказанной OriC на известные OriC, лежащие в базе(Fig.7). Предсказанная OriC на 100% идентична лежащей в базе для Shigella flexneri 2a, так что программа для поиска работает.