|
Главная
I Семестр
II Семестр
Проекты
Обратная Связь
|
Работа в командной строке Linux. Программы выравнивания последовательностей пакета EMBOSS.
Задание 1 (Работа в командной строке Linux)
1) Смена активной директории и просмотр содержимого директории:
- “ls” – показало мне содержимое текущей директории
- “ls ..” – показало мне содержимое родительской директории (т.е. содержимое той директории, в которой лежит данная).
- "cd .." – ничего не отображает. “ls” – показало мне содержимое родительской директории. Исходя из этого я понял,
что "cd .." - это выход из данной поддиректории
- “cd Term_2” переход в поддиректорию с названием Term_2
- "pwd" - сообщает полное имя директории, в которой ты сейчас находишься
2) Создание и просмотр файлов:
- “seqret sw:p0aa43 –auto” - совершив данную команду я обнаружил, что у меня в папке появилась последовательность белка в fasta-формате
"more rsua_ecoli.fasta" – показало мне содержимое данного файла
- “entret sw:p0aa43 –auto” - совершив данную команду я обнаружил, что у меня в папке появился файл, в которой содержится информация о моем белке на основе банка Uniprot
"more rsua_ecoli.entret" – показало мне содержимое данного файла
С помощью мы можем пролистывать по одной строчке, а с помощью "пробел" пролистывается целая страница
3) Некоторые способы облегчения работы в командной строке:
- С помощью клавиш "стрелка верх" и "стрелка вниз" мы можем смотреть на предыдущие команды, которые мы совершили
- “history” – выдает нам полный перечень команд, которые мы когда-либо вводили
- Клавиша “TAB” – помогает нам быстро выбирать файлы. То есть если мы ввели букву и на нее начинается только один файл, то он нам выведет именно этот файл.
Если же на одну букву начинается название нескольких файлов, то при нажатие “TAB” он нам выдает общую часть в название, учитывая все файлы, начинающиеся на
данную букву. При нажатие двух раз “TAB” он нам выдает список файлов, которые начинаются на данную общую часть (это может быть и одна буква)
Задание 2 (Построить и сравнить оптимальные глобальное и оптимальное локальное выравнивание 2-х последовательностей)
1) Построить полное (глобальное) оптимальное выравнивание с помощью программы needle пакета EMBOSS
- a)Введя команду: “needle rsua_ecoli.fasta y129_borbu.fasta seq_1n.needle –auto” я обнаружил у себя в папке файл с названием seq_1n.needle.
Вот собственно какая информация в нем содержится: вверху имеется две выделенных шапки.
В первой шапке содержится информация о том:
- какая программа совершила данное выравнивание
-когда собственно было совершенно выравнивание
-какие файлы выравнивались
-какой файл получился
-автоматическое выравнивание, т.е. без введения своих значений
-какой формат выравнивания использовался
-отчетный файл
Во второй шапке содержится информация о том:
-какие последовательности выравнивались
-какая матрица замен была использована для данного выравнивания
-штраф за гэп
-штраф за удлинение
-длина выравнивания
-идентичность выровненных последовательностей
-сходность выровненных последовательностей
-количество гэпов
-вес выравнивания
А далее собственно идет само выравнивание.
- b) При изменение параметров штрафов за гэпы, изменились также и другие параметры такие, как: длина выравнивания, идентичность последовательностей,
сходность последовательностей, количество гэпов, вес выравнивания(файл seq_2n.needle).
- c) Введя команду: “needle rsua_ecoli.fasta y129_borbu.fasta seq_3n.msf –auto -aformat msf ” я обнаружил у себя в папке файл с названием seq_3n.msf .
Аналогично проделал и без команды –auto и получил файл seq_4n.msf
2) Построить локальное (частичное) оптимальное выравнивание с помощью программы water пакета EMBOSS
- Введя команду: “water rsua_ecoli.fasta y129_borbu.fasta seq_1w.water –auto” я обнаружил у себя в папке файл с названием seq_1w.water.
В файле также имеется две шапки, содержимое которых точно такое же как и в результате команды “needle”.
Введя команду: “water rsua_ecoli.fasta y129_borbu.fasta seq_3w.msf –auto -aformat msf ” я обнаружил у себя в папке файл с названием seq_3w.msf
Изменил параметры штрафов за гэпы:
- a) Gap_penalty: 20.0
Extend_penalty: 1.0
Сохранил в файл seq_2w.water
- b) Gap_penalty: 5.0
Extend_penalty: 0.25
Сохранил в файл seq_5w.water
Соответственно к этим файлам также имеются .msf файлы, seq_4w.msf (для seq_2w.water) и seq_6w.msf (для seq_5w.water) соответственно.
3) Сравнить полученные выравнивания
- a)Имеются примеры того, что одной и той же позиции первой последовательности в
разных глобальных выравниваниях сопоставлены разные позиции второй последовательности.
Это показано на картинке. Слева соответственно выравнивание с автоматическим штрафом за гэп, а слева с удвоенным штрафом за гэп.
Синем обозначено, где сопаставлены разные позиции. А зеленым, где позициям сопоставлены гэпы.
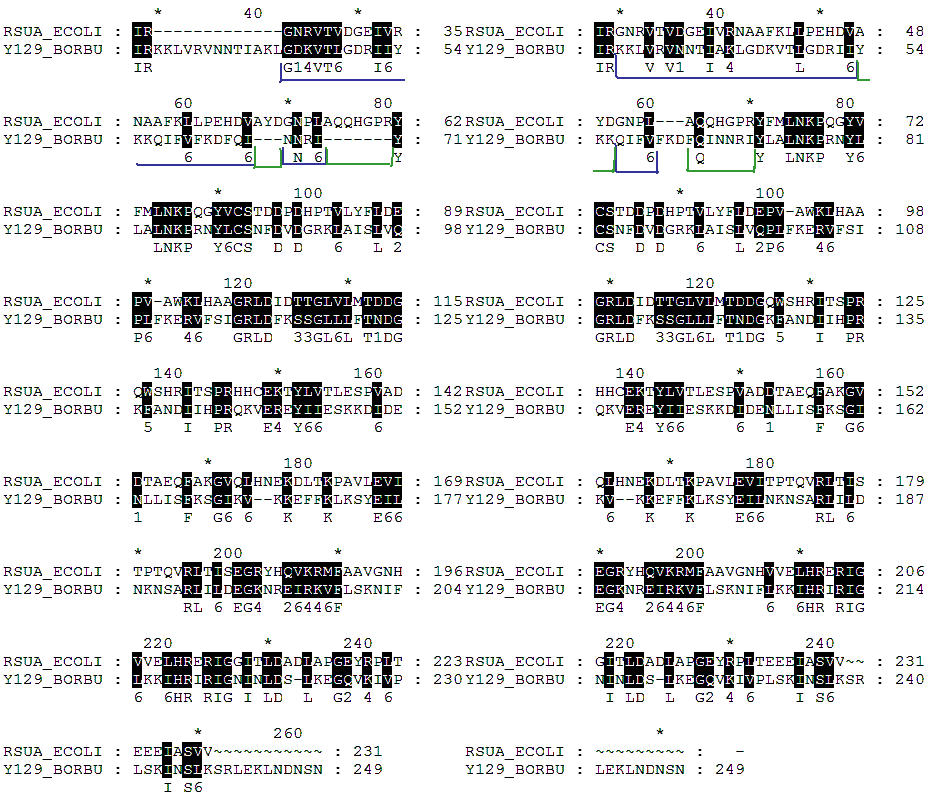
№(24-47)-rsua_ecoli соответсвует №(43-66) у 129_borbu(из seq_3n.msf) и №(30-53)(из seq_4n.msf)
№(51-54)-rsua_ecoli соответсвует №(67-70) у 129_borbu(из seq_3n.msf) и №(57-60)(из seq_4n.msf)
Позиции считаются близкородственными, когда значение элемента матрицы BLOSUM62 положительно.
Имеется три примера того, что одной и той же позиции первой последовательности в разных глобальных выравниваниях
близкородственны разные позиции второй последовательности:
№33-rsua_ecoli соответсвует №52 у 129_borbu(из seq_3n.msf) и №39(из seq_4n.msf)
№47-rsua_ecoli соответсвует №66 у 129_borbu(из seq_3n.msf) и №53(из seq_4n.msf)
№54-rsua_ecoli соответсвует №70 у 129_borbu(из seq_3n.msf) и №60(из seq_4n.msf)
- b)Имеются примеры того, что одной и той же позиции первой последовательности в разных локальных выравниваниях
сопоставлены разные позиции второй последовательности.
Это показано на картинке. Слева соответственно выравнивание с автоматическим штрафом за гэп, а слева выравнивание с уменьшиным вдвое штрафом за гэп.
Синем обозначено, где сопаставлены разные позиции. А зеленым, где позициям сопоставлены гэпы.

№(11-12)-rsua_ecoli соответсвует №(11-12) у 129_borbu(из seq_3w.msf) и №(10-11)(из seq_6w.msf)
№(30-31)-rsua_ecoli соответсвует №(43-44) у 129_borbu(из seq_3w.msf) и №(42-43)(из seq_6w.msf)
№(32-36)-rsua_ecoli соответсвует №(45-49) у 129_borbu(из seq_3w.msf) и №(46-50)(из seq_6w.msf)
№(92-93)-rsua_ecoli соответсвует №(96-97) у 129_borbu(из seq_3w.msf) и №(94-95)(из seq_6w.msf)
№(136-137)-rsua_ecoli соответсвует №(140-141) у 129_borbu(из seq_3w.msf) и №(138-139)(из seq_6w.msf)
№(140-145)-rsua_ecoli соответсвует №(144-149) у 129_borbu(из seq_3w.msf) и №(140-145)(из seq_6w.msf)
№(192-197)-rsua_ecoli соответсвует №(194-199) у 129_borbu(из seq_3w.msf) и №(192-197)(из seq_6w.msf)
Имеется два примера того, что одной и той же позиции первой последовательности в разных локальных
выравниваниях близкородственны разные позиции второй последовательности:
№32-rsua_ecoli соответсвует №45 у 129_borbu(из seq_3w.msf) и №46(из seq_6w.msf)
№196-rsua_ecoli соответсвует №198 у 129_borbu(из seq_3w.msf) и №196(из seq_6w.msf)
Если сравнивать еще файл seq_4w.msf, то он точно такой же как и seq_3w.msf, только значительно короче. То есть если смотреть по seq_3w.msf
он начинается с 61(rsua_ecoli) и 64(129_borbu), а кончается 211(rsua_ecoli) и 213(129_borbu). Поэтому у него будет такие же отличия с
seq_6w.msf как у seq_3w.msf. Поэтому начиная с этого задания seq_4w.msf мы учитывать не будем.
- c)Имеются примеры того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а
в другом выравнивании против той же позиции оказался пропуск. Смотри картинку для глобального выравнивания выше.
№(48-50)-rsua_ecoli соответсвует гэпы у 129_borbu(из seq_3n.msf) и №(54-56)(из seq_4n.msf)
№(55-61)-rsua_ecoli соответсвует гэпы у 129_borbu(из seq_3n.msf) и №(64-70)(из seq_4n.msf)
Имеется один пример того, что в одном глобальном выравнивании какой-либо позиции первой последовательности близкородственна некоторая позиция второй,
а в другом выравнивании против той же позиции оказался пропуск:
№56-rsua_ecoli соответсвует гэп у 129_borbu(из seq_3n.msf), а №65(из seq_4n.msf)
- d)Имеются примеры того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а
в другом выравнивании против той же позиции оказался пропуск. Смотри картинку для локального выравнивания выше.
№10-rsua_ecoli соответсвует №10 у 129_borbu(из seq_3w.msf), гэп (из seq_6w.msf)
№49-rsua_ecoli соответсвует гэп у 129_borbu(из seq_3w.msf), №56 (из seq_6w.msf)
№(134-135)-rsua_ecoli соответсвует №(138-139) у 129_borbu(из seq_3w.msf), гэпы (из seq_6w.msf)
№(138-139)-rsua_ecoli соответсвует №(142-143) у 129_borbu(из seq_3w.msf), гэпы (из seq_6w.msf)
№(190-191)-rsua_ecoli соответсвует №(192-193) у 129_borbu(из seq_3w.msf), гэпы (из seq_6w.msf)
Имеется один пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности близкородственна некоторая позиция второй, а в
другом выравнивании против той же позиции оказался пропуск:
№40-rsua_ecoli соответсвует №53 у 129_borbu(из seq_3w.msf), гэп (из seq_6w.msf)
- e)Как я уже упоминал выше, что seq_4w.msf соответствует seq_3w.msf, хотя были использованы разные параметры.
- f)seq_4w.msf полностью совпадает с seq_3n.msf. Если смотреть по seq_3n.msf он начинается с 62(rsua_ecoli) и 71(129_borbu), а кончается 212(rsua_ecoli) и 220(129_borbu).
seq_3w.msf практически полностью совпадает с seq_3n.msf. Если смотреть по seq_3n.msf он начинается с 2(rsua_ecoli) и 8(129_borbu), а кончается 224(rsua_ecoli) и 233(129_borbu).
Не совпадают только последний участок. На картинке представлена эта разница. Первым соответственно идет seq_3n.msf, а ниже seq_3w.msf.
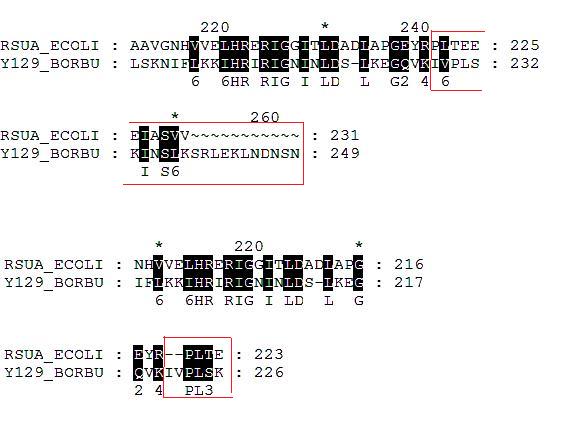
seq_6w.msf отличается от seq_3n.msf на столько же, сколько он отличается от seq_3w.msf
Отличия seq_4n.msf заключаются лишь в его отличии от seq_3n.msf. И поэтому если у него есть отличия с seq_3n.msf, то эти же отличия у него и с seq_3w.msf, seq_4w.msf и seq_6w.msf.
Скачать потокол c проделанным заданием.
Дополнительные задания
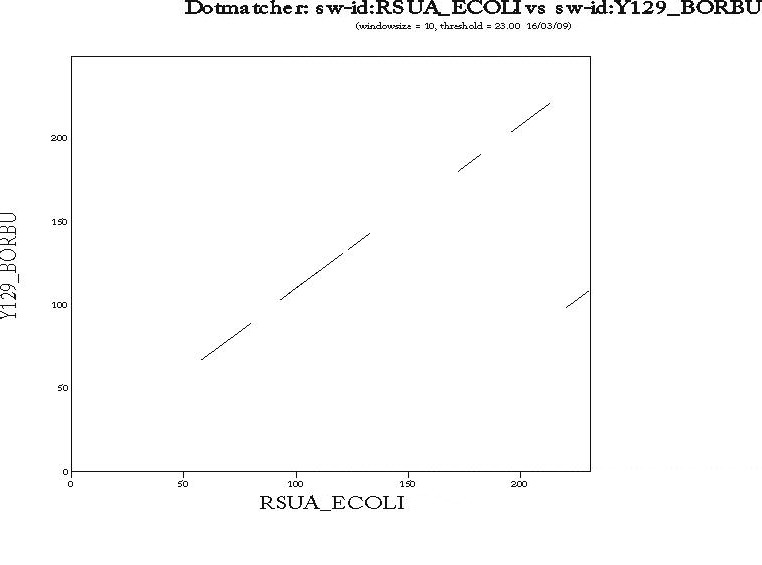
Построил карту локального сходства заданных последовательностей с помощью программы dotmatcher пакета EMBOSS. Изображение приведено ниже:
Получил несколько субоптимальных локальных выравниваний заданных последовательностей с помощью программы matcher пакета EMBOSS. Сохранил их в файл.
Выберем несколько субоптимальных выравниваний, в которых фрагменты последовательностей не совпадают с выровненными фрагментами в оптимальном выравнивании:
#===================================================
220 230
RSUA_E LAPGEYRPLTEEEIASV
:: .:: .: . :.
Y129_B LAISLVQPLFKERVFSI
100
#===================================================
160 170 180 190
RSUA_E LHNEKDLTKPAVLEVITPTQVRLTISEGRYHQVKRMF
.: . . . ..: : :: .::. :
Y129_B IHPRQKVEREYIIESKKDIDENLLISFKSGIKVKKEF
140 150 160
#===================================================
190 200
RSUA_E VKRMFAAVGNHVVEL
... : : . .:
Y129_B IRKKLVRVNNTIAKL
30 40
#===================================================
70 80 90 100
RSUA_E KPQGYVCSTDDPDHPTVLYFLDEPVAWKLHAAGRLDIDTTGLV
: : . :.: . . : . . ::: :.
Y129_B KSSGLLLFTNDGKFANDIIHPRQKVEREYIIESKKDIDENLLI
120 130 140 150
#===================================================
|



