|
Главная
I Семестр
II Семестр
Проекты
Обратная Связь
|
Множественное выравнивание последовательностей
1)Ознакомление с программой Muscle
Воспользовавшись SRS я получил из банка SwissProt файл с 34 последовательностями дельта-антигенов в формате fasta.
Сделал это ограничив по полю таксономии(Taxonomy): Deltavirus, а также по полю описание(Description): delta. Попробовал на глаз вставить пару гэпов. В
результате чего поставилв пару гэпов в колонку 163, я получил достаточно хорошее совпадение. Вот дана
картинка с получившимся выравниванием.
Затем выровнял данные последовательности воспользовавшись программой muscle, введя команду
muscle -in delta.fasta -out delta_aligned.fasta. В результате чего получил файл с выравниванием. Вот дана
картинка с получившимся выравниванием. По результатам выравнимания можно с уверенностью сказать, что
белки являются близкими гомологами, так как имеется большое количество консервативных участков.
Файл в формате msf, сохранённый из GeneDoc для невыровненных последовательностей: aln_hand.msf
Файл в формате msf, сохранённый из GeneDoc для выровненных последовательностей: aln_muscle.msf
2)Выравнивание набора гомологов своего белка
Для выравнивания получим гомологи белка RSUA_ECOLI посредством BLAST. Ограничиваем выдачу таксоном Bacteria и поставив порог на E-value, равный 0.001.
И берем тех гомологов, для которых процент идентичности с белком RSUA_ECOLI меньше 90%. Для того чтобы гомологи не были слишком близки друг к другу, выбираем их из разных родов организмов.
Ниже в таблице приведены выбранные мною гомологи белка RSUA_ECOLI.
Гомологи белка RSUA_ECOLI
| Индификатор |
e-value |
Идентичность |
Сходство |
Таксономия |
| Y807_RICCN |
2e-10 |
24% |
46% |
Bacteria; Proteobacteria; Alphaproteobacteria; Rickettsiales;
Rickettsiaceae; Rickettsieae; Rickettsia; Rickettsia conorii |
| RLUF_SHIFL |
4e-17 |
27% |
49% |
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Shigella; Shigella flexneri |
| Y1370_MYCLE |
2e-23 |
35% |
51% |
Bacteria; Actinobacteria; Actinobacteridae; Actinomycetales;
Corynebacterineae; Mycobacteriaceae; Mycobacterium; Mycobacterium leprae |
| RLUE_XANAC |
3e-19 |
36% |
52% |
Bacteria; Proteobacteria; Gammaproteobacteria; Xanthomonadales;
Xanthomonadaceae; Xanthomonas; Xanthomonas axonopodis pv. citri |
| Y361_SYNY3 |
6e-28 |
35% |
56% |
Bacteria; Cyanobacteria; Chroococcales; Synechocystis; Synechocystis sp. PCC 6803 |
| RSUA_PSEAE |
3e-50 |
46% |
59% |
Bacteria; Proteobacteria; Gammaproteobacteria; Pseudomonadales;
Pseudomonadaceae; Pseudomonas; Pseudomonas aeruginosa |
| Y554_AQUAE |
1e-38 |
38% |
62% |
Bacteria; Aquificae; Aquificales; Aquificaceae; Aquifex; Aquifex aeolicus |
| RSUA_VIBPA |
2e-64 |
53% |
68% |
Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales;
Vibrionaceae; Vibrio; Vibrio parahaemolyticus |
| RSUA_HAEIN |
3e-75 |
57% |
74% |
Bacteria; Proteobacteria; Gammaproteobacteria; Pasteurellales;
Pasteurellaceae; Haemophilus; Haemophilus influenzae |
Для сравнения приведу таксономию для бактерии из которой был выделен мой белок: Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Escherichia; Escherichia coli K-12
Создаю файл, в котором записал список выбранных белков-гомологов и мой белок. Затем с помощью команды
seqret @myproteins.list myproteins.fasta получаю файл в fasta формате. Затем с помощью программы muscle построил множественное выравнивание.
Вот представлена картинка с получившимся выравниванием. Ниже приведена таблица с
описанием консервативных выравненных участков и участков не имеющих биологического значения
Таблица описания выравнивания
| № а.о. белка RSUA_ECOLI |
№ столбцов выравнивания |
Комментарии |
Консервативные участки |
| 116-173 |
95-152 |
Мне кажется, что это наиболее консервативный участок, так как наблюдается достаточно большое количество совпадений а.о. К тому же
в этом диапазоне а.о. лежит Asp102, которая является для RSUA_ECOLI активным центром. А как мы знаем, что белки выполняющие приблизительно одинаковые
функцию должны иметь приблизительно одинаковые активные центры. |
| 216-250 |
176-210 |
Мне кажется, что этот участок также является консервативным, так как наблюдаем достаточное количество совпадений а.о. |
Остальным же учаткам, как мне кажется не стоит придовать особого биологического смысла |
| 178-213 |
157-173 |
Как мне кажется этот участок не несет какого-либо особого биологического смысла. |
Файл в формате fasta для полученных последовательностей: myproteins.fasta
Файл в формате msf, сохранённый из GeneDoc для выровненных последовательностей: myprot_mus.msf
3*)Другие программы множественного выравнивания
Попробовал разобраться с другими программами выравнивания, установленными на kodomo-count, а именно mafft и edialign. И выравнял последовательности,
которые выравнивал с помощью программы Muscle в предыдущем задание. Для этого вводил команду mafft либо edilign, после чего отвечал на вопросы.
В результате чего получил два множественных выравнивания. Вот картинки множественного выравнивания полученные с
помощью mafft и edialign соответственно.
Сравнивая выравнивания полученные с помощью 3 программ я пришел к выводу, что они практически полностью совпадают в плане консервативных участках. Одно из
отличий Mafft от Muscle и Edialign в том что он выравнивает первый столбец метионинов, но это не несет особого биалогического смысла, а просто разница в алгоритмах.
А отличие Edialign от Muscle и Mafft в том что он не выравнивает столбец а.о. в котором стоит 152 а.о. по RSUA_ECOLI. А также Mafft и Edialign не меняют порядок
последовательностей из вводимого файла, в отличие от Muscle.
Файл в формате msf, сохранённый из GeneDoc для выровненных последовательностей полученных с помощью программы Mafft: myprot_maf.msf
Файл в формате msf, сохранённый из GeneDoc для выровненных последовательностей полученных с помощью программы Edialign: myprot_edi.msf
4*)Знакомство с некоторыми программами обработки множественных выравниваний
CONSAMBIG - программа пакета EMBOSS, которая создает неоднозначную последовательность от множественного выравнивания. А именно последовательность на местах совпадения
а.о. в этой последовательности стоят совпавшие а.о. Когда а.о. не совпадают стоит Х. Также ставят а.о. если на этом участке есть только одна последовательности,
так как другие либо закончились, либо просто стоят гэпы.
Файл в формате fasta для неоднозначной последовательности зависящей от множественного выравнивания, полученного с помощью программы Muscle: myprot_mus_con.fasta
DISTMAT - программа пакета EMBOSS, которая создает матрицу расстояния от множественного выравнивания. Она вычисляет расстояние между каждой парой последовательностей в множественном выравнивании.
Методы для того чтобы оценить это расстояние можно выбрать и отличаться по тому, как исправляются замены. Я выбрал метод который обзывается: "Неисправленные расстояния", т.е.
не делаются попраки на множественные замены. Ниже приведена матрица расстояния для множественного выравнивания полученного с помощью программы Mafft.
| RSUA_ECOLI 1 |
0.00 |
|
|
|
|
|
|
|
|
|
| Y807_RICCN 2 |
74.18 |
0.00 |
|
|
|
|
|
|
|
|
| RLUF_SHIFL 3 |
69.16 |
66.06 |
0.00 |
|
|
|
|
|
|
|
| Y1370_MYCLE 4 |
63.56 |
67.73 |
67.11 |
0.00 |
|
|
|
|
|
|
| RLUE_XANAC 5 |
62.20 |
71.17 |
71.69 |
60.24 |
0.00 |
|
|
|
|
|
| Y361_SYNY3 6 |
63.72 |
65.64 |
66.23 |
58.72 |
58.72 |
0.00 |
|
|
|
|
| RSUA_PSEAE 7 |
53.04 |
71.36 |
70.42 |
65.18 |
66.87 |
64.00 |
0.00 |
|
|
|
| Y554_AQUAE 8 |
60.54 |
70.37 |
68.04 |
59.23 |
63.03 |
64.94 |
65.77 |
0.00 |
|
|
| RSUA_VIBPA 9 |
45.65 |
72.09 |
72.69 |
64.76 |
64.07 |
64.91 |
55.46 |
59.56 |
0.00 |
|
| RSUA_HAEIN 10 |
42.42 |
74.77 |
68.37 |
67.26 |
64.85 |
67.40 |
59.13 |
63.39 |
45.89 |
0.00 |
|
1 |
2 |
3 |
4 |
5 |
6 |
7 |
8 |
9 |
10 |
PLOTCON - программа пакета EMBOSS, которая строит график зависимости сходства а.о. от положения а.о. множественного выравнивания.
Ниже приведены графики множественных выравниваний, полученных с помощью программ Muscle и Mafft.
График множественного выравниваний, полученных с помощью программы Muscle
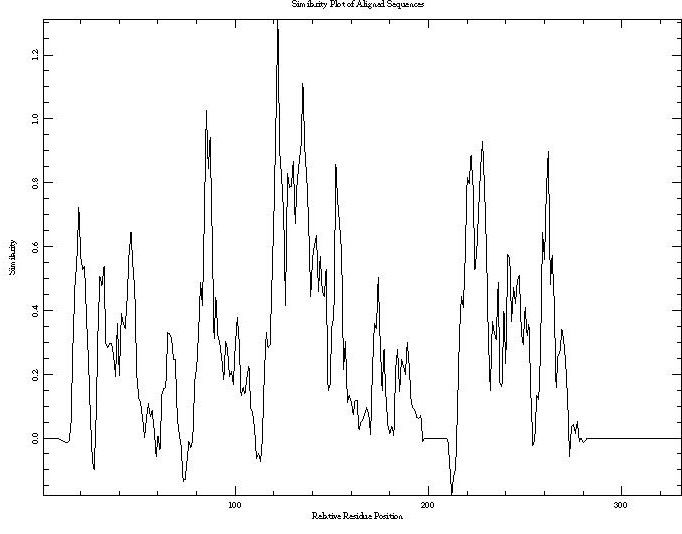
График множественного выравниваний, полученных с помощью программы Mafft
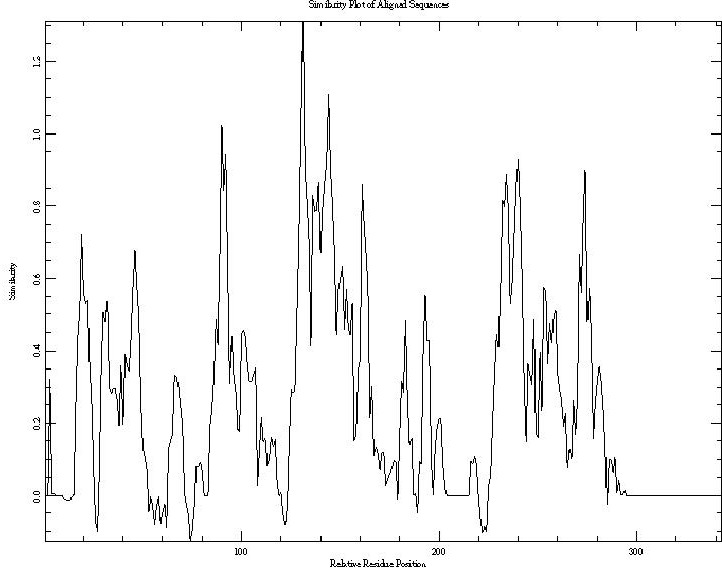
Из полученных графиков видно, что данные выравнивания не сильно отличаются. Отличаются тем что Mafft выравнивает первый столбец метионинов. А также
участками: 90-110 и 190-220.
|

