|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
Проекты
Обратная Связь
|
Ферменты и метаболические пути
- ЕС код фермента белка GLK_ECOLI
Открыл страницу UniProt с описанием белка
GLK_ECOLI и на ней был найден код EC=2.7.1.2.
По данным базы International Union of Biochemistry and Molecular Biology
код был расшифрован:
EC 2 - Трансферазы (Transferase)
EC 2.7 - Переносчики фосфор-содержащие группы (Transferring phosphorus-containing groups)
EC 2.7.1 - Фосфотрансферазы со спиртовой акцепторной группой (Phosphotransferases with an alcohol group as acceptor)
EC 2.7.1.2 - Глюкокиназа (Glucokinase)
Фермент осуществляет следующую реакцию:
ATP + D-glucose = ADP + D-glucose 6-phosphate
Графическое изображение катализируемой реакции:
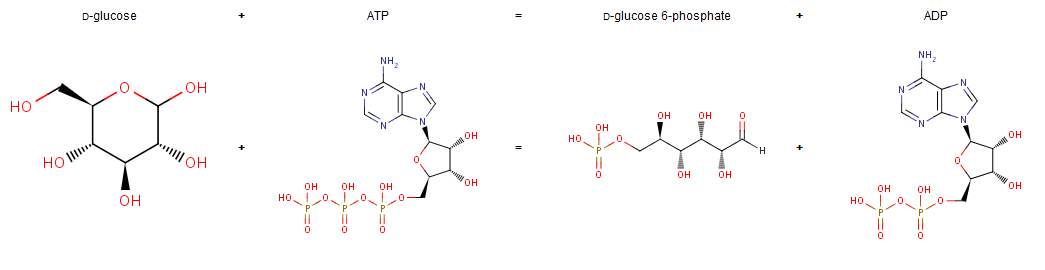
- Метаболические пути фермента EC=2.7.1.2
В записи о белке GLK_ECOLI в БД UniProt было найдено название локуса гена: b2388.
В результате поиска гена по названию локуса в базе KEGG среди генов Escherichia coli K12
была найдена запись eco:b2388. В поле Pathways этой записи содержатся 6 идентификаторов метаболических путей, ассоциированных с этим геном:
| ID |
Русское название |
Английское название |
| eco00010 |
Гликолиз / Глюконеогенез |
Glycolysis / Gluconeogenesis |
| eco00052 |
Метаболизм галактозы |
Galactose metabolism |
| eco00500 |
Метаболизм крахмала и сахарозы |
Starch and sucrose metabolism |
| eco00520 |
Метаболизм амино сахара и нуклеотид сахара |
Amino sugar and nucleotide sugar metabolism |
| eco00521 |
Биосинтез стрептомицина |
Streptomycin biosynthesis |
| eco01100 |
Метаболические пути |
Metabolic pathways |
Скачать карту гликолиза. Красным отмечен фермент глюкокиназа.
- Структурные формулы химических соединений
В базе данных химических соединений KEGG LIGAND был проведен поиск двух веществ:
| ID |
Русское название |
Английское название |
Структура |
| C00147 |
Аденин |
Adenine; 6-Aminopurine |
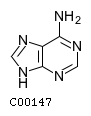
|
| C00242 |
Гуанин |
Guanine; 2-Amino-6-hydroxypurine |
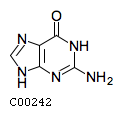
|
- Поиск метаболического пути между двумя веществами
Поиск осуществлялся в БД KEGG Pathway между веществами аденин и гуанин (см. предыдущее задание):
Выбранная цепочка ферментативных реакций:
путь: метаболизм пурина
цепочка: аденин → гуанин
Путь от аденина к гуанину через промежуточные соединения AMP (C00020), IMP (C00130), XMP (C00655) и GMP (C00144) -
5 стадий катализа, был отмечен на карте: красным отмечено первое
соединение цепочки, желтым - промежуточные, зеленым - последнее соединение цепочки.
- Сравнение метаболических путей у разных организмов
Карта, полученная в предыдущем задании, была переведена в режим разных организмов. Полученные результаты занесены в таблицу.
Возможность выбранной цепочки ферментативных в разных организмах с известными полными геномами.
| Организм |
Возможна ли цепочка реакций
(да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 |
нет
показать карту |
так как нет необходимых ферментов (отсутствует фермент EC=3.5.4.6, осуществляющий превращение AMP в IMP) |
| Archaeoglobus fulgidus |
нет
показать карту |
так как нет необходимых ферментов (есть только ферменты EC=1.1.1.205 и EC=6.3.5.2, осуществляющие соответственно превращающие IMP в XMP и XMP в GMP) |
| Arabidopsis thaliana |
да
показать карту |
присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Homo sapiens |
да
показать карту |
присутствуют все ферменты, необходимые для осуществления цепочки реакций |
- Сравнение ферментов из далеких организмов
Для сравнения был взят фермент с ЕС=4.2.1.11.
Этот фермент, согласно расшифровке IUBMB, является лиазой, а конкретно - фосфопируват гидратазой (энолазой).
В SRS был создан запрос для поиска фермента среди белков человека и археи Archaeoglobus fulgidus.
Опция маски для EC номера была снята, чтобы не были найдены такие номера, как 4.2.1.110, 4.2.1.111 и другие.
Текст запроса:
(([uniprot-ID:*_HUMAN*] | [uniprot-ID:*_ARCFU*]) & [uniprot-ECNumber:4.2.1.11])
Было найдено 13 находки, среди которых 12 принадлежат человеку, а одна - архее.
В режиме SW_InterProMatches были сравнены доменные организации найденных белков.
| ID |
Pfam AC |
Изображение |
| ENOA_HUMAN |
PF00113
PF03952 |
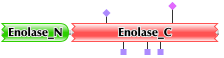
|
| ENOB_HUMAN |
PF00113
PF03952 |
|
| ENOG_HUMAN |
PF00113
PF03952 |

|
| ENO_ARCFU |
PF00113
PF03952 |
|
| A4QMW8_HUMAN |
PF00113 |

|
| A4UCS8_HUMAN |
PF00113
PF03952 |

|
| A8K3B0_HUMAN |
PF00113
PF03952 |

|
| B4DUJ6_HUMAN |
PF00113
PF03952 |

|
| B7Z2X9_HUMAN |
PF00113
PF03952 |

|
| Q6FHV6_HUMAN |
PF00113
PF03952 |

|
| Q96GV1_HUMAN |
PF00113 |

|
| Q9BT62_HUMAN |
PF00113 |

|
| Q9NPL4_HUMAN |
PF00113 |

|
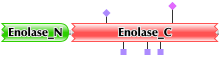
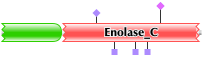
Из таблицы можно видеть то, что все находки имеют похожую доменную организацию. В одних находках встречается два домена:
PF00113 (Enolase, C-terminal TIM barrel domain) и
PF03952 (Enolase, N-terminal domain),
а в каких-то только домен: PF00113. И домены в значительно степени отличаются по длине.
Для дальнейшего анализа были выбраны белки: ENOA_HUMAN и ENO_ARCFU
Последовательности домена PF00113 из археи и человека были вырезаны из последовательностей белков.
Затем для определения процента сходства между ними эти последовательности были выравнены при помощи программы needle.
В результате чего получил файл.
В файле указан процент идентичности: 113/303 (37.3%). Импортировал выравнивание в msf формат и открыл в GeneDoc.
В результате чего получил изображение.
Ген, соответствующий белку ENOA_HUMAN - ENO1, id в KEGG - hsa:2023.
Ген, соответствующий белку ENO_ARCFU - eno, id в KEGG - afu:AF1132.
Лучший ортолог из архей для гена белка ENOA_HUMAN:
Организм: Sulfolobus islandicus Y.G.57.14
Ген: YG5714_1302
Функция: фосфопируват гидратаза
UniProt AC находки: C3NE30
Процент идентичности: 0.422
Лучший ортолог из эукариот для гена белка ENO_ARCFU:
Организм: Cryptococcus neoformans JEC21
Ген: CNC00160
Функция: фосфопируват гидратаза
UniProt AC находки: Q5KLA7
Процент идентичности: 0.403
Гомолог человеческого белка в археях имеет ферментативную функцию фосфопируват гидратазы (энолазы), также как и белок человека.
Гомолог белка археи в эукариотах также имеет ферментативную функцию фосфопируват гидратазы (энолазы), как и белок археи, причем организм, в
котором нашелся гомолог, принадлежит к царству грибы.
|















