Поиск белка с заданной функциональной специфичностью
- Первый этап: описание функциональных особенностей заданной группы
- Второй этап: создание множественного выравнивания доменов с разметкой по группам специфичности
- Создание хорошее множественное выравнивание доменов заданной группы белков
Посмотрим на разметку всех мотивов для белка прототипа MALI_ECOLI в БД InterPro. Используем домен из БД SMART, так как длина ДНК-связывающего домена SM00354 из БД SMART превосходит длины ДНК-связывающих доменов из других баз. Таким образом, мы получим больше информации. В таком случае его можно будет впоследствии "отрезать". Если бы были выбраны домены из другой БД, то часть информации могла потеряться.
Затем перешли по ссылке SM00354. Представительское выравнивание сохранено в файл SMART.fasta.
Последовательности всех бактериальных белков, содержащих домены данного семейства были сохранены в формате FASTA в файл family.fasta.
Был получен список идентификаторов последовательностей, по мнению эксперта, обладающих данной специфичностью. Было обнаружено, что в файле family.fasta, полученном из БД SMART, отсутствует один белков, указанных в списке идентификаторов последовательностей, по мнению эксперта, обладающих данной специфичностью. Эта последовательность была получены из БД UniProt.Был запущен скрипт script.txt:
#!/bin/bash for i in `cat mali`; do grep -A 5 ${i} family.fasta >> family_sort.fasta doneДанный скрипт ищет в файле family.fasta последовательности из списока, лежащего в файле mali. Для правильной работы скрипт и список должны быть сохранены в UNIX-формате. Выдача скрипта: файл family_sort.fasta. Затем к данному файлу была приписанна последовательность белка B5XRE8.С помощью программы ClustalW2 последовательности белков, содержащих нужные ДНК-связывающие домены были выровнены под профиль представительского выравнивания SMART.fasta. В результате чего были получены: файл с выравниванием и файл с деревом. Из файла с выравниванием были вырезаны представительские последовательности и позиции выравнивания, не содержащие ДНК-связывающего домена. Полученное выравнивание было сохранено в файл dna_mali.fasta
- Создание единое множественное выравнивание заданных доменов всех групп специфичности
Импортируем выравнивания всех групп специфичности в GeneDoc с раскраской по группам отдельно для ДНК-связывающих доменов и для эффекторных доменов. А также удалим колонки с гэпами для удобства просмотра. Последовательности доменов заданной группы специфичности mali расположены наверху, названия содержащих их белков окрашены в красный цвет. В другие цвета окрашены названия белков, содержащих домены разных групп специфичности (например, названия белков с доменами группы специфичности frur окрашены в светло-зелёный цвет, galrs - в салатовый цвет и т.д.). Колонки букв, окрашенные цветом, совпадающим с цветом названия белков указывают на позиции, консервативные в доменах соответствующей группы специфичности. Позиции, консервативные в доменах всех групп специфичности, окрашены в черный цвет. Для заданной мне группы MALI позиции характерные только для неё, покрашенные в светло-красный цвет. Для нахождения консервативных позиций была нажата кнопка "D", в результате чего буквы в позициях, соответсвующих консенсусу всего выравнивания были заменены на точки. Были выбраны позиции двух типов: консервативные внутри группы и не заменённые точками; консервативные внутри группы и заменённые точками, но не консервативные в других последовательностях.
- Эффекторный домен: effect.png. Выравнивание сохранено в файле effect.msf.
Консервативна для всех белков семейства только позиция: лейцин/изолейцин/валин/метионин 146. Для изучаемой группы специфичности были найдено 35 консервативных позиций:Позиции Аминокислотные остатки 65 S, T 69 E 74 L, M 76 F 87 G 169 P 176 A 181 T 209-210 SL 214 A 220 G 223 C 225 T, S 228 L, V, M 230 Y, F 232 G 235-236 PF 248 E 250 C 257 Q 261 A 280 V, I 297-298 YG, FG 305 R 310 G 322 Q 326 L 329 F 331 D 337 L 344 F, W 346 S, T
- ДНК-связывающий домен: dna_bind.png. Выравнивание сохранено в файле dna_bind.msf.
Позиции, консервативные для всего выравнивания: серин 19, лейцин/изолейцин/валин (метионин только в одной последовательносте) 22, валин/изолейцин 39, тирозин/фенилаланин 48. Для изучаемой группы специфичности были найдено две консервативных позиции (лизин 28 и глицин 29).
- Эффекторный домен: effect.png. Выравнивание сохранено в файле effect.msf.
- Создание лого-изображения полного выравнивания заданных доменов и выравнивания доменов заданной группы специфичности
ДНК-связывающий домен:
- Logo для полного выравнивания: logo_dna.png
- Logo для выравнивания группы mali: logo_mali_d.png
Эффекторный домен:
- Logo для полного выравнивания: logo_effect.png
- Logo для выравнивания группы mali: logo_mali_e.png
- Третий этап: поиск белка заданной группы специфичности в протеоме заданного организма
- pfw - для добавления весов в выравнивание группы специфичности
- pfmake - для составления профиля группы специфичности
- autoscale - для нормирования этого профиля
- pfsearch - для для поиска последовательностей в протеоме по профилю (со значением порога 16.0 для ДНК-связывающего домена и 27.0 для эффекторного домена)
- ClustalW2 - для выравнивания найденных последовательностей под выравнивание семейства
- ДНК-связывающий домен: bac_thu_dna.msf, bac_thu_dna.png
Были найдены 27 последовательсностей. Среди них многие последовательсности оказалисьидентичных с разными AC. Поэтому в выравнивание оставлена только 4 уникальных последовательности. Некоторые позиции в найденных последовательностях не совпадают с консервативными в группе. Кроме того среди специфических для данной группы позиций, в найденной последовательности есть только одна совпадающая позиция (Gly 26) показана красным.
Для проверки воспользуемся файлом 1BDH
Цепь A - белковая(голубая), цепь B - ДНК (расскраска по элементам), эффектор: гипоксантин (красным)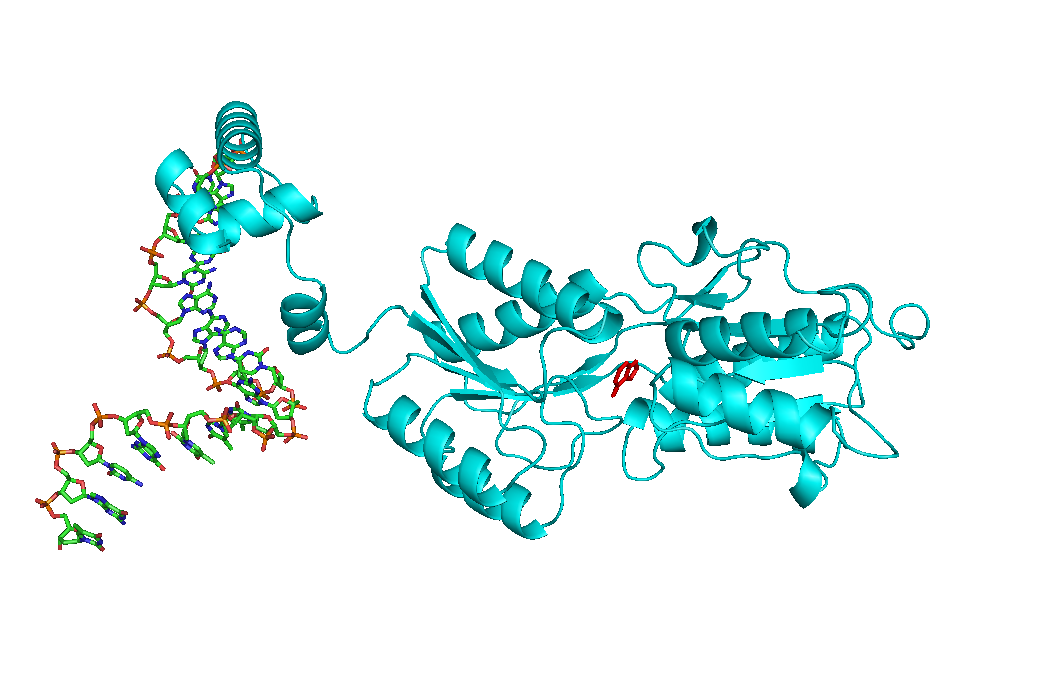
С помощью запроса в PyMOL: select contact, (c. a&!r. HOH&!r. HPA) w. 4.0 of (c. b)
Были выбраны атомы ДНК-связывающего домена, находящиеся на расстоянии не более 4 ангстрем от ДНК. Гидрофобные и гидрофильные взаимодействия не различались. Это позиции Val13-Thr17, Arg26-Thr32, Leu54-Ala55. Позиции 15, 30 и 31 не были найдены, но были добавлены, так как находятся между другими позициями, определяющими специфичность. Не все из этих позиций совпадают в найденной последовательности с группой специфичности, но совпадающие позиции не являются специфичными только для данной группы. - Эффекторсвязывающий домен: bac_thu_effect.msf, bac_thu_effect.png
Были найдены 23 последовательсностей. Среди них многие последовательсности оказалисьидентичных с разными AC. Поэтому в выравнивание оставлена только 3 уникальных последовательности. Консервативных в группе позиций, совпадающих в найденных последовательностях, меньше половины от всех консервативных. Количество совпадений (покрашены красным) с специфическими позициями невелико.
- Вывод
Был выдан белок-прототип MALI_ECOLI. Белок выделен из бактерии Escherichia coli K-12 Имя гена - malI, длина последовательности 342 аминокислотных остатков.
Доменная структура белка MALI_ECOLI (информация из БД Pfam):
Cхема из Pfam: |
||||
| Пояснения к схеме |
||||
| Pfam AC | Pfam ID | Положение в последовательности белка MALI_ECOLI | Клан | |
| PF00356 | LacI | 8-53 | HTH (CL0123) | |
| PF00532 | Peripla_BP_1 | 64-342 | Periplas_BP-like (CL0144) | |
В БД EcoCyc был произведен поиск информации о данном белке. Была найденна страница с описанием белка MALI_ECOLI.
Транскрипционный фактор malI (Мальтозный репрессор) отрицательно саморегулируется и контролирует транскрипцию оперона, который кодирует мальтоза-глюкоза PTS пермеазу (фермент, участвующие в транспорте метаболитов) и бифункциональный белок, который взаимодействует с активатором транскрипции MalT.
Термины GO, связанные с белком MALI_Ecoli:
| GO | Онтология GO (название словаря) | Термин |
| GO:0006355 | biological process | regulation of transcription, DNA-dependent |
| GO:0045449 | biological process | regulation of transcription |
| GO:0006350 | biological process | transcription |
| GO:0003677 | molecular function | DNA binding |
| GO:0003700 | molecular function | transcription factor activity |
| GO:0005622 | cellular component | intracellular |
| Где? (cellular component) |
Внутриклеточный |
|---|---|
| Зачем, для чего? (biological process) |
Транскрипция; регуляция ДНК-зависимой транскрипции |
| Молекулярный механизм? (molecular function) |
Связывание с ДНК; активность транскрипционного фактора |
| Специфичность? (molecular function) |
- |
Таким образом, в аннотации GO нет ни слова об эффекторе белка MALI_ECOLI - мальтозе.
Структурная формула мальтозы (БД KEGG, ID C00208):