|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
V Семестр
Проекты
Обратная Связь
|
SCOP и CATH
CATH, не выдал классификация доменов, данного мне белка 1KSK (написал, что домены находится на HOMCHECK_REVIEW flow stage).
Поэтому я взял два других белка: 1JHF и 4TMK.
1) Классификация доменов записей PDB 1JHF и 4TMK согласно SCOP
С помощью SCOP было найдено 2 домена в записи 1JHF:
Первый из них - N-концевой
домен репрессора LexA из E. coli (LexA repressor, N-terminal DNA-binding domain from Escherichia coli):
- Расположен в цепи А белка 1JHF, занимает в цепи позиции 2-72.
- Классификация по SCOP: класс - All alpha proteins; укладка - DNA/RNA-binding 3-helical bundle;
суперсемейство - "Winged helix" DNA-binding domain; семейство - LexA repressor, N-terminal DNA-binding domain.
- Суперсемейство ДНК-связывающий домен содержащий "крыло", небольшой бета-слой ("Winged helix" DNA-binding domain) содержит 84 семейства.
- Укладку ДНК/РНК-связывающий 3-спиральный "пучок" (DNA/RNA-binding 3-helical bundle) содержит 14 суперсемейств.
Второй из них -
С-концевой домен белка LexA из E. coli (LexA C-terminal domain from Escherichia coli), имеется две копии данного домена:
- Расположен в цепи A белка 1JHF, занимает в цепи позиции 73-198 (А также в цепи В, занимает всю цепь целиком).
- Классификация по SCOP: класс - All beta proteins; укладка - LexA/Signal peptidase;
суперсемейство - LexA/Signal peptidase ; семейство - LexA-related.
- Суперсемейство LexA/Сигнальных пептидаз (LexA/Signal peptidase) содержит 2 семейства.
- Укладку LexA/Сигнальных пептидаз (LexA/Signal peptidase) содержит 1 суперсемейство.
Возьмем для дальнейшего исследования запись 4TMK (белок KTHY из E. coli). Согласно SCOP белок состоит из 1 домена -
Тимидилат киназа из
E. coli (Thymidylate kinase from Escherichia coli):
- Расположен в цепи А белка 4TMK, занимает всю цепь целиком;
- Классификация по SCOP: класс - альфа/бета белки (Alpha and beta proteins (a/b)); укладка - нуклеозидтрифосфат-гидролаз, содержащих P-петлю
(P-loop containing nucleoside triphosphate hydrolases); суперсемейство - нуклеозидтрифосфат-гидролаз, содержащих P-петлю
(P-loop containing nucleoside triphosphate hydrolases); семейство - нуклеотид и нуклеозид киназ (Nucleotide and nucleoside kinases).
- Суперсемейство нуклеозидтрифосфат-гидролаз, содержащих P-петлю (P-loop containing nucleoside triphosphate hydrolases), содержит 24 семейства.
- Укладку нуклеозидтрифосфат-гидролаз, содержащих P-петлю, (P-loop containing nucleoside triphosphate hydrolases) содержит 1 суперсемейство.
2) Классификация доменов записей PDB 1JHF и 4TMK согласно CATH
С помощью CATH было найдено 3 домена в записи 1JHF,
домены 1jhfA01 и 1jhfB00 это фактически один и тот же домен, но CATH распознает их по разному, так как они расположены на разных цепях.
Поэтому рассмотрими один из них, а именно - 1jhfA01:
Первый из них - домен 1jhfA01:
- Расположен в цепи А белка 1JHF, занимает в цепи позиции 80-198.
- Классификация по CATH: 2.10.109.10.1.1.1.1.2
- Класс - Mainly Beta; архитектура - Ribbon; топология - Umud Fragment, subunit A; суперсемейство - Umud Fragment, subunit A.
- В данном суперсемействе содержится 4 семейства.
- Данную топологию имеют 1 суперсемейство.
Второй из них - домен 1jhfA02:
- Расположен в цепи А белка 1JHF, занимает в цепи позиции 2-70.
- Классификация по CATH: 1.10.10.10.37.1.1.1.1
- Класс - Mainly Alpha; архитектура - Orthogonal Bundle; топология - Arc Repressor Mutant, subunit A;
суперсемейство - "winged helix" repressor DNA binding domain.
- В данном суперсемействе содержится 174 семейства.
- Данную топологию имеют 37 суперсемейств.
Для записи 4TMK CATH определил 1 домена - 4tmkA00:
- Расположен в цепи А белка 4TMK, занимает в цепи позиции 2-211.
- Классификация по CATH: 3.40.50.300.39.1.1.1.1
- Класс - Alpha Beta; архитектура - 3-Layer(aba) Sandwich; топология - Rossmann fold;
суперсемейство - P-loop containing nucleotide triphosphate hydrolases.
- В данном суперсемействе содержится 208 семейства.
- Данную топологию имеют 120 суперсемейств.
3) Различия между CATH и SCOP в описании доменов записей 1JHF и 4TMK
Доменная организация белка записи 1JHF определена в CATH и SCOP одинаково.
Доменная организация белка записи 4TMK определена в CATH и SCOP одинаково.
N-концевой домен белка 1JHF определен в CATH и SCOP практически одинаково (за исключением того, что в SCOP его
укладка называется ДНК/РНК-связывающий 3-спиральный "пучок", а в CATH топология называется
"Мутантный Арк репрессор"; суперсемейства, следующие далее по классификации, для этого домена по CATH и SCOP совпадают).
Уровни С-концевого домен белка белка 1JHF по CATH и SCOP называются по-разному.
Укладка этого домена по SCOP - "LexA/Сигнальные пептидазы"; топология по CATH - "Umud фрагмент, субъединица A".
Суперсемейство по SCOP - "LexA/Сигнальные пептидазы"; суперсемейство по CATH - "Umud фрагмент, субъединица A".
Домен записи 4TMK описанный в CATH и SCOP практически одинаково (за исключением того, что в SCOP его
укладка называется "нуклеозидтрифосфат-гидролазы, содержащие P-петлю", а в CATH топология называется
"укладка Россманна"; суперсемейства, следующие далее по классификации, для этого домена по CATH и SCOP совпадают).
Некоторые домены имеют разную классификацию в CATH и SCOP.
Домен 1je8E00 из записи 1JE8, имеющий согласно CATH относится к тому же суперсемейству,
что и N-концевой домен белка 1JHF ("winged helix" repressor DNA binding domain),
но не входит в то же суперсемейство согласно SCOP (не "winged helix" repressor DNA binding domain, а
C-terminal effector domain of the bipartite response regulators).
Причинами таких различий в доменной классификации могут быть:
- Дата последнего релиза: у CATH не известна, но значительно позже - 7 июля 2009 года, в то время как SCOP последний раз обновлялся в июне 2009).
- Разное количество уровней: в SCOP их значительно меньше, чем в СATH.
4) Выравнивание доменов, имеющих одну укладку по SCOP (или топологию по CATH), но состоящих в разных суперсемействах
Построим выравнивание двух доменов с одной укладкой по SCOP, но из разных суперсемейств.
Для задания были выбраны цепь А записи 1jhf и цепь А
записи 1sfe.
Оба домена имеют укладку ДНК/РНК-связывающий 3-спиральный "пучок" (DNA/RNA-binding 3-helical bundle),
но первый из них принадлежит суперсемейству: "Winged helix" DNA-binding domain, а
второй - суперсемейству: Methylated DNA-protein cysteine methyltransferase, C-terminal domain .
Воспользуемся программой PDBeFOLD (SSM).
На выходе получаем файл с жестким выравниванием последовательностей в
формате fasta 1jhf_1sfe.fasta.
Подадим его на вход программе Geometrical core
для нахождения геометрического ядра с порогом 1.5Å.
На выходе получаем таблицу с остатками из структур, образующими геометрическое ядро:
| Pos. |
1JHF_A |
1SFE_A |
| 82 |
LEU4 |
GLY93 |
| 83 |
THR5 |
THR94 |
| 84 |
ALA6 |
ALA95 |
| 85 |
ARG7 |
PHE96 |
| 86 |
GLN8 |
GLN97 |
| 87 |
GLN9 |
GLN98 |
| 88 |
GLU10 |
GLN99 |
| 89 |
VAL11 |
VAL100 |
| 90 |
PHE12 |
TRP101 |
| 91 |
ASP13 |
GLN102 |
| 92 |
LEU14 |
ALA103 |
| 93 |
ILE15 |
LEU104 |
| 94 |
ARG16 |
ARG105 |
| 95 |
ASP17 |
THR106 |
| 106 |
ARG28 |
TYR115 |
| 107 |
ALA29 |
GLN116 |
| 108 |
GLU30 |
GLN117 |
| 109 |
ILE31 |
LEU118 |
| 111 |
GLN33 |
ASN120 |
| 112 |
ARG34 |
ALA121 |
| 115 |
PHE37 |
LYS124 |
Совместим в PyMOL командой pair-fit структуры по Cα-атомам, входящим в геометрическое ядро.
В результате получаем следующее изображение совмещенных структур:
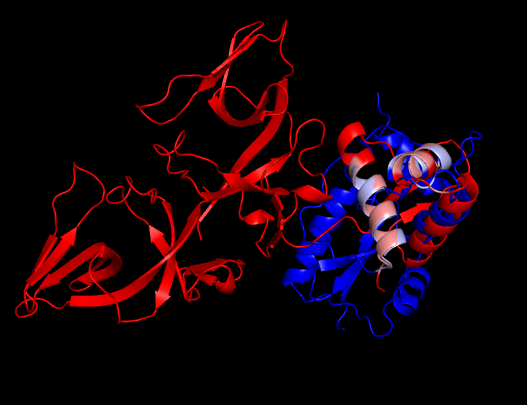
Красным на рисунке окрашена цепь А записи 1jhf, синим - цепь А записи 1sfe.
Светло-красным окрашены остатки цепи А записи 1jhf, входищие в состав геометрического ядра;
светло-синим - остатки цепи А записи А записи 1sfe, входищие в состав геометрического ядра.
RMSD совмещения по геометрическому ядру составляет 0.849 (21 to 21 atoms). Значение RMSD достаточно хорошее,
что говорит об успешном выравнивании и совмещении доменов.
Построим выравнивание двух доменов с одной топологией по CATH, но из разных суперсемейств.
Для задания были выбраны цепь А записи 4tmk и цепь А
записи 1c0p.
Оба домена имеют топологию "укладка Россманна" (Rossmann fold),
но первый из них принадлежит суперсемейству: P-loop containing nucleotide triphosphate hydrolases, а
второй - суперсемейству: NAD(P)-binding Rossmann-like Domain.
Воспользуемся программой PDBeFOLD (SSM).
На выходе получаем файл с жестким выравниванием последовательностей в
формате fasta 4tmk_1c0p.fasta.
Подадим его на вход программе Geometrical core для нахождения геометрического ядра с порогом 1.5Å.
На выходе получаем таблицу с остатками из структур, образующими геометрическое ядро:
| Pos. |
1C0P_A |
4TMK_A |
| 10 |
VAL1008 |
VAL7 |
| 11 |
VAL1009 |
ILE8 |
| 12 |
LEU1010 |
GLU9 |
| 23 |
ALA1020 |
ARG20 |
| 24 |
LEU1021 |
ASN21 |
| 25 |
ILE1022 |
VAL22 |
| 26 |
LEU1023 |
VAL23 |
| 101 |
HIS1031 |
GLY98 |
| 102 |
ILE1032 |
ASP99 |
| 243 |
ASP1173 |
ASP134 |
| 244 |
LEU1174 |
LEU135 |
| 245 |
VAL1175 |
THR136 |
| 246 |
VAL1176 |
LEU137 |
| 247 |
ASN1177 |
TYR138 |
| 440 |
GLN1340 |
PRO192 |
| 444 |
ALA1344 |
VAL196 |
| 450 |
GLN1350 |
THR202 |
| 451 |
LEU1351 |
THR203 |
| 452 |
VAL1352 |
VAL204 |
| 453 |
ASP1353 |
THR205 |
| 454 |
GLU1354 |
HIS206 |
| 455 |
ALA1355 |
TRP207 |
| 456 |
PHE1356 |
VAL208 |
| 457 |
GLN1357 |
LYS209 |
| 458 |
ARG1358 |
GLU210 |
Совместим в PyMOL командой pair-fit структуры по Cα-атомам, входящим в геометрическое ядро.
В результате получаем следующее изображение совмещенных структур:
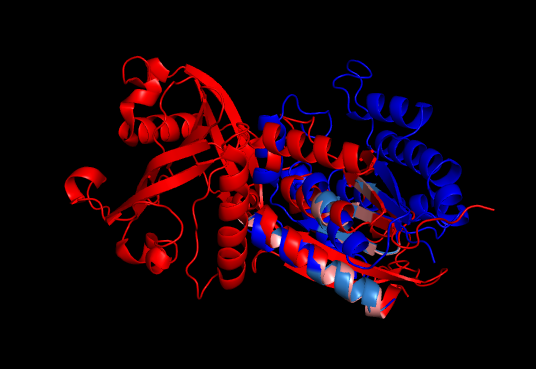
Красным на рисунке окрашена цепь А записи 4tmk, синим - цепь А записи 1c0p.
Светло-красным окрашены остатки цепи А записи 4tmk, входищие в состав геометрического ядра;
светло-синим - остатки цепи А записи А записи 1c0p, входищие в состав геометрического ядра.
RMSD совмещения по геометрическому ядру составляет 0.902 (25 to 25 atoms). Значение RMSD достаточно хорошее,
что говорит об успешном выравнивании и совмещении доменов. Но все же в данном случае домены выравнялись хуже,
чем в предыдущем случае.
Таким образом, можно сделать вывод, что домены, имеющие одинаковую укладку (топологию),
но входящие в разные суперсемейства, все же имеют достаточно сходную пространственную структуру.
|

