|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
V Семестр
Проекты
Обратная Связь
|
Поверхность, гидрофобное взаимодействие, домены
Поверхность
Для выполнения заданий была взята сборка биологической единицы 1QP0. Цепи A и M соответствуют цепям белка и ДНК из одной асимметрической
единицы, C и B соответствуют цепям белка и ДНК из другой асимметрической единицы.
Для каждого пункта были написаны скрипты для PyMOL, а затем создавалось изображение:
- Поверхность контакта мономера белка с симметричным мономером на фоне остовной
(ribbon) модели мономера (script_monom.pml).

- Поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка,
вовлечённой в контакт (script_dimer.pml).
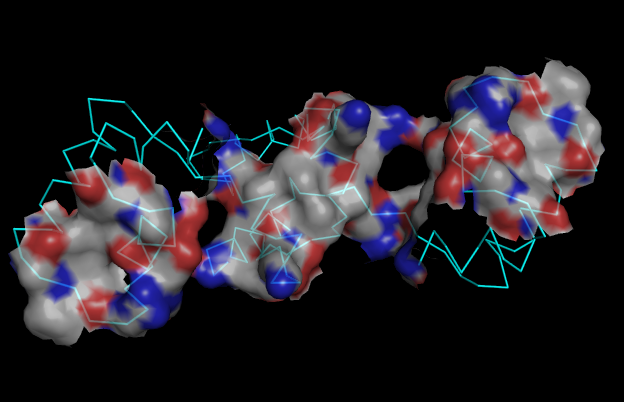
- Поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели
двойной спирали (script_dna.pml).

Площадь контактов
Найдем площадь контатов момомеров белка из сборки биологической единицы 1QP0.
Для этого воспользуемся сервисом
PROTORP.
Загрузим собственный файл, для чего воспользуется Option 3. Получили следующие
результаты:
площадь контакта белковых мономеров составляет 2570.26 Å2;
43.41% этой площади приходится на гидрофобное взаимодействие.
Гидрофобные кластеры мономеров белка записи 1QP0
Воспользуемся сервисом
CluD
и определим гидрофобные кластеры на интерфейсе мономеров белка записи 1QP0 объемом не менее 10 атомов.
Выходной файл сервиса - перечень атомов,
относящимися к гидрофобным кластерам.
Создано то же изображение, что в упр. 1A, на котором поверхность, относящаяся к атомам,
входящим в найденные гидрофобные кластеры, выделена различными цветами для каждого кластера.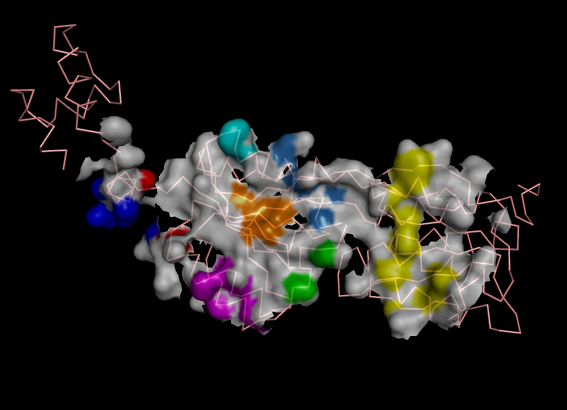
Доменная структура цепей белков записей 1KSK и 1QP0 согласно данным сервиса pDomains
Определим на сервисе pDomains
доменную структуру белков:
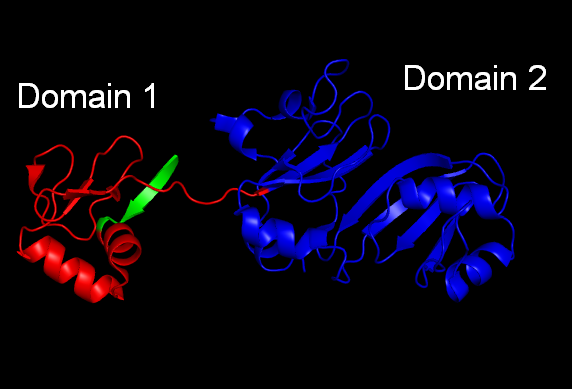
- а) Для цепи А белка записи 1KSK большинство методов выделения доменов определили 2 домена. Наиболее адекватными мне показались результаты метода SCOP.
Он выделил следующие домены:
- красным: 4-62
- синим: 63-234
Судя по изображению, это выглядит вполне достоверно, так как, действительно, отчетливо выделяются 2 гидрофобных ядра в структуре.
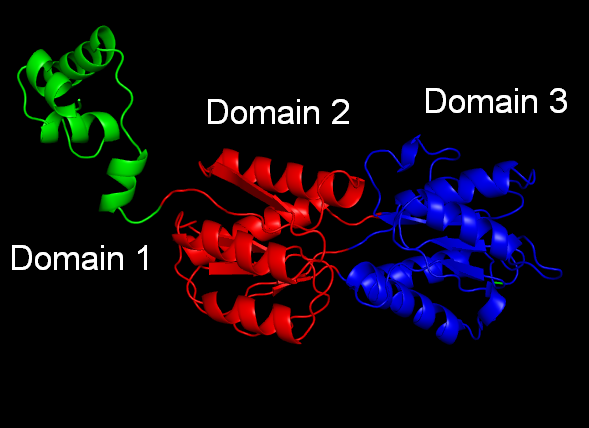
- б) Для цепи A белка записи 1QP0 различные методы определили доменную структуру по-разному.
Наиболее адекватными мне показались результаты метода CATH. Он выделил следующие домены:
- зеленым: 2-58
- красным: 59-160, 291-322
- синим: 161-290, 323-339
Судя по изображению, это выглядит вполне достоверно, так как, действительно, отчетливо выделяются 3 гидрофобных ядра в структуре.
|





