|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
V Семестр
VI Семестр
Проекты
Обратная Связь
|
Докинг низкомолекулярных лигандов в структуру белка
Цель работы - ознакомиться с возможностями докинга низкомолекулярного лиганда в структуру белка и пакета Autodock Vina и Autodock tools.
Программе Autodock Vina для докинга необходимы специально форматированные файлы pdb c зарядами и указанием торсионных углов. Для начала попробуем провести докинг одного из мономеров сахара (NAG) из прошлого занятия.
Работать будем с белком лизоцимом LYSC_LEPWE, структура которогобыла построена на основе гомологичного моделирования на прошлом занятии.
-
Найдем в банке PDB (www.pdb.org) SMILES аннотацию для NAG и сохраним ее в файле nag.smi.
-
Затем с помощью obgen построим 3D-структуру этого сахара в pdb-формате:
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb
В результате был получен файл nag.pdb.
-
С помощью cкрипта prepare_ligand4.py из пакета Autodock tools создадим pdbqt файл нашего лиганда:
prepare_ligand4.py -l nag.pdb
Полученный pdbqt-файл лиганда NAG: nag.pdbqt
-
Аналогично скриптом prepare_receptor4.py из пакета Autodock tools создадим pdbqt файл нашего белка LYSC_LEPWE.
prepare_receptor4.py -r lepwe_5.pdb
Полученный файл: lepwe_5.pdbqt.
-
Итак, мы получили необходимые входные файлы. Теперь создадим файл с параметрами докинга vina.cfg. Для докинга необходимо указать область структуры белка, в которой будет происходить поиск места для связывания. Удобно его задать как куб с неким центром. Координаты центра определим из модели комплекса, построенной на прошлом занятии. Выберем атом сахара, находящийся в центре сайта связывания (атом N2B), и извлечем из pdb-файла его координаты (40.781 42.027 26.420).
Создадим по этим данным файл vina.cfg.
-
Теперь можно провести первый докинг:
vina --config vina.cfg --receptor lepwe_5.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
В результате докинга были получены файлы nag_prot.pdbqt и nag_prot.log.
-
Просмотрим файл nag_prot.log. Энергии трех лучших расположений и геометрическая разница между ними представлена в таблице:
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 1 |
-5.9 |
0.000 |
0.000 |
| 2 |
-5.8 |
2.768 |
4.581 |
| 3 |
-5.6 |
2.029 |
3.194 |
Файлы nag_prot.pdbqt и lepwe_5.pdbqt были загружены в PyMOL. Все состояния на одной картинке изображены ниже:

Такая картинка, содержащяя все состояния лиганда, показывает, что молекула лиганда свободно перемещается внутри центра связывания белка (возможно, это связано с тем, что это лишь один из трех сахарных остатков реального лиганда).
Также видно, что в 4 случаях лиганд выходит из центра связывания. При том, что 2 из этих состояний локализованы в одном месте, а другие 2 состояния - в другом.
-
Теперь проведем докинг, рассматривая подвижность некоторых боковых радикалов белка.
Сначала разобьем белок на две части: подвижную и неподвижную. Для подвижной части выберем 3 аминокислоты, которые мы использовали в прошлом задании для позиционирования лиганда (Glu53, Trp82 и Asp120).
Для создания pdbqt-файла воспользуемся скриптом prepare_flexreceptor4.py:
prepare_flexreceptor4.py -r lepwe_5.pdbqt -s GLU53_TRP82_ASP120
В результате были получены файл lepwe_5_flex.pdbqt и lepwe_5_rigid.pdbqt.
Теперь проведем докинг:
vina --config vina.cfg --receptor model5_rigid.pdbqt --flex model5_flex.pdbqt
--ligand nag.pdbqt --out vina_prot_flex.pdbqt --log vina_prot_flex.log
В результате докинга были получены файлы vina_prot_flex.pdbqt и vina_prot_flex.log.
-
Просмотрим файл vina_prot_flex.log. Энергии трех лучших расположений и геометрическая разница между ними представлена в таблице:
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 1 |
-5.0 |
0.000 |
0.000 |
| 2 |
-4.9 |
1.808 |
3.207 |
| 3 |
-4.7 |
1.484 |
2.373 |
Файлы vina_prot_flex.pdbqt и lepwe_5_rigid.pdbqt были загружены в PyMOL. Все состояния на одной картинке изображены ниже:
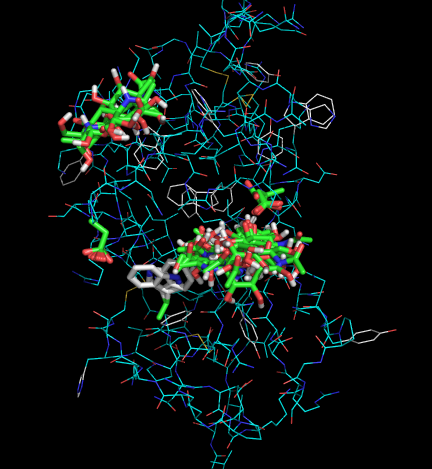
Как видно, теперь свое положение меняет не только лиганд, но и три аминокислоты белка (правда, из них сильно изменяет положение, пожалуй, только Trp-82; это связано с тем, что другие два остатка взаимодействовали в модели с другими мономерами лиганда).
Перемещение лиганда в подвижном докинге куда больше, чем в обычном, что вполне естесственно объясняется подвижностью аминокислотных остатков. В некоторых состояниях очень хорошо видно, как Trp82 сильно меняет конформацию, контактируя с лигандом.
Видно, что в случае подвижного докинга лиганд выходит из центра связывания в 5 случаях. Но во всех этих случаях лиганд, локализован примерно в одной области, в отличие от жесткого докинга.
-
Стоит отметить, что лучше с задачей справился обычный докинг, а не подвижный.
Обычный докинг, в одном случае, смог примерно расположить лиганд так, как он располагался в полученной модели.
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 20 |
-4.0 |
3.263 |
4.633 |
На картинке приведено расположение лиганда наиболее близкое к тому, что было получено в моделировании:
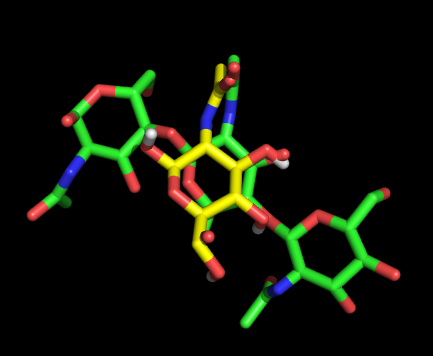
-
Создадим три лиганда, где метильный радикал СH3C(=O)NH группы NAG заменен на OH, NH2 и H.
Для этого были получены файлы nag_oh.smi, nag_nh.smi и nag_h.smi соответственно со SMILES измененных лигандов.
Затем был написан bash-скрипт: make.bash
- Таблица трех лучших расположений для первого лиганда:
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 1 |
-5.8 |
0.000 |
0.000 |
| 2 |
-5.7 |
2.221 |
4.472 |
| 3 |
-5.7 |
1.647 |
2.154 |
Изображение результата докинга для первого лиганда представлено ниже:

Как видно, из картинки, что лиганд не выходит из центра связывания, возможно это связано с возникновением дополнительных водородных связей, которые стабилизируют лиганд в центре связывания.
- Таблица трех лучших расположений для второго лиганда:
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 1 |
-6.0 |
0.000 |
0.000 |
| 2 |
-5.8 |
2.733 |
4.545 |
| 3 |
-5.8 |
1.600 |
2.165 |
Изображение результата докинга для второго лиганда представлено ниже:

В этом случае лиганд выходит из центра связывания, лишь в одном случае. Это связано с возникновением дополнительных водородных связей, которые стабилизируют лиганд в центре связывания.
- Таблица трех лучших расположений для третьего лиганда:
| Расположение |
Энергия (ккал/моль) |
rmsd l.b. |
rmsd u.b. |
| 1 |
-5.3 |
0.000 |
0.000 |
| 2 |
-5.1 |
1.592 |
2.085 |
| 3 |
-5.1 |
1.978 |
4.740 |
Изображение результата докинга для третьего лиганда представлено ниже:
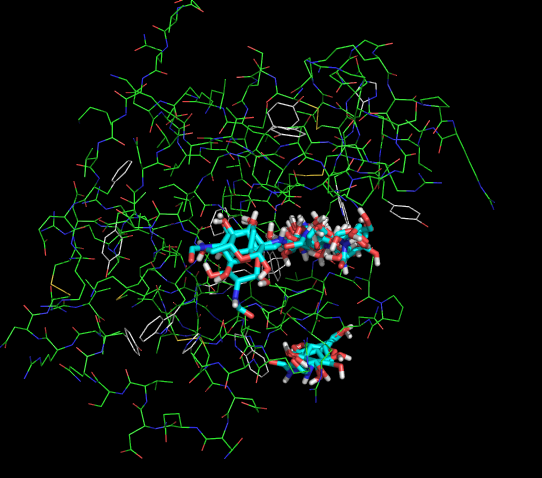
В этом случае лиганд выходит из центра связывания, так как не появилось дополнительных связей (по сравнению с лигандом, содержащим метильный радикал), которые могли бы стабилизировать лиганд в центре связывания.
|





