| % Identities | % Positives | Gaps | Length | Score | Score (bits) |
| 29 | 48 | 59(8%) |
P06210: 615 P03306: 644 |
636 | 249 |
| 36 | 49 | 43(13%) |
P06210: 306 P03306: 275 |
397 | 157 |

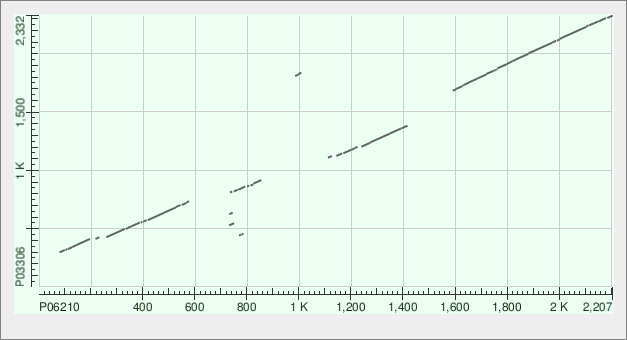
Рисунок 1.Карта локального сходства белков P06210 и P03306
| Координаты участка белка | Названия зрелых белков |
| (I)P06210: 1592-2206 | Protease 3C; RNA-directed RNA polymerase |
| (I)P03306: 1684-2327 | Picornain 3C; RNA-directed RNA polymerase 3D-POL |
| (II)P06210: 1112-1417 | Protease 2A; Protein 2B; Protein 2C |
| (II)P03306: 1109-1383 | Protein 2C |
| Характеристика | Uniprot ID | Score (оптимального локального выравнивания) | Score (bits) | Median | Верхний квартиль | P-значение |
| Пара гомологичных белков | TSAD_ECOLI и TSAD_BACSU | 711.5 | 7.125 | 49.5 | 55.625 | 7.2×10-3 |
| Пара негомологичных белков | FRMA_ECOLI и MURB_BACSU | 54.0 | 1.9 | 48.5 | 54.625 | 2.7×10-1 |
| Uniprot ID | Uniprot AC | Organism | % Identities | % Positives | Length | Gaps | Score | Score (bits) | Expect | % Coverage |
| CATJ_PSEKB | Q8VPF2.1 | Pseudomonas knackmussii B13 | 31 | 47 | 260 | 25 | 228 | 92.4 | 3e-21 | 89 |
| GCTB_ACIFV | Q59112.3 | Acidaminococcus fermentans DSM 20731 | 27 | 44 | 266 | 29 | 211 | 85.9 | 7e-19 | 91 |
© Наумова Юлия, 2018