Из списка функций белков был выбран фактор элонгации трансляции Ts (EFTS).
С помощью Swiss-Prot были получены последовательности белков с данной функцией
из отобранных ранее бактерий (Таблица 1.).
| Идентификатор | Мнемоника | Название белка |
| Q6G5C8 | EFTS_BARHE | Elongation factor Ts |
| Q1BHI1 | EFTS_BURCA | Elongation factor Ts |
| P43894 | EFTS_HAEIN | Elongation factor Ts |
| A1B8E8 | EFTS_PARDP | Elongation factor Ts |
| B4F2D2 | EFTS_PROMH | Elongation factor Ts |
| A4SYV0 | EFTS_POLAQ | Elongation factor Ts |
| P57983 | EFTS_PASMU | Elongation factor Ts |
Все последовательности были собраны в один файл (pr2t4in.fasta) и выровнены
с помощью программы: muscle -in pr2t4in.fasta -out pr2t4out.fasta.
Также для удобства в файле были отредактированы названия последовательностей (до мнемоник).
Итоговый файл с выравниваниями: pr2t4new.fasta
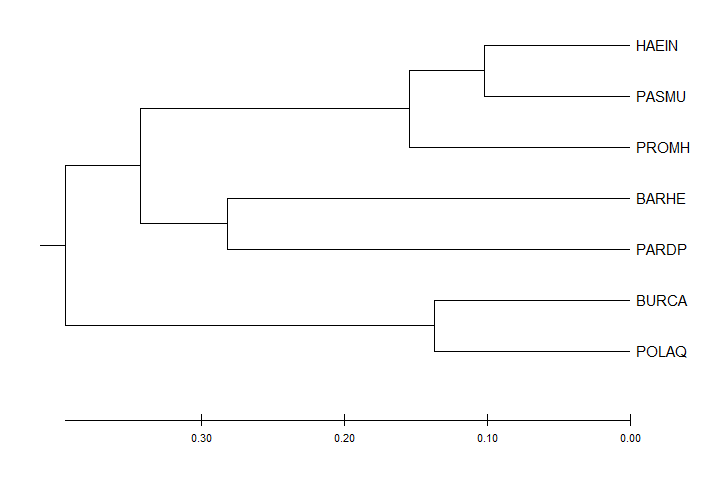
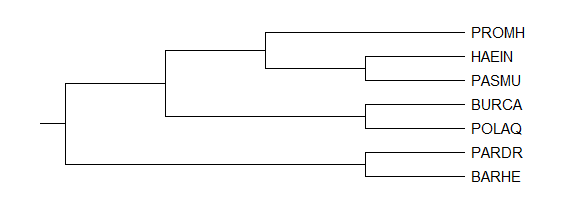
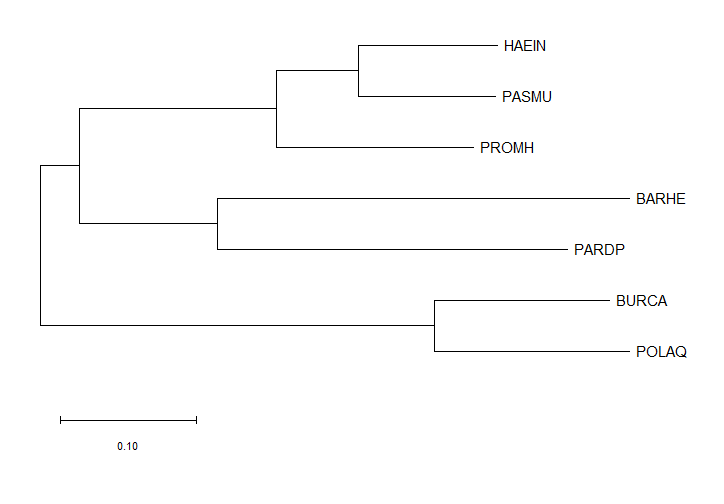
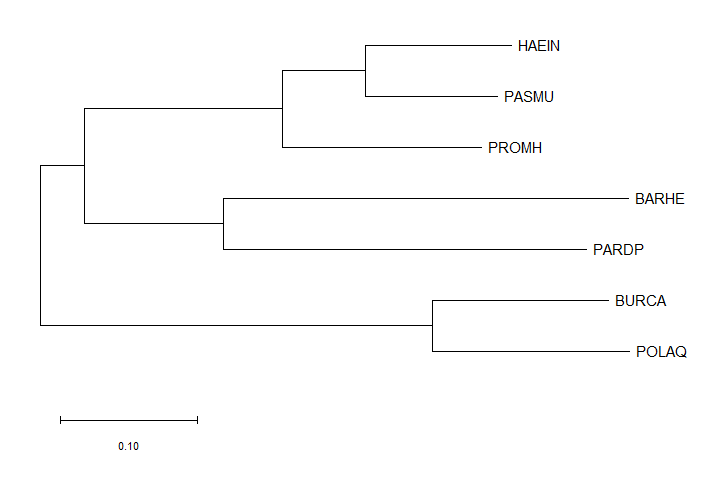
Далее была произведена реконструкция филогении тремя методами: UPGMA, Minimum-Evolution, Neighbor-Joining.
UPGMA
* использует молекулярные часы (строит укорененное ультраметрическое дерево)
* дистанционный
* реконструирует длины ветвей
 |  |
Minimum-Evolution
* переборный
* может использовать молекулярные часы
* дистанционный
* реконструирует длины ветвей
 |  |
Neighbor-Joining
* прямой
* не использует молекулярные часы (строит неукорененное дерево)
* дистанционный
* реконструирует длины ветвей
 |  |
© Наумова Юлия, 2019