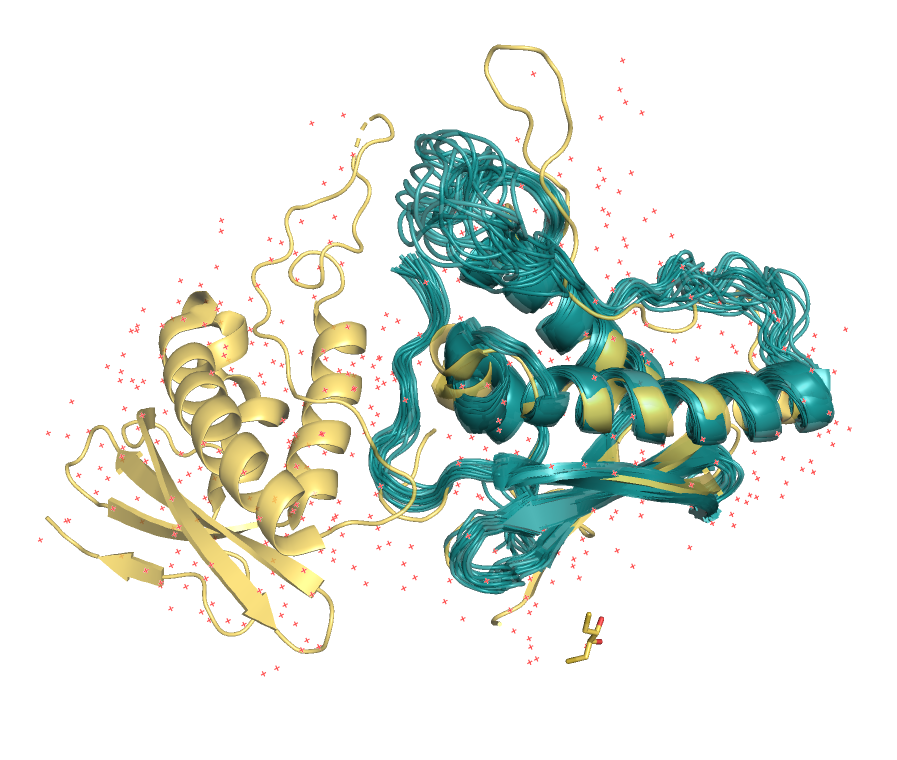
В данном задании нам были даны два PDB ID структуры PX-домена Bem1p, полученные методами ЯМР
(2CZO)
и РСА (2V6V).
Для 2CZO представлено 20 моделей, а для 2V6V разрешение составляет 1.50 ангстрем.
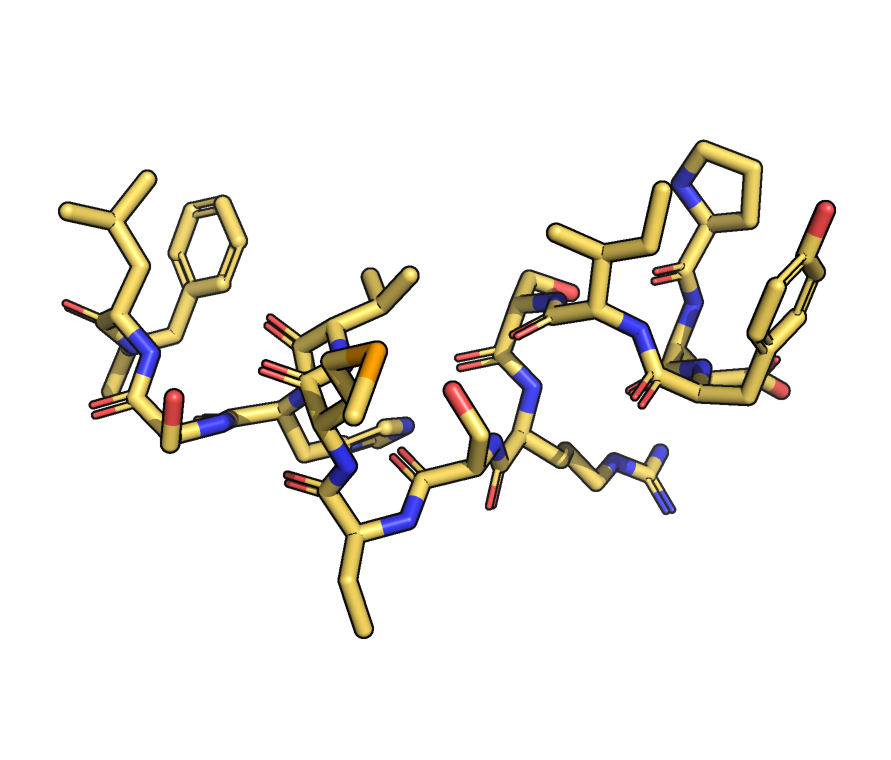
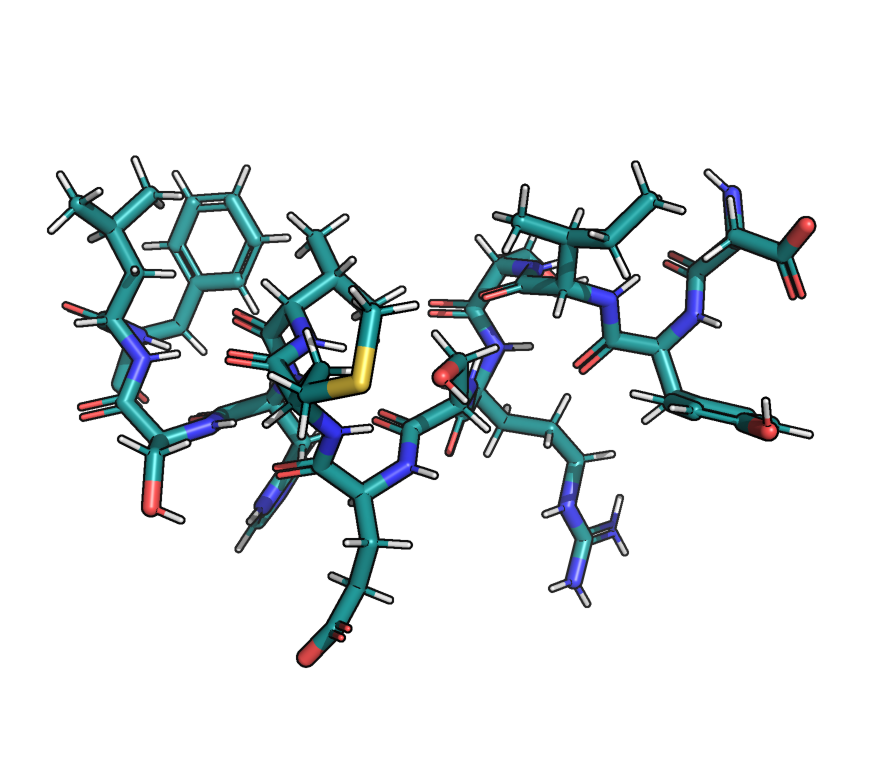
На рисунке 1 показаны данные структуры в "совмещенном" состоянии.

Различия моделей:
- Молекулы воды, а также отдельный лиганд DTT (2,3-дигидрокси-1,4-дитиобутан) принадлежат
структуре РСА (так как в ЯМР такие молекулы не детектируются). Рисунки 1 и 2.
- Структура, полученная методом РСА представляет собой 2 молекулы белка
в кристаллографической ячейке, в то время как структура ЯМР состоит из 20 моделей.
- В целом структуры отлично совпадают в альфа-спиралях и бета-листах.
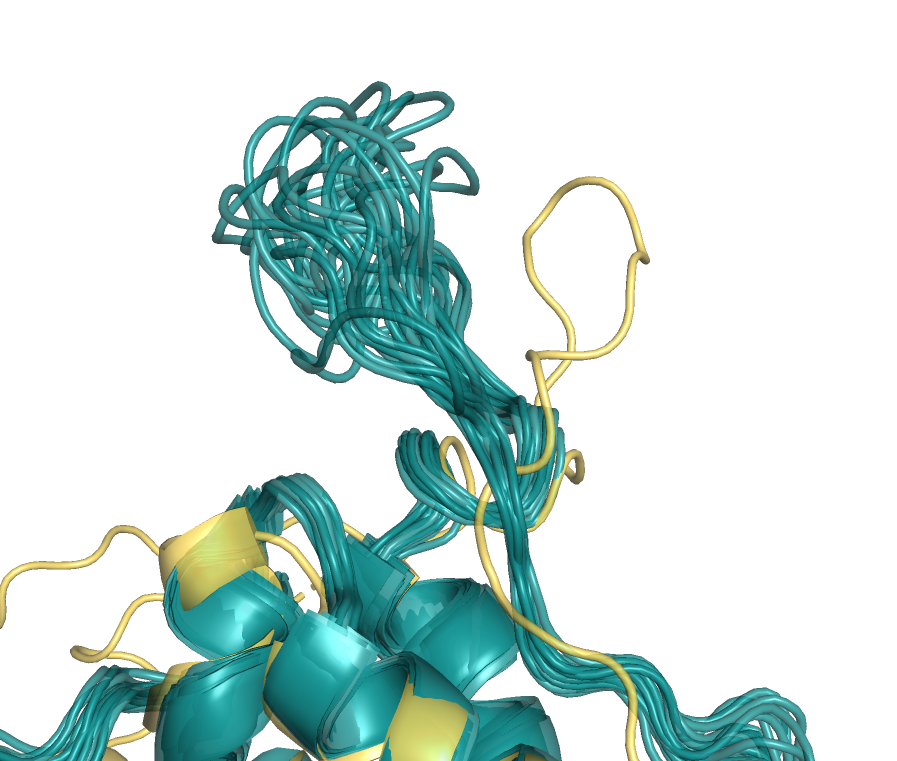
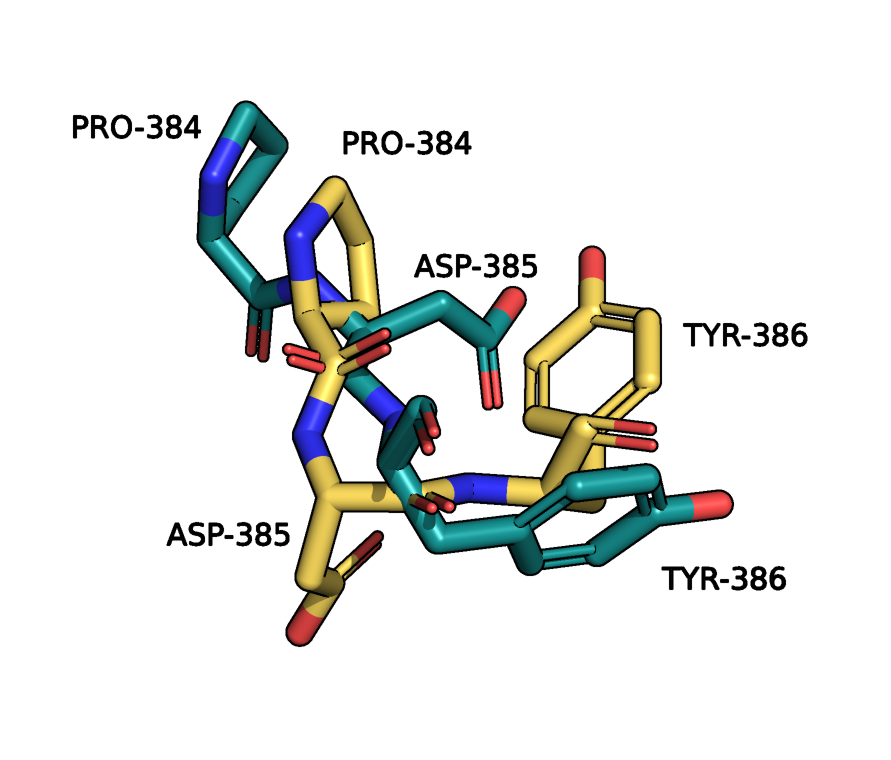
- В начале и конце молекулы структуры не совпадают (с одного конца
длиннее ЯМР модель, с другого длиннее РСА). Рисунок 3.
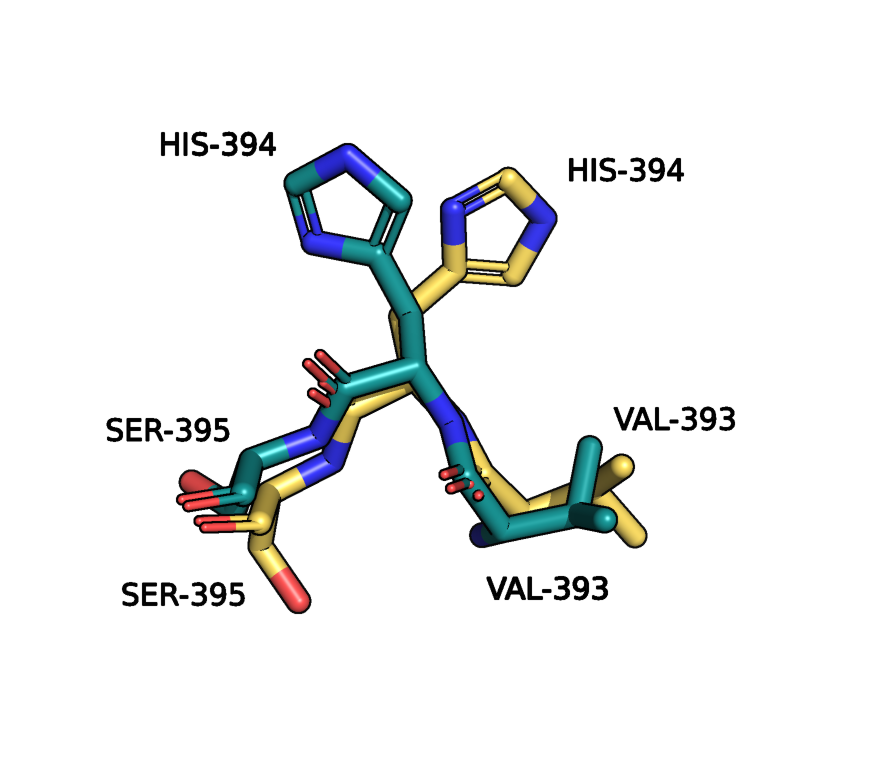
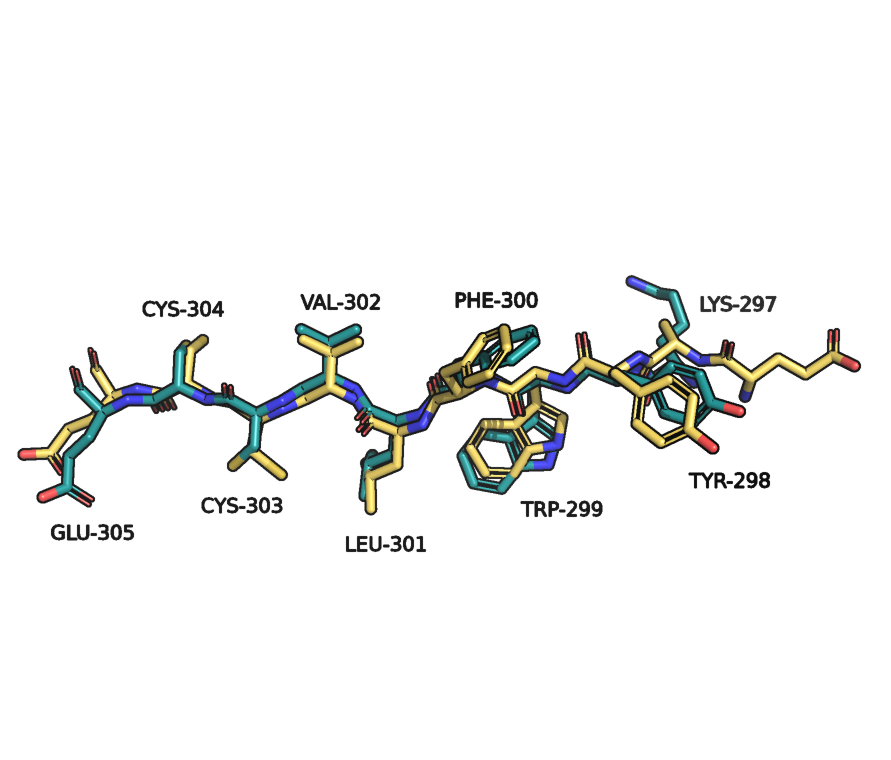
- Также есть сильно различающиеся участки (боковая цепь смотрит в разные стороны). Рисунок 4.
- Одна из альфа-спиралей выглядит довольно странно в обоих структурах, и "съезжает" при их сравнении. Рисунок 5.
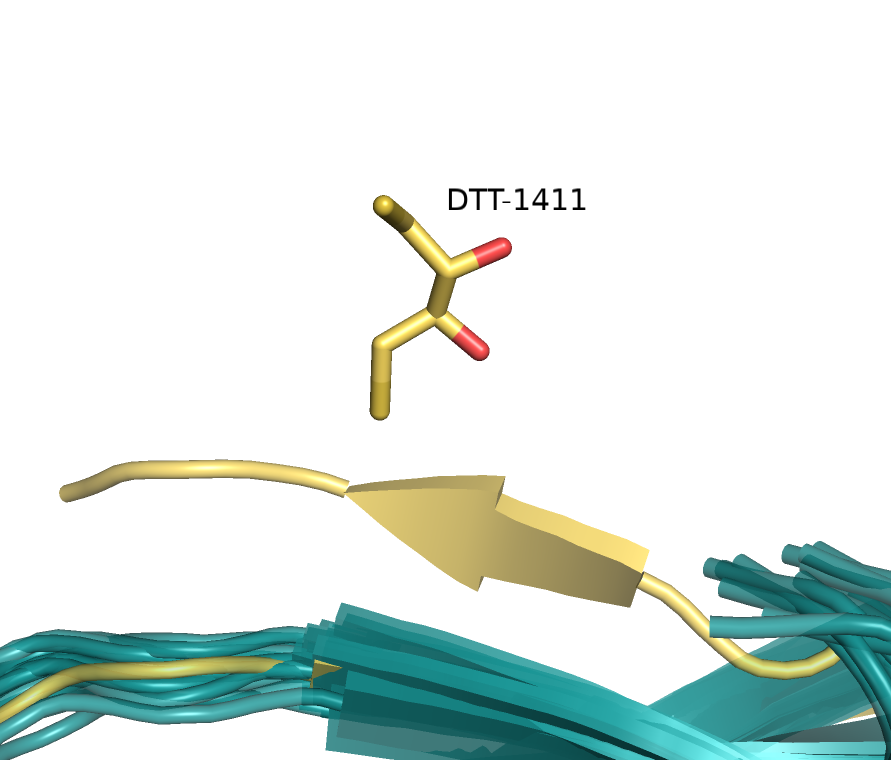
2V6V, полученной методом РСА. |

РСА (желтая окраска) и ЯМР (синяя окраска). |
в структурах РСА (желтая окраска) и ЯМР (синяя окраска). |
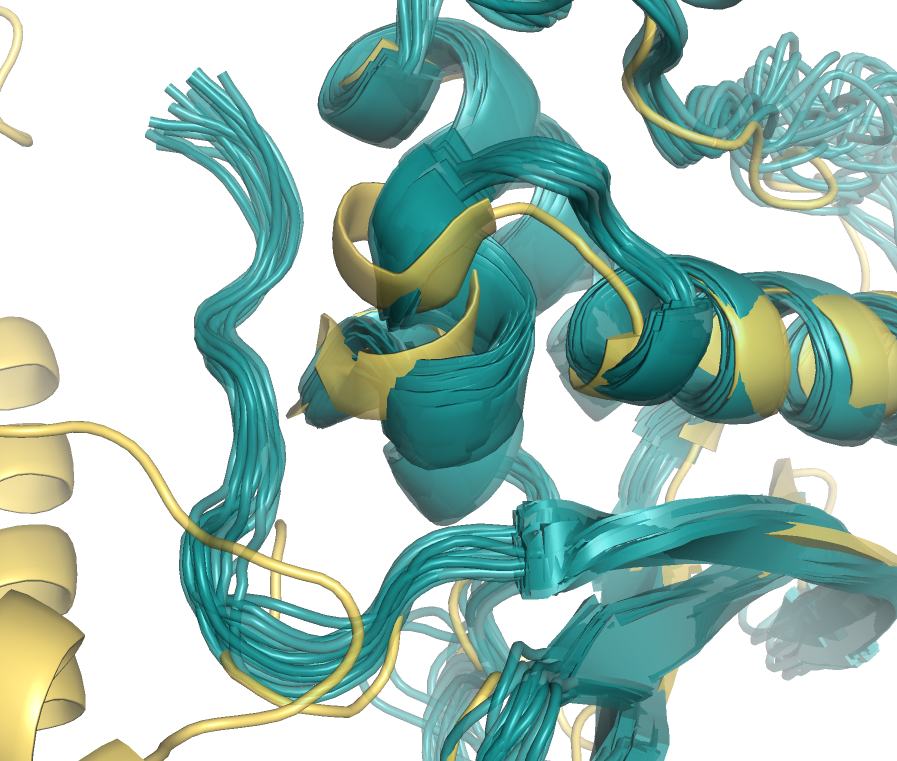
РСА (желтая окраска) и ЯМР (синяя окраска). |
Далее модели были рассмотрены более детально:
- При просмотре участка структуры ЯМР мы видим водороды, тогда как в структуре
РСА их нет (но можно вычислить с помощью PyMOL). Рисунки 6 и 7.
- При сравнении структур альфа-спирали и бета-листы в целом не сильно различаются
(хотя в ЯМР модели расположение АК как будто более хаотичное). Рисунки 6 и 7.
- В альфа-спирали я выделила несколько АК, которые сильно отличаются при сравнении моделей. Рисунки 8 и 9.
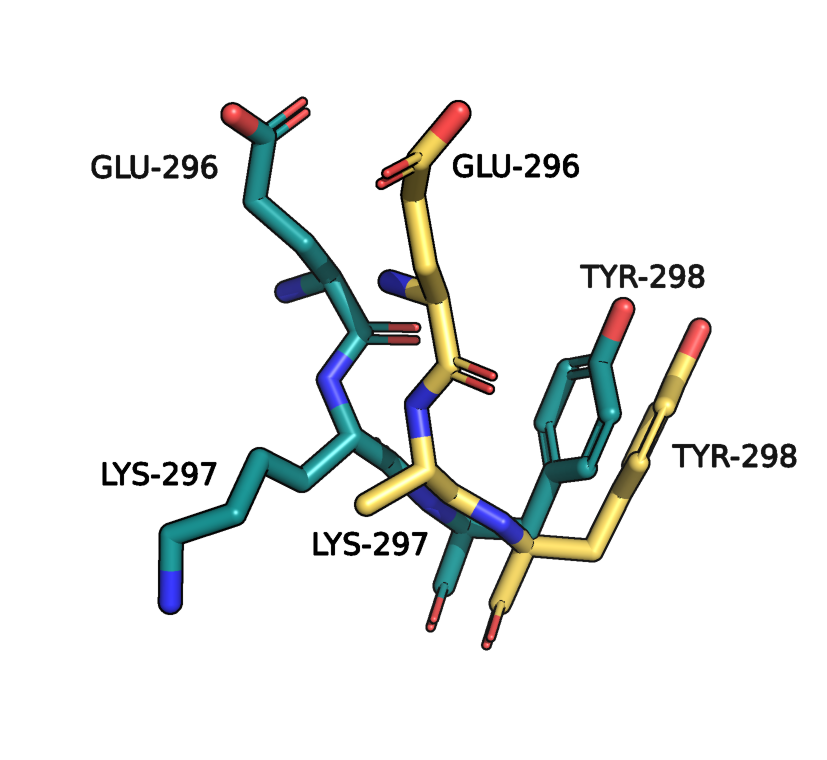
- Для бета-тяжа (рисунок 10) также был выбран плохо совпадающий участок.
Здесь в одной из структур произошла ошибка. В модели ЯМР LYS-297 вполне нормальный,
тогда как в модели РСА он состоит из 5 атомов (по сути, просто "обрублен").
Скорее всего, это произошло из-за того, что не удалось определить ЭП. Рисунок 11.
|
|
(желтая окраска) и ЯМР (синяя окраска). |
структурах РСА (желтая окраска) и ЯМР (синяя окраска). |
(желтая окраска) и ЯМР (синяя окраска). |
(желтая окраска) и ЯМР (синяя окраска). |
В данном задании мы оценивали В-фактор остатков в зависимости от их подвижности (RMSF).
Была построена зависимость рассчитанных В-факторов модели РСА от RMSF.
Для этого "обрезали" не совпадающие остатки на концах молекул и выровняли модели.
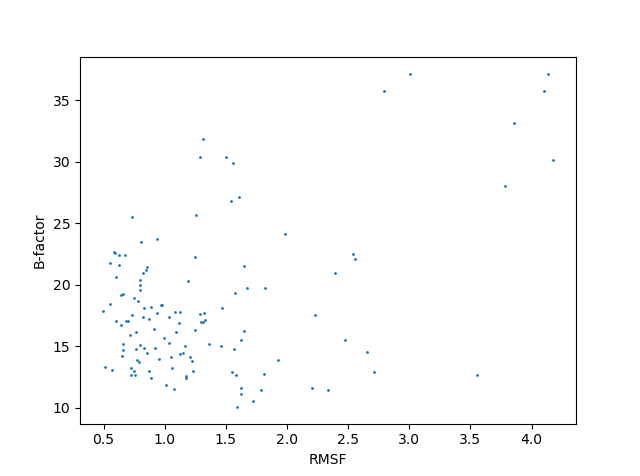
Можно заметить, что большое значение RMSF соответствует большому значению В-фактора.
Однако, высокий В-фактор есть и у остатков с меньшим значением RMSF.
Таким образом, делаем вывод о том, что здесь В-фактор означает не подвижность остатка, а качество ЭП.
Здесь мы сравнивали водородные связи моделей РСА и ЯМР в различных местах белка.
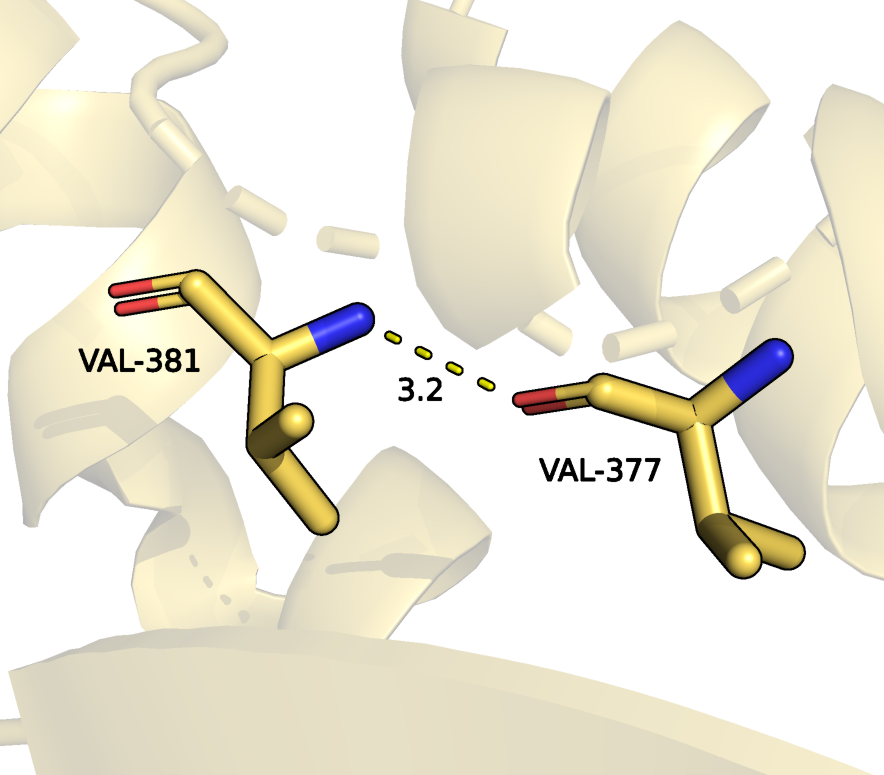
1. Связь между атомами остова в альфа-спирали.
Для сравнения была выбрана водородная связь в структуре РСА между VAL-377 и VAL-381
(длина 3.2 ангстрема), которые находятся в альфа-спирали внутри глобулы.
В модели ЯМР данная связь также присутствует, однако ее длина составляет 3.6 ангстрем.
Но я считаю это допустимым в данном случае.
Также эта связь присутствует во всех 20 моделях ЯМР.
альфа-спирали. Длина 3.2 ангстрем. Структура РСА. |
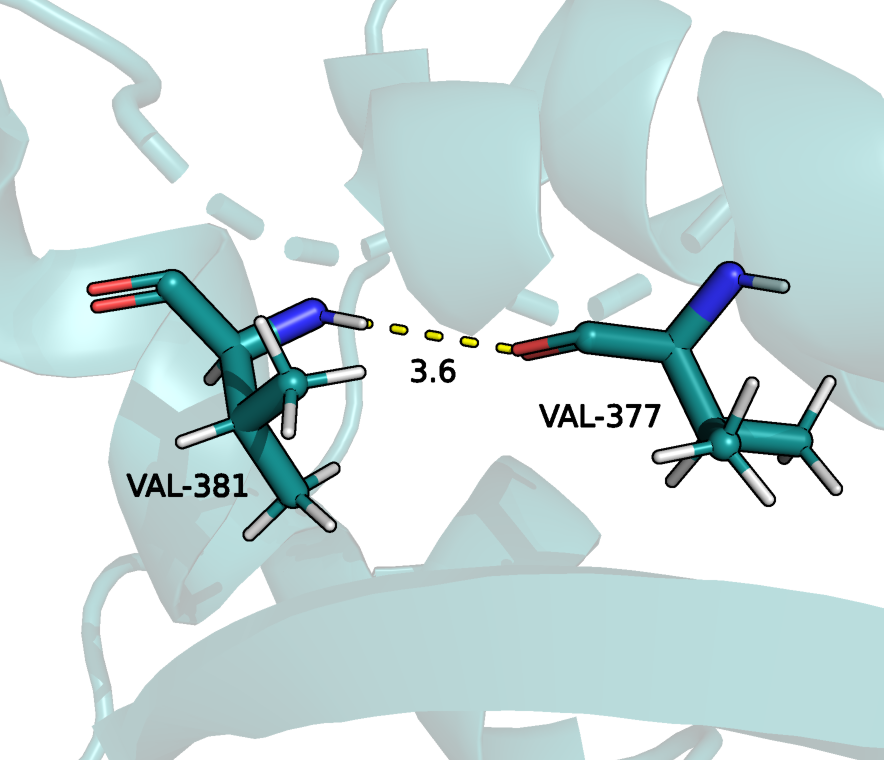
альфа-спирали. Длина 3.6 ангстрем. Структура ЯМР. |
2. Водородная связь боковых цепей в ядре белка.
Здесь рассматривалась связь между ASN-374 из альфа-спирали и VAL-288 из бета-тяжа
(внутри глобулы белка). В структуре РСА данная связь возможна и ее длина составляет 2.9 ангстрем.
Однако во всех 20 ЯМР моделях такое взаимодействие невозможно (большие расстояния и плохие углы).
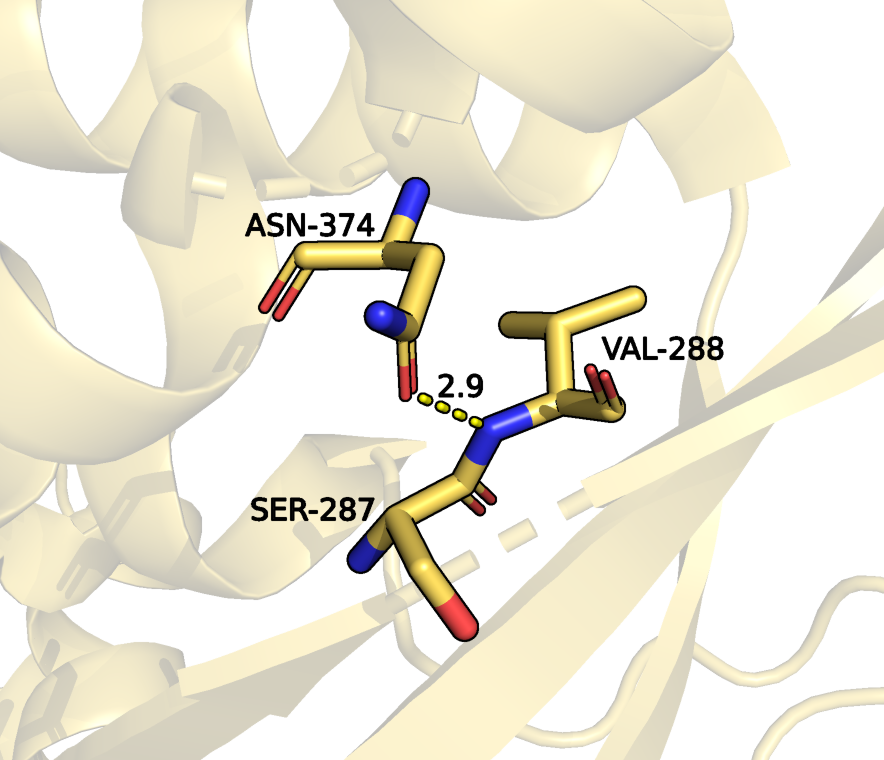
и VAL-288 бета-тяжа. Длина 2.9 ангстрем. Структура РСА. |
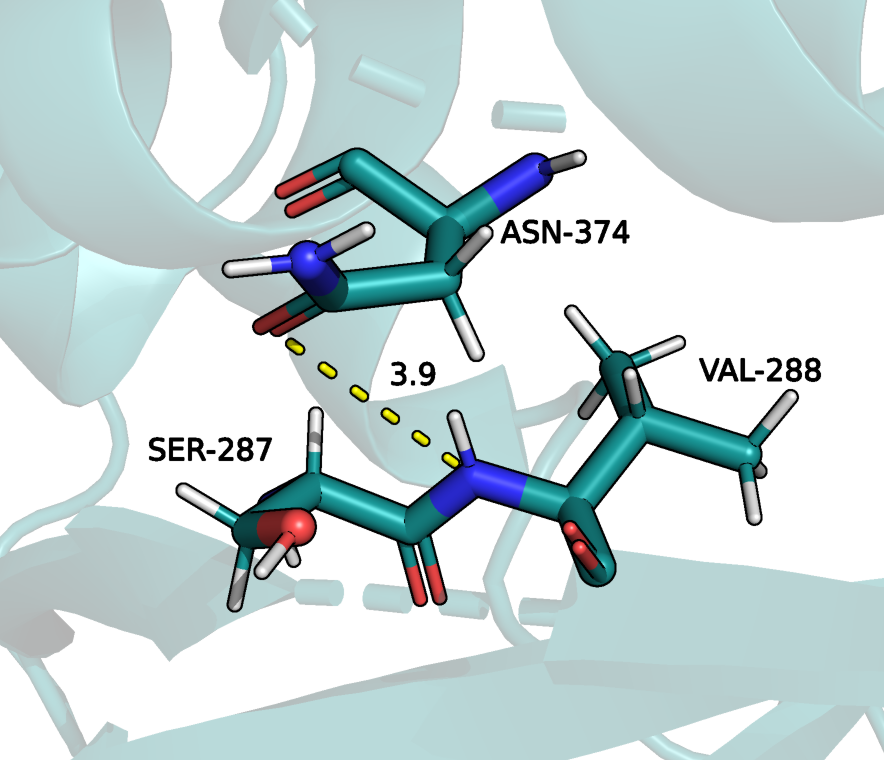
альфа-спирали и VAL-288 бета-тяжа. Структура ЯМР. |
3. Водородная связь в петле, выходящей на поверхность глобулы.
Тут для сравнения были выбраны ARG-340 и GLY-344 из петли, выходящей на
поверхность глобулы белка. В структуре РСА связь составляет 2.7 ангстрем.
А во всех 20-ти моделях ЯМР такая связь невозможна, так как АК находятся на
большом расстоянии (от 5.4 до 15 ангстрем).
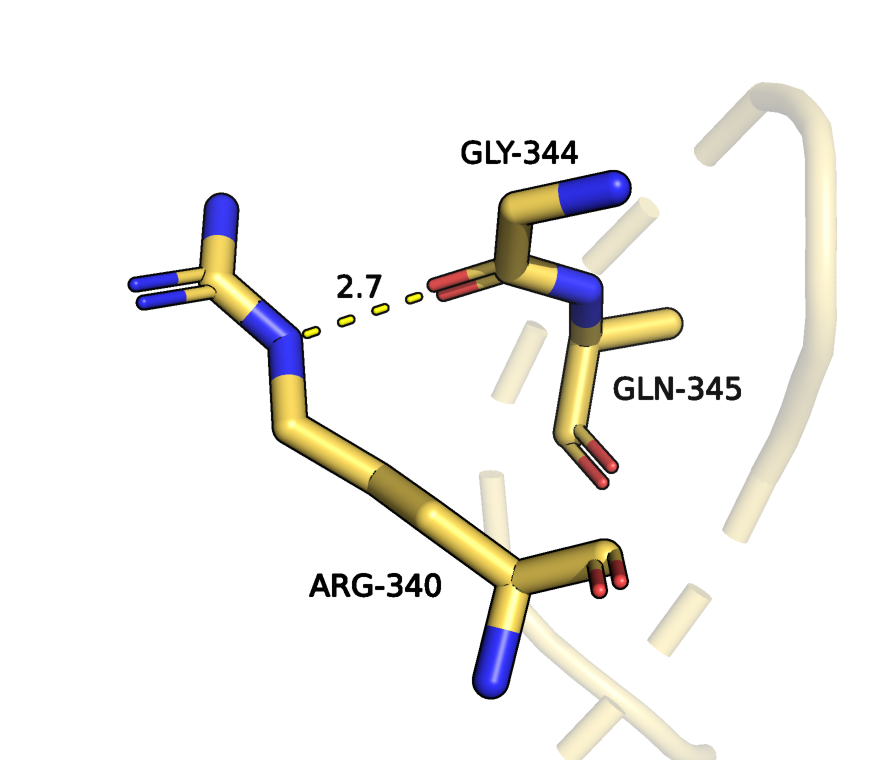
выходящей на поверхность глобулы. Длина 2.7 ангстрем. Структура РСА. |
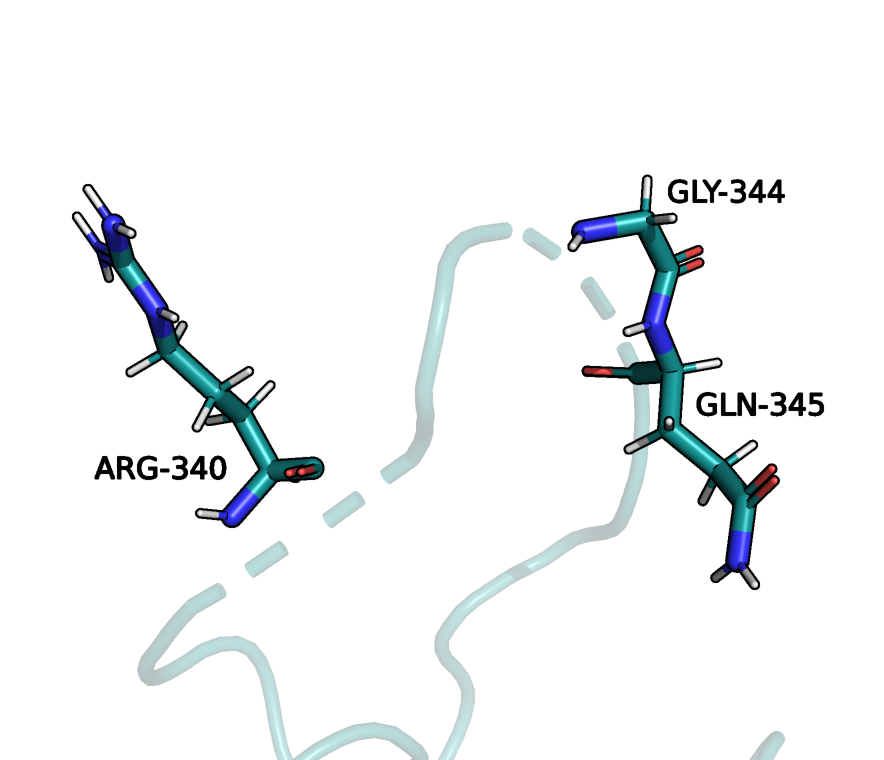
GLY-344 из петли, выходящей на поверхность глобулы. Структура ЯМР. |
| Номер связи | Расстояние в РСА | Процент присутствия в ЯМР | Минимальное расстояние в ЯМР, А | Максимальное расстояние в ЯМР, А | Медианное расстояние в ЯМР, А |
| 1 | 3.2 | 100% | 3.4 | 3.7 | 3.6 |
| 2 | 2.9 | 0 | 2.5 | 6.0 | 4.0 |
| 3 | 2.7 | 0 | 5.4 | 15.0 | 10.2 |
1. В первом случае все логично. Альфа-спираль в глобуле является довольно устойчивой структурой.
Также можно упомянуть, что альфа-спирали в структурах ЯМР и РСА хорошо совпадали.
Следовательно, во всех структурах мы увидели связь.
2. Во втором случае можно предположить ошибку в модели РСА, так как ни в одной структуре ЯМР
не было нужной для образования связи конформации (большое расстояние и неправильные углы).
3. В третьем случае я не могу дать однозначного ответа, почему связь в РСА существует, а в ЯМР - нет.
Возможно это связано с кристаллизацией, подвижностью петель или, как во 2 пункте, с ошибкой в модели.
© Наумова Юлия, 2020