Была получена консенсусная последовательность из выравнивания прямой и обратной последовательностей (программы consambig)
С помощью BLASTN было установлено к какому гену принадлежит эта последовательность, а так же определена таксономия организма.
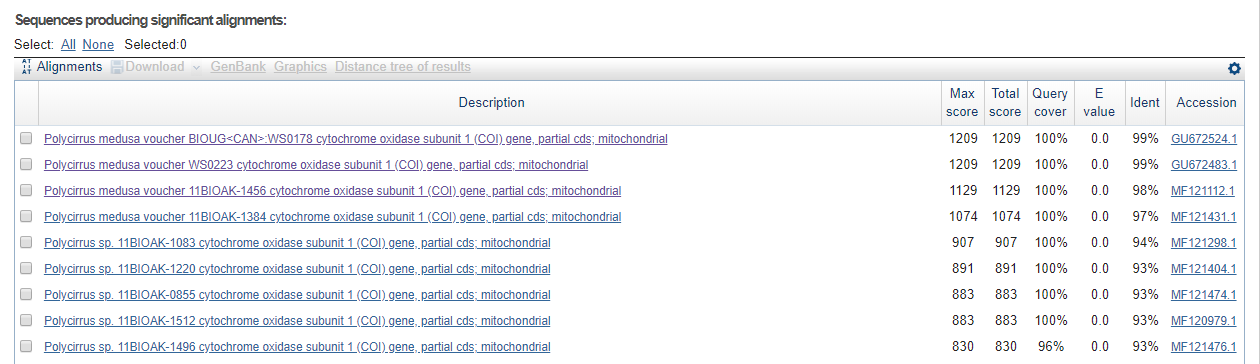
Blast нашёл для данной последовательности несколько достоверных находок, из которых были выбраны 3 лучшие:
Query cover 100%, E-value равно 0 и процент идентичных позиций превышает 90% (Рис 3).
Заданные параметры и выдача:

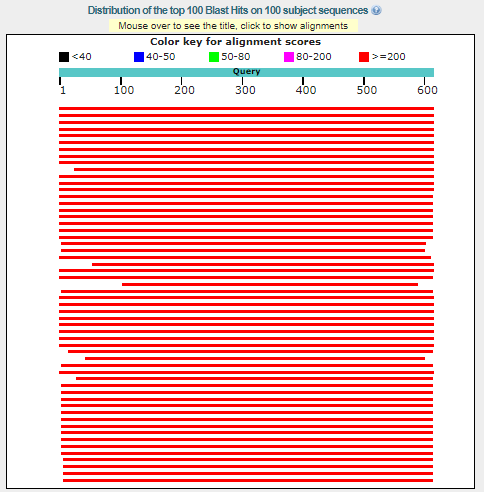
Рис 2.
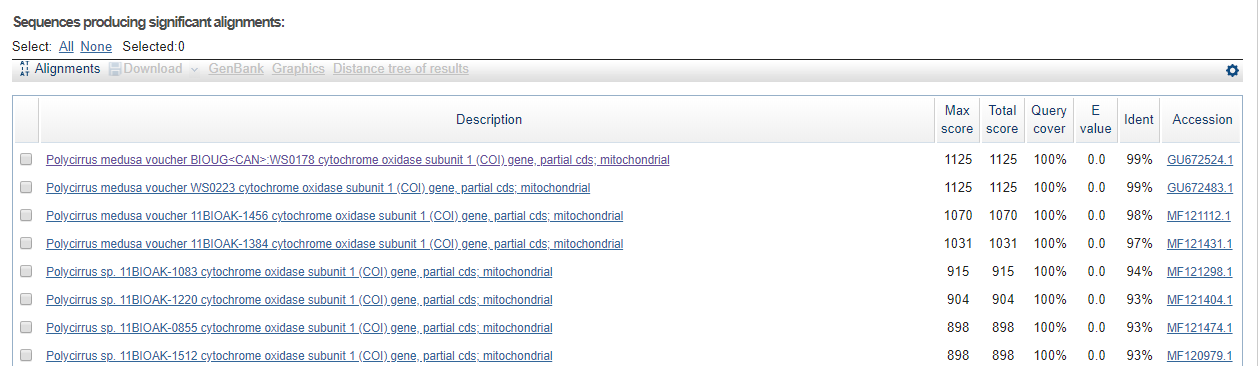
Рис 3.
Последовательности во всех трёх находках представляют собой один и тот же ген,
кодирующий субъединицу 1 митохондриального фермента цитохромоксидазу (COI).
Все находки из организма Polycirrus medusa.
Оба организма принадлежат к одному роду многощетинковых кольчатых червей - Polycirrus.
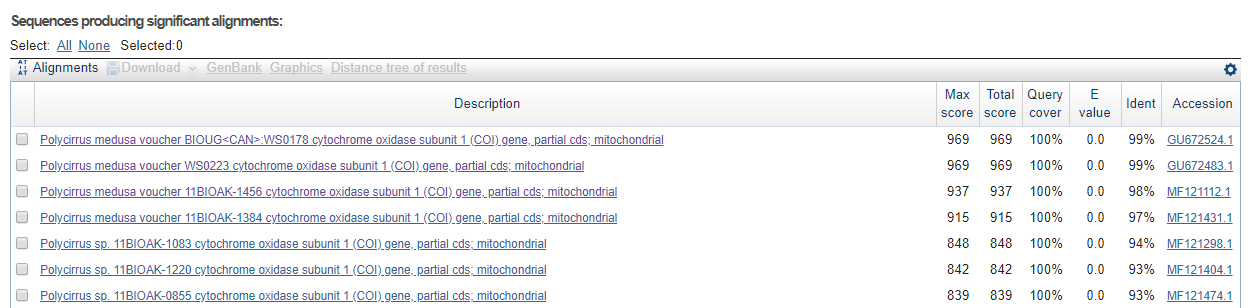
Таксономия (взята в прямоугольник) представлена на Рис 4.

Рис 4.
| Алгоритм | Word size | Max score | Gap Costs |
| megablast | 28 | 1, -2 | Linear |
| blastn | 11 | 1, -3 | 5, 2 |
| blastn | 7 | 1, -1 | 5, 2 |
Просто по данной последовательности blastn находил более 1000 находок, поэтому область поиска была ограничена семейством,
к которому принадлала лучшая находка - Terebellidae (taxid:32261).
Выдача:
Megablast предназначен для очень похожих последовательностей.
Полученные результаты демонстрируют это: процент сходства варьирует от 79% до 99%.
Всего находок получено 32.
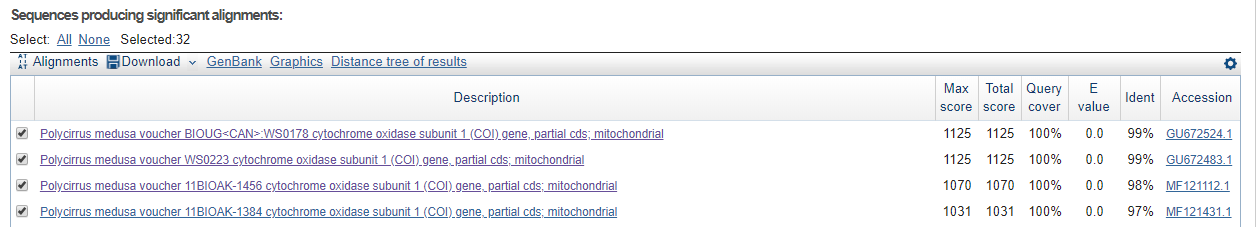
Blastn предназначен для поиска похожих последовательностей (найдены участки с высоким процентом идентичности, но с E-value больше 0).
Всего находок получено 118.
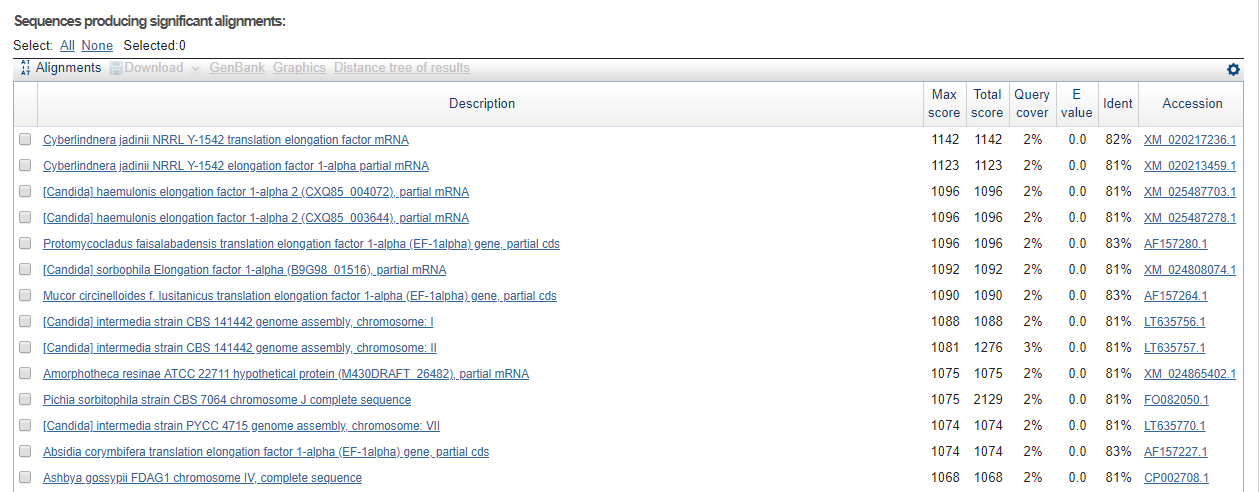
При более чувствительных параметрах получилось меньше находок (112 находок).
Таким образом, сравнение разных алгоритмов демонстрирует их функциональное различие. Количество находок Blastn больше, но и их качество хуже.
Для выполнения задания была взята сборка генома Amoeboaphelidium protococcarum
С помощью tblastn был произведён поиск против локально созданной из сборки базы данных.
Для выполнения задачи из банка данных были скачаны:
HSP71_YEAST - шаперон HSP71, белок теплового шока;
TBB_NEUCR - тубулин, белок, участвующий в образовании микротрубочек;
PRPC_EMENI - митохондриальная цитратсинтаза
Пример команды запуска: "tblastn -query hsp71.fasta -db X5.fasta -outfmt 7 > hsp71.out"
Выдача программы для каждого белка:
Из сборки генома Amoeboaphelidium protococcarum был извлечён скэффолд scaffold-6 с длиной 53904. Для поиска был использован megablast.
Результаты выдачи приведены ниже:
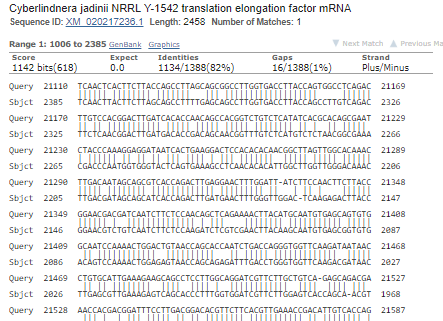
Лучшая находка - Cyberlindnera jadinii NRRL Y-1542 translation elongation factor mRNA
Ее длина - 2458, выровнялось с 1006 по 2385 аминокислоты. Примерные границы гена в скаффолде: 21110-22489.
Для выравнивания были выбраны геномы Staphylococcus lugdunensis HKU09-01 и Staphylococcus warneri SG1 (CP001837.1 и CP003668.1 соответсвенно),
Использовался megablast.
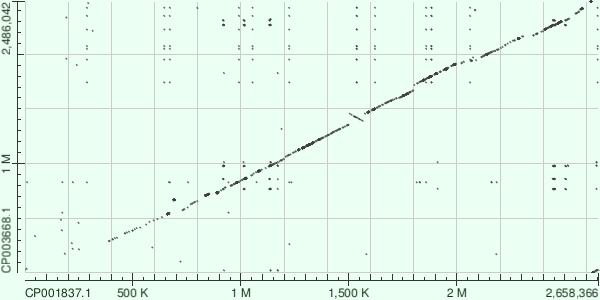
По изображению можно предположить, что в районе 1500 Kb произошла небольшая инверсия.
© Наумова Юлия, 2018